부처연구성과
암 관련 유전자 검색 엔진 개발
- 등록일2014-05-23
- 조회수5084
-
성과명
암 관련 유전자 검색 엔진 개발
-
연구자명
박종철
-
연구기관
카이스트 전산학과
-
사업명
중견연구자지원사업
-
지원기관
미래창조과학부
-
보도자료발간일
2014-05-23
- 원문링크
-
키워드
#암 유전자
- 첨부파일
핵심내용
관련 연구문헌의 쉽고 빠른 검색으로 효율적인 암 연구 도와
□ 국내 연구진이 암 관련 유전자를 보다 빠르고 정확하게 찾을 수 있는 특화된 검색엔진을 개발했다. 메드라인*에 탑재된 300만 건에 육박하는 암 관련 유전자에 대한 연구문헌에서 암-유전자 관련성에 대한 정보를 빨리 검색할 수 있어 암 연구를 위한 새로운 도구가 될 것으로 기대된다.
* 메드라인(Medline) : 생물학 및 의학 연구문헌을 저장하는 데이터베이스
o 카이스트(KAIST) 전산학과 박종철 교수 연구팀이 지스트(GIST) 이현주 교수와 함께 수행한 이번 연구는 언어학, 컴퓨터공학, 생물학 및 의학을 포괄적으로 연계하는 융합연구로 미래창조과학부가 추진하는 중견연구자지원사업의 지원으로 수행되었고 연구결과는 생물학 분야 학술지 핵산연구(Nucleic Acids Research) 온라인판 5월 9일자에 게재됐다. (논문명: OncoSearch: Cancer Gene Search Engine with Literature Evidence)
□ 암은 수천 개 이상 유전자의 비정상적 변화와 그에 따른 신호전달 체계 교란이 주요 원인으로 암의 원인을 이해하고 치료하기 위해서는 이들 유전자의 변화와 암과의 관련성을 이해하는 것이 중요하다.
o 이에 수많은 유전자 변화의 암 관련성에 관한 연구결과들을 바로 파악하고 수집하여 종합적으로 분석, 판단하는 것이 요구된다.
□ 연구팀은 의학 및 생물학 연구문헌에서 유전자의 발현량 변화*와 유전자 변화에 따른 암 상태 변화를 기술하는 문장을 찾아내는 검색엔진 온코서치(OncoSearch, http://oncosearch.biopathway.org)를 개발했다.
* 유전자 발현량 변화 : 유전자의 발현이란 DNA가 RNA 및 단백질로 전사 및 번역되는 것으로 유전형이 표현형으로 반영되는 가장 기초적인 단계이다. 따라서 유전자 발현량의 변화는 유전자가 표현형을 조절하는 가장 기본적 도구가 된다.
o 개발된 온코서치는 첨단 텍스트마이닝 기술인 사건 정보 추출 시스템과 최대 엔트로피* 분류기를 사용해 문장의 구조를 심도 있게 분석, 유전자 발현량의 증감 및 암의 진행상태 파악을 용이하도록 했다.
* 최대 엔트로피(maximum entropy) : 알려진 사전 정보를 기반으로 엔트로피가 최대가 되는 확률분포를 최적으로 선정하는 원리로 최대 엔트로피 분류기란 이러한 원리를 기반으로 확률모델을 만들어 이종데이터를 분류하는 분류기이다. 연구에 사용된 최대 엔트로피 분류기는 연구팀이 자체 연구, 개발한 말뭉치인 코맥씨(CoMAGC)를 기반으로 개발됐다.
o 특히 각 유전자가 암 진행에 기여하는 정도를 문장 구조 분석 및 추론을 통해 파악하기 때문에, 기존의 암 관련 유전자 정보 수집 기법들과는 달리 암 관련 유전자 역할에 대한 명시적인 표현(oncogene, tumor suppressor 등)이 없어도 관련된 정보를 파악할 수 있다는 점에서 수월성을 보인다.
□ 또한 메드라인에 등재된 모든 논문에서 1,700종 이상의 악성종양과 7,500개 이상의 유전자에 관한 문헌정보를 빠르고 정확하게 검색, 대량의 정보를 쉽게 수집할 수 있게 됨에 따라 암 연구의 질적향상에 기여할 것으로 기대된다.
□ 박 교수는 “온코서치가 첨단 텍스트마이닝 기술을 사용해 연구문헌에서 자동으로 수집한 암 관련 유전자에 대한 대량의 정보는 향후 자동 추론기술 등을 활용해 암 연구를 위한 새로운 도구로 활용될 수 있다”고 의의를 밝혔다.
상세내용
연 구 결 과 개 요
1. 연구배경
암은 유전자의 이상 변화가 주요한 발생원인 중 하나로 알려져 있다. 따라서 암 연구에서는 암 발생에 관련된 유전자들을 파악하고 이들 유전자들이 암 발생 기작에 어떻게 기여하는 지를 잘 이해하는 것이 매우 중요하다. 이에 따라 각종 암 관련 유전자에 관하여 국제적으로 수많은 연구가 진행되었으며, 그 결과가 연구 문헌 데이터베이스로 축적되어 왔다. 암 연구 진행을 위해서는 이러한 기존 연구 결과들을 수집 및 분석하는 것이 요구되지만, 관련 연구 문헌의 양이 너무 방대하기 때문에 텍스트마이닝 기술을 기반으로 하는 검색 엔진의 활용이 필수적이다.
그러나 기존의 암 관련 문헌 검색 엔진들은 유전자가 암에 어떻게 영향을 미치는지를 이해하는데 필수적인 정보인 유전자 발현량 변화 및 이에 따른 암의 상태 변화에 대한 검색 기능을 제공하지 못하고 있다. 이에 보다 효율적인 암 연구를 위해서는 이러한 구체적 정보에 특화된 검색 엔진이 필요하다.
2. 연구내용
본 연구에서는 의료, 생물학 연구 문헌에서 유전자 발현량 및 이에 따른 암의 상태 변화를 기술하는 문장을 쉽고 빠르게 검색할 수 있도록 하는 검색 시스템인 온코서치(OncoSearch)를 개발하였다. 특히 온코서치는 각 문장의 구조를 심층 분석하여 유전자 발현량의 증감 여부와 유전자 변화에 따른 암의 진행 상태를 자동으로 파악하고, 이에 대한 검색 결과를 제공한다. 또한 이렇게 자동으로 파악한 문장내용을 기반으로 해당 유전자의 암 관련 역할을 추론하여 각 유전자를 종양형성유전자(oncogene) 및 종양억제유전자(tumor suppressor gene) 등으로 분류하며, 이에 대한 검색 기능도 제공하고 있다.
이렇게 유전자 발현량 변화, 암 진행 상태 변화, 유전자의 암 관련 역할에 대한 검색을 제공하는 문헌 검색 시스템은 현재 온코서치가 유일하다. 특히 각 유전자가 암 진행에 기여하는 정도를 문장 구조 분석 및 추론을 통해 파악하기 때문에, 기존의 암 관련 유전자 정보 수집 기법들과는 달리 암 관련 유전자 역할에 대한 명시적인 표현(oncogene, tumor suppressor 등)이 없어도 관련된 정보를 파악할 수 있다는 점에서 수월성을 보인다.
문장 구조 파악을 위해서는 첨단 텍스트마이닝 기술인 사건 정보 추출 시스템(event extraction system) 및 최대 엔트로피(maximum entropy) 분류기가 사용되었다. 최대 엔트로피 분류기들은 본 연구팀에서 자체 개발한 말뭉치인 코맥씨(CoMAGC)를 사용해 학습시켰으며, 교차확인(cross-validation) 및 별도 테스트 데이터를 사용한 성능 평가에서 높은 정확도를 보였다. 유전자 역할에 대한 추론을 위해서는 특화된 추론 규칙이 사용되었으며, 이들 추론 규칙의 유효성은 선행 연구를 통해 이미 검증된 바 있다.
개별 사용자들의 검색 결과는 각 문장 분석과정의 신뢰도에 따라 신뢰도가 높은 순으로 정렬되어 제시된다. 또한 유전자 발현량 변화에 대한 표현 간의 공기(co-occurrence) 정보를 바탕으로 암에 특화된 유전자 네트워크 그래프를 구성하여 제공함으로써, 유전자 간 상호작용을 통한 암 발생 기작의 규명을 돕도록 하였다.
3. 기대효과
온코서치 시스템의 개발로 암 관련 유전자에 관한 문헌 정보를 빠르고 정확하게 검색할 수 있게 되어 보다 효율적인 암 연구가 가능해 질 것으로 기대된다. 또한, 대량의 정보를 쉽게 수집, 분석할 수 있게 되어 암 연구의 질적 향상도 기대된다. 특히, 온코서치는 첨단 텍스트마이닝 기술들을 통해 유전자의 암 관련성에 대한 대량의 정보를 수집하는데, 이렇게 수집된 정보는 향후 데이터 마이닝 및 자동 추론 기술 등에 결합되어 암 연구를 위한 신개념 도구로 활용될 수 있을 것으로 기대된다.
연 구 결 과 문 답
|
이번 성과 뭐가
다른가 |
|
뉴라미니데이즈의 잘 알려진
억제제인 타미플루, 리렌자 등과는 전혀 다른 천연물 유래 플라보노이드 화합물과의 복합체 삼차구조를 규명함 |
|
|
|
|
|
어디에 쓸 수
있나 |
|
플라보노이드는 기존의 항균,
항바이러스제에 듣지 않는 내성균에 대한 저해제로서, 천연물 유래 신약개발에 활용이 가능함 |
|
|
|
|
|
실용화까지 필요한
시간은 |
|
항 뉴라미니데이즈 개발에 삼차구조
정보를 바로 사용하여 항바이러스 후보물질을 도출하는데 1년 정도의 시간이 필요할 것임 |
|
|
|
|
|
실용화를 위한 과제는
|
|
플라보노이드 유래 항
뉴라미니데이즈 신약개발을 위한 천연물질 최적화, 동물실험 등의 추가연구가 필요함 |
|
|
|
|
|
연구를 시작한
계기는 |
|
타미플루, 리렌자 등 상용화된 항
뉴라미니데이즈에 대한 내성균에 대한 대책으로, 천연물
유래 플라보노이드의 저해제로서의 기능에 관심을 갖게 됨 |
|
|
|
|
|
에피소드가
있다면 |
|
천연 플라보노이드 화합물의
용해도를 높이는 과정과 삼차구조 해석과정에서 어려움이 커 여러 번 포기하려 하였으나 열정과 노력으로 규명할 수
있었음 |
|
|
|
|
|
꼭 이루고 싶은
목표는 |
|
추가적인 천연 플라보노이드 유래
항뉴라마니데이즈의 연구를 통해 인체 내에서의 독성 문제해결 및 내성균에 대해 저해활성을 가진 천연물유래
신약을 개발하고자 함 |
|
|
|
|
|
신진연구자를 위한
한마디
|
|
연구 중 발생하는 여러 난관에
물러서지 말고, 자연이 숨겨놓은 퍼즐을 슬기롭게 풀어 신비에 감춰진 과학적 의미를 밝혀나가기 위해 노력했으면
함 |
용 어 설 명
1. Nucleic Acids Research지
○ Oxford University Press가 발행하는 생물학분야 국제학술지
2. 유전자 발현량
○ DNA가 RNA 및 단백질로 전사 및 번역되는 양
3. 최대 엔트로피 분류기
○ 최대 엔트로피 원리에 의해 확률 모델을 계산하는 기계 학습 방법을 바탕으로 이종 데이터를 분류하는 프로그램
4. 종양형성유전자
○ 활성화된 경우 암을 유발시키는 유전자
5. 종양억제유전자
○ 정상세포에 존재하면서 그 세포가 암세포화가 되는 것을 막는 유전자. 그 기능을 상실하는 경우 종양이 발생함
6. 사건 정보 추출 시스템
○ 생물학 문헌에서 유전자 발현(gene expression), 조절(regulation), 인산화(phosphorylation), 전사(tranion), 단백질의 세포 내 위치 확인(localization), 단백질 이화(protein catabolism), 단백질 상호작용 등 분자 수준의 사건 정보를 추출하는 시스템
7. 교차확인
○ 데이터의 양이 충분하지 않을 때, 분류기 성능측정의 통계적 신뢰도를 높이기 위해 쓰는 방법
○ 전체 데이터를 k개의 집합으로 나눈 뒤, k-1개의 집합을 이용해서 학습하고 나머지 한 개의 집합을 이용해서 테스트 하는 것을 k번 반복하여 그 평균을 측정
8. 말뭉치
○ 언어 연구 및 언어 처리 시스템 개발 등을 위해 텍스트를 컴퓨터가 읽을 수 있는 형태로 모아 놓은 언어 자료
9. 공기 정보
○ 하나의 문헌 또는 일정한 단위의 글에 두 가지 이상의 단어 또는 표현 등이 얼마나 자주 함께 등장하는 지에 관한 정보
그 림 설 명
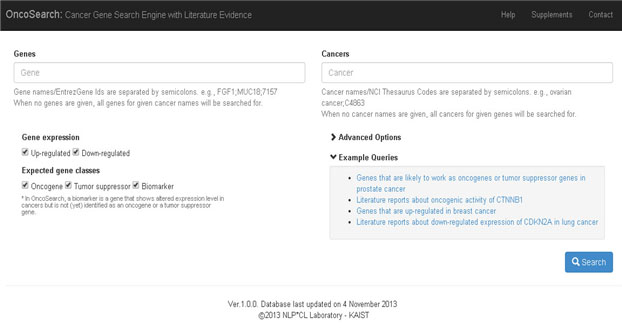
그림 1. 온코서치 웹사이트 검색창. 유전자 이름, 암 종류, 유전자 발현량 변화, 암 관련 유전자 기능 등에 따라 검색을 가능하게 한다.
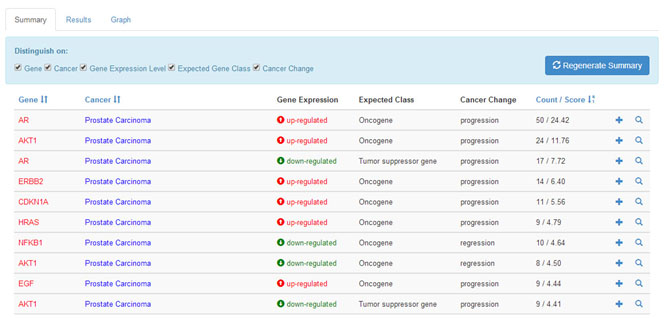
그림 2. 온코서치 검색결과 화면. 유전자-암 관련성에 대한 검색결과를 정확도가 높은 순으로 정렬해서 보여준다.
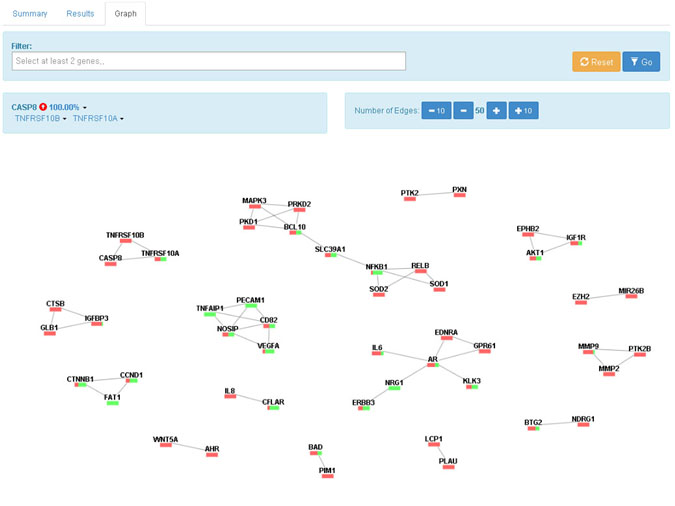
그림 3. 온코서치가 생성하는 유전자 네트워크 그래프. 암에 특화된 유전자 네트워크 그래프를 보여준다. 그래프에서 각 점은 개별 유전자를 나타내며, 점을 잇는 선들은 해당 유전자들 간의 강한 관련성을 표시한다.
관련정보


 암관련+유전자+검색+엔진+개발.hwp
암관련+유전자+검색+엔진+개발.hwp