기술동향
Whole-Genome and Whole-Population 분석을 이용한 진화 공학의 합성생물학적 적용 및 연구 동향
- 등록일2015-06-25
- 조회수6225
- 분류기술동향
-
자료발간일
2015-05-27
-
출처
지능형 바이오시스템 설계 및 합성 연구단
- 원문링크
-
키워드
#Genome#Population#진화공학#합성생물학#연구동향
- 첨부파일
출처 : 지능형 바이오시스템 설계 및 합성 연구단
Whole-Genome
and Whole-Population 분석을 이용한 진화 공학의
합성생물학적 적용 및 연구
동향

아주대학교 이평천 교수
1. 개요
기존의 비교 또는 이론적 방법으로만 접근이 가능했던 진화적, 그리고
생태학적 연구는 빠르게 발전하는 기술의 발달로 인해 점점 더 접근하기 용이해져 가고 있다. 인위적 진화 연구에서, 생물의 전체 수는 환경의
조절에 따라 유지되며 genotype과 phenotype은 짧게는 수십 또는 길게는 수만 세대가 지나는 동안 확인할 수 있다. 미생물을 이용한
연구는 빠른 세대 교체 기간과 냉동 후 재생이 용이하다는 장점이 있어, ancestor와 descendants간의 경쟁적 진화를 유도하거나
과거의 진화 과정의 특정한 시점에서 우연히 발생한 유익한 유전적 진화를 그 시점에서 재 시작할 수 있어 인위적 진화 연구의 주요한 롤 모델로
사용되고 있다. Population genetics 분야는 돌연변이, 재조합, genetic drift, 그리고 자연 선택과 같은 기본적인 진화
과정의 형태에서 진화적 변화의 비율을 설명하기 위해 수학적 모델을 바탕으로 발달해 왔다. 이러한 이론은 진화의 역학과 형태의 일반적인 이해를
도울수는 있으나, 특정 진화의 결과는 발달, 조절, 대사를 조절하는 특정 지놈의 분자생물학적 기능성에 바탕을 두기 때문에 한계가
있다.
최근 DNA 서열 분석 기술의 진보는 종 (species)에 관계 없이
전체 유전자 범위에서 ancestor와 그로부터 유도된 생물 사이의 유전적 차이의 증명을 가능하게 만들었다. 또한 Whole-genome 그리고
whole population 서열 분석은 작은 실험실에서 진화에 대한 자세한 접근이 가능하게 만들었다. 본고에서는 인위적 진화 실험에서
유전적 역동성을 유전 서열 분석 기술이 가장 많이 발달되어 있는 미생물 시스템을 바탕으로 살펴보고, 유성생식 생물계의 유전적 변화를 함께
살펴봄으로써 진화 연구와 대사 공학을 접목시킨 진화 공학으로의 적용 가능성을 이야기 하고자 한다.
2. 인위적 진화 방법의 종류
일반적으로
인위적 진화 연구에서 population을 증가시키는 세가지 방법으로 Mutation accumulation, continuous 배양, 그리고
serial transfer이 있으며, 이 세가지 방법 모두 진화 과정에서 유전적 역동성이 다르게 나타난다. (그림 1)의 위를 보면 각각의
탑에 따라 미생물의 population이 어떻게 유지되는 지에 대한 메커니즘이 설명되어 있으며, 아래를 보면 각각의 방법에 따라 실험의 진행 시
시간의 흐름에 따라 population이 어떻게 변화하는지를 나타내고 있다.
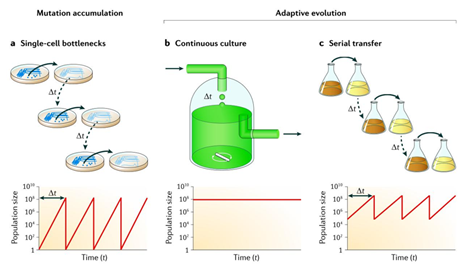
그림 1. 인위적 진화 방법에 따른 population의 변화
Mutation accumulation은 하나 또는 몇 개의 무작위로 선택된
단일 접종을 통한 빈번하고 신중한 population bottleneck들이agar plate에서 단일 세포로부터 자란 미생물 군집의 선택으로
인해 이루어 진다. 이러한 bottleneck들은 유전적 다양성을 제거하고, 무작위적 돌연변이의 적절한 효과와는 관계 없이 고정된다 (그림
1, a). Continuous 배양의 경우 population이 일정한 영양소의 유입과 무작위적 배양액과 폐기물의 배출이 이루어지는 환경으로
인해, 적응 진화가 유도되고, 거의 일정한 크기의 유전적 다양성을 가진 population을 가진 상태로 유지된다 (그림 1, b).
Serial transfer는 일정한 population 비율이 주기적으로 신선한 배지로 옮겨 지고 제한된 영양소가 소진될 때까지 재 배양
한다. 이러한 성장은 풍부한 유전적 다양성이 매 이동 시 유지되기 때문에 적응 진화를 유도한다. 또한 이동은 영양소의 소진 전 이루어 지기
때문에 population의 성장은 끊임없이 이루어 진다 (그림 1, c). Population bottleneck은 선택적 진보의 결과로서
특히 무성생식 population에서 발생하며, 계통간의 경쟁을 통해 유전적 다양성이 줄어들게 된다.
최근 고전적인 long-term mutation accumulation
연구가 Saccaromyces cerevisiae, Arebidopsis thaliana, Drosophila melanogaster,
Caenorhabditis elegans 와 같은 다양한 종을 이용한 돌연변이 발생의 변화를 진보된 whole-genome 서열 분석 기술을
사용해 이루어 지고 있다. 이러한 연구에서 나오는 주요 결론은 자연스러운 돌연변이의 발생 비율은 매우 희박하며, 박테리아와 단일 진핵 세포를
이용한 mutation accumulation 연구에서 돌연변이 발생으로 인한 단일 염기 서열의 변화는 유전자의 매 복제과정 시 10-10 ~
10-9의 비율로 나타남을 확인하는 등 많은 연구가 이루어 지고 있다.
3. 인위적 진화에 따른 mutation과
substitution
살아남은 계통에서 어떤 자연스러운 돌연변이가 발생하고 어떤 유전적 돌연변이가 축적되는지를 구별하는
것은 인위적 진화 연구에서 중요한 일이다. 유전적 서열의 변화는 unrepaired DNA 손상, 중합효소의 오류, 유전적 재조합, 전이인자의
이동, 그리고 유전정보의 전이과정에서 발생하는 오류와 같은 기타 분자 과정들의 조합의 결과이다. 돌연변이는 biological fitness에
그 영향에 따라 유익한, 무해한, 유해한, 치명적인 돌연변이로 분류할 수 있다. Biological fitness란 각기 다른 생존 환경이나
번식 또는 두 가지 모두의 경우에 따라, 즉 표현형과 관계되어 특정 생물종이나 유전형이 후 세대에 기여하는 정량적인
측정이다.
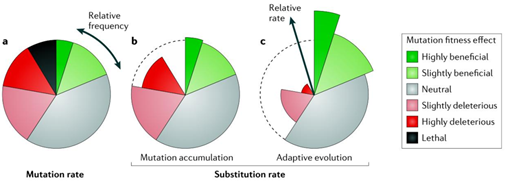
그림 2. Substitution 비율과 mutation 비율의 비교
다양한 생물 종에서 대부분의 환경에서 발생하는 새로운 돌연변이는 무해하거나
유해한 돌연변이가 유익한 돌연변이에 비해 압도적으로 많은 비율을 차지한다 (그림 2, a). 일부 돌연변이는 다른 돌연변이로 인한 형질 특성
변환의 하나의 지표가 되거나 다른 돌연변이의 영향력을 변화 시키기도 하는데, 이는 소위 epistasis라 한다. Mutation
accumulation에서는 전체 population에서 단일 또는 몇몇 bottleneck으로 지속적인 강압이 이루어 지기 때문에 살아남은
세대의 의 형질 특성과 그 효율성은 독립적이다. 그와 대조적으로 adopted evolution의 경우 유익한 돌연변이의 발생으로 인한 유전적
역동성이 강압되기 때문에 유익한 돌연변이의 발생 비율은 실제 돌연변이 발생 비율을 초과한다 (그림 2, c). 유전적으로 갈라지는 계통간의
자연스러운 진화 경쟁population 내에서 돌연변이의 다양성의 확장에 영향을 끼치지만, 살아남은 계통에서 기대되는 돌연변이의 축적은 결국
위에서 명시한 유익한, 무해한, 유해한, 치명적인 돌연변이의 범주를 기초로 동일하게 이루어 진다.
4. 적응 진화 (Adopted evolution)
Adopted
evolution 과정에서 새로운 유익한 돌연변이가 존재하는 유전적, 대사, 그리고 발생에 있어서 일련의 과정들을 최적화 할 때, 시간이
지날수록 종의 형질적 특성은 향상되어 간다. 일부에서는 이러한 돌연변이들의 형질적 특성 사이에서의 관계를 diminishing-returns
epistasis라 한다. 즉, 각각의 유익한 돌연변이는 ancestor의 유전적 배경에서 단일 돌연변이로 나타나게 되면 다른 유익한
돌연변이의 존재 하에서 형질적 특성의 범위를 더 작게 증가시킨다 (그림 3, a). Optimization 기간에 전형적인 유익한 돌연변이는
유전자의 발현을 조절하거나 대사의 흐름을 바꾸고, 대사 조절인자를 조정한다. 진화에서 가장 큰 효과를 나타내는 초기 유익한 돌연변이는 대사
네트워크에서 중심이 되는 대사에 영향을 나타내는 반면, 후에 발생하는 유익한 돌연변이는 특정 대사회로를 중심으로 영향을 나타낸다. 이러한
돌연변이가 발생한 대사 네트워크와 회로가 특정 환경에서 좀더 정교하게 작동하게 되고, 각 돌연변이가 발생하면 발생할수록 종합적인 특성의 향상은
발생하기 힘들어진다.
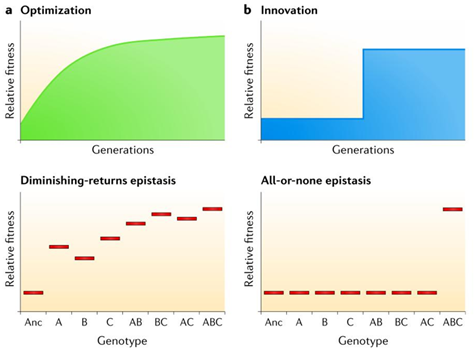
그림 3. 적응 진화에서 Optimization과 Innovation 진화적 차이
좀더 갑작스럽고 극적인 변화는 때때로 인위적 진화 연구에서 발생하는데, 이는 유익한 돌연변이가 한 생물종이 새로운 생태적 지위를 차지할만한 innovation을 만들어 내는 경우이다 (그림 3, b). Innovation은 all-or-none epistasis를 보이는 돌연변이로부터 나타나게 된다. 즉, 몇몇의 효과가 미약하거나 전혀 효과가 없는 돌연변이가 먼저 발생하고, 이러한 변이들은 후에 새로운 표현형이 나타나게 되는 주요한 돌연변이 발생의 배경이 됨을 의미한다. 새로운 표현형이 갑작스럽게 나타나는 이러한 형태의 진화는 독립적인 유전자의 진화와 관계가 있으며, 계속되는 적응 진화와 유전적 다양성의 발생을 위한 새로운 기회를 제공한다.
최근 이러한 인위적 진화에 대한 연구가 유전자 서열 분석 기술의 진보로
whole-genome, whole-population 분석이 용이해 지면서 innovation을 이용해 유용한 특정 대사 산물에 초점을 맞춘
공학적 접근이 시도되고 있다. Long-term evolution experiment (LTEE)를 이용한 인위적 진화를 통해 glucose
가 제한된 환경 하에서 E.coli가 탄소 원으로 일반적으로 이용하지 못하는 에너지원인 citrate를 탄소 원으로 사용 가능하도록 진화
시키거나, 또는 배지 내에서 Na+나 K+와 같은 cation의 농도 조절을 통해 산업적으로 많이 사용되고 있는 젖산의 생산량을 증가시키는 등의
연구가 이루어 지고 있다.
5. 결론
Whole-genome 그리고 whole-population 분석 연구는 다양한 연구
분야에서 유전적 다양성의 이해를 돕기 위해 사용 될 수 있는 가능성을 가지고 있다. 따라서 더욱 많은 유전학, 시스템 생물학, 그리고 합성
생물학 연구에서 발생하는 예상치 못한 유전적 변이의 분석에 적용 가능하며, 미생물학에서는 기존에 밝혀지지 않았던 다양한 형질학적 특성의 작동
원리와 같은 기초 연구의 주요한 도구가 될 수 있다. 또한 인류에 가장 밀접한 질병과 관련되어 다양한 병원균들과 바이러스의 유전학적 특성을
빠르고 정확하게 분석함으로써 질병 발생의 근본적인 원인을 밝히고 특정 생태학적 특징과의 연계성을 밝혀 낼 수 있는 중요한 기술이 될 것이라
기대된다.
6. 참고문헌
1. German P, Cristopher S. Henry &
Dennis V (2015) Long-term phenotypic evolution of bacteria. Nature
517:369-372
2. Ofer F, Amir G, Irine R, Noam S & Nathalie Q.B (2014)
Optimization oflag time underlies antibiotic tolerance in evolved bacterial
populations. Nature 513:418-421
3. Jeffrey E.B, Richard E.L (2013) Genome
dynamics during experimental evolution. Nat Rev Genet 14(12):827-39
4. Omar
E.C, Tristan L, Paulina D.P.B, Ping L, Vincent P.R, Kisten E, Thuy D, David B,
Lin Z, Sang-Joon A, Robert A. B, Adam S, Carlos D. B, Michael J. S (2012)
Evolution and population genomics of the cavity causing bacteria Streptococcus
mutants. Mol Biol Evol 30(4):881-93
5. Bruce R. L, Omar E. C (2009) The
population and evolutionary dynamics of homologous gene recombination in
bacteria. PLOS Genet 5(8):e1000601
6. Fernando B, (2004) From pieces to
patterns: evolutionary engineering in bacterial pathogens. Nat Rev Micro
2:501-518
7. Garland, T. & Rose, M. R. (2009) Experimental Evolution:
Concepts, Methods, and Applications of Selection Experiments (Univ. of
California Press)
8. Kawecki, T. J.(2012) Experimental evolution. Trends
Ecol. Evol. 27:547?560
9. Mardis, E. R. (2008) Next-generation DNA sequencing
methods. Annu. Rev. Genom. Hum. Genet. 9:387?402
10. Schadt, E. E., Turner,
S. & Kasarskis, A.(2010) A window into third-generation sequencing. Hum.
Mol. Genet. 19: 227?240
11. Lind, P. A. & Andersson, D. I. (2008)
Whole-genome mutational biases in bacteria. Proc. Natl Acad. Sci. USA
105:17878?17883
12. Lee, H., Popodi, E., Tang, H. & Foster, P. L. (2012)
Rate and molecular spectrum of spontaneous mutations in the bacterium
Escherichia coli as determined by whole-genome sequencing. Proc. Natl Acad. Sci.
USA 109:2774?2783
13. Sung, W., Ackerman, M. S., Miller, S. F., Doak, T. G.
& Lynch, M. (2012) Drift-barrier hypothesis and mutation-rate evolution.
Proc. Natl Acad. Sci. USA 109:18488?18492
14. Loewe, L. & Hill, W. G.
(2010) The population genetics of mutations: good, bad and indifferent. Phil.
Trans. R. Soc. 365:1153?1167
15. Eyre-Walker, A. & Keightley, P. D.
(2007) The distribution of fitness effects of new mutations. Nature Rev. Genet.
8:610?618
16. De Visser, J. A. G. M., Cooper, T. F. & Elena, S. F. (2011)
The causes of epistasis. Phil. Trans. R. Soc. 278:3617?3624
17. Kondrashov,
F. A. & Kondrashov, A. S. (2010) Measurements of spontaneous rates of
mutations in the recent past and the near future. Phil. Trans. R. Soc.
365:1169?1176
18. Wagner, A. (2011) The Origins of Evolutionary Innovations
(Oxford Univ. Press)
19. Blount, Z. D., Borland, C. Z. & Lenski, R. E.
(2008) Historical contingency and the evolution of a key innovation in an
experimental population of Escherichia coli. Proc. Natl Acad. Sci. USA
105:7899?7906
20. Carroll, S. M. & Marx, C. J. (2013) Evolution after
introduction of a novel metabolic pathway consistently leads to restoration of
wild-type physiology. PLoS Genet. 9:e1003427
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
-
이전글
- GMO 이슈 리플릿 시리즈6 - GM감자
-
다음글
- 세균의 번역 스트레스 반응
지식
- BioINwatch 미국 NIH의 체세포 유전체편집 프로그램 (Somatic Cell Genome Editing, SCGE) 2단계 진입 2023-08-29
- BioINwatch NIH, 체세포 유전체 편집 프로그램(Somatic Cell Genome Editing, SCGE Consortium) 전략 2021-04-20
- BioINwatch 인간 게놈 프로젝트 발표 20주년 기념, 향후 유전체 데이터 정책 방향 제시 2021-04-01
- BioINwatch GenomeAsia 100K Project, 최대 규모의 아시아인 유전체 분석결과 발표 2020-01-06
- BioINwatch 지구 바이오게놈 프로젝트(EBP) 착수 2018-11-21
동향


 Whole-Genome and Whole-Population 분석을 이용한 진화 공학의 ...
Whole-Genome and Whole-Population 분석을 이용한 진화 공학의 ...