기술동향
나노포어(Nanopore) 단분자 탐지의 응용과 기술 개발에 대한 연구 동향
- 등록일2021-07-20
- 조회수4979
- 분류기술동향 > 플랫폼바이오 > 바이오융합기술
-
자료발간일
2021-07-20
-
출처
생물학연구정보센터(BRIC)
- 원문링크
-
키워드
#나노포어#단분자 탐지 응용#단분자 탐지의 기술
- 첨부파일
나노포어(Nanopore) 단분자 탐지의 응용과 기술 개발에 대한 연구 동향
◈목차
1. 서문
2. 본문
2.1. 나노포어의 등장
2.2. 나노포어의 종류
2.3. 나노포어 기술의 적용 분야
2.3.1. DNA 나노기술(DNA Nanotechnology)
2.3.2. 타일 기반 자체 조립 DNA 나노구조(Tile-based DNA Self-assembly Nanostructure)
2.3.3. DNA 접기 기반 자가조립(DNA Origami-based Self-assembly)
2.3.4. 나노포어 단분자 탐지에서의 DNA 나노기술의 응용
2.3.5. 나노포어 통로를 통해 내용물을 운반하는 DNA 자가조립 운반체
2.3.6. 나노포어를 조절하기 위한 DNA 접기 구조체 이용
2.3.7. 나노포어로 직접 작용하는 DNA 조립 구조물
2.3.8. DNA 조립체 기반 나노포어 진단 및 치료법
3. 맺는 말
4. 참고문헌
◈요약문
나노포어 기술은 매우 유망한 단분자 검출 방법으로, 다양한 분석 물질의 식별과 정량화에 이용되고 있다. 이 기술은 제 4세대 염기서열 분석 기법(4th generation sequencing technique)으로써, 별도의 라벨이 필요 없고, 신속하고, 저렴한 기술로 자리 잡을 수 있는 가능성을 지니고 있어 미국 국립보건원(National Institute of Health)이 정한 당찬 포부인, “사람 1명의 총 유전체(whole genome) 시퀀싱 단가를 $1,000 (한화로 약 112만 원)로 끌어내리기”의 실현을 크게 앞당길 수 있을 것으로 보인다.
동시에 나노포어는 검출에 필요한 샘플의 양이 적고, 증폭이나 복잡한 교정이 필요치 않으며, 읽을 수 있는 길이가 매우 길다는(약 1만~5만 basepairs) 장점도 가진다. 나노포어 DNA 염기서열은 잠재력이 무한하지만 아직 극복해야 할 중요한 과제 또한 남아 있다. 이러한 난관이 해결될 수 있다면 나노포어 기술은 DNA 시퀀싱을 뛰어넘어 향후 개인화 의학의 실현에 가장 주도적인 역할을 할 것으로 예상된다. 본 동향리포트에서는 최근의 나노포어 기법과 이와 연관된 DNA 나노기법 등에 대하여 간단히 살펴본다.
키워드: nanotechnology, nanopores, DNA self-assembly, DNA origami, 4th generation sequencing
분야: Biotechnology, Medicine, Nanobio
◈본문
1. 서문
“나노포어(nanopores)”란 나노미터(nm, 10-9 m) 크기의 기공(pore)을 지칭하는 용어이다. 나노포어는 채널 형성 단백질(channel proteins)에 의해 생성될 수 있고, 실리콘(silicone) 이나 그래핀(graphene)과 같은 합성 물질로 이루어지는 경우도 있다. 이 나노포어는 단일 분자 검출기(single molecule sensor)로 사용할 수 있다는 점에서 매우 중요하다. 나노포어를 이용한 단일 분자의 검출 원리는 기공의 양쪽 끝에 전압이 작용하면서 나노포어를 통과하는 이온전류를 감지하는 것이다 [1].
나노포어 단분자 탐지 기술은 분자진단(molecular diagnostics) 분야에서 떠오르는 플랫폼으로써 저렴한 비용과 함께 높은 효율의 정보 처리능력 그리고 따로 라벨링(labeling)없이도 샘플 분석이 가능한 점 등 [2] 기존의 검출법에 비해 여러 장점을 가진다. 이에 따라 나노포어 단분자 검출 분석은 DNA 시퀀싱, 개인의학(personalized medicine), 식품 안전(food safety) [3], 환경 엔지니어링(environmental engineering) [4] 등, 폭넓은 분야에 활발히 적용되고 있다. 전형적인 나노포어 단일 분자 검출 기술에서는 1nm 크기의 기공을 통해 전기장(electric field)을 가하게 되면, 전하(charge)를 띤 분자가 전자기장력(electromagnetic force)을 발생 시켜 나노포어를 통과하는 펩티드 또는 단백질을 번역(translation)하도록 하는 것이다.
이렇게 되면 완충재(buffer)의 전도성 이온(conductive ions)이 나노포어에서 걸러지게 되어 측정이 가능한 전류의 강하(charge drop)가 발생한다 [5]. 이에 따라 통과 시간, 전류 강하, 진폭 등 단백질 번역과정에 관계된 지수들에 따라 다양한 크기와 전하를 띄는 생체 분자를 구분해 낼 수 있다. 본질적으로 나노포어 단분자 탐지 기술은 뉴클레오티드 염기인 A (adenine), T (thiamine), G (guanine), C (cytosine) 사이의 미세한 차이를 구별할 수 있는 높은 민감도(sensitivity)와 해상도(resolution)를 보이게 된다. 주로 유전자 염기서열 분석에 초점을 맞췄던 기존의 나노포어 센싱 기술과 비교해 최근의 나노포어 단분자 검출 기술은 이온(ions), DNA, RNA, 단백질(proteins) 및 기타 거대분자(macromolecules)와 미세분자(micromolecules)의 분석과 같은 폭넓은 분야에서 큰 잠재력을 지니고 있다.
또한, 최근에는 DNA를 이용하여 다양한 자체 조립 구조를 만드는 데 초점을 맞춘 DNA 나노기술이 개발되고 있다 [6]. 다용도 기술로써 DNA 자가조립 구조는 이미 다양한 나노포어 기반 분석에 적용되고 있는데, 예를 들어, 목표가 되는 생체 분자를 포착하여 복잡한 공간적 배열을 이루거나, 약물 전달(drug delivery)에서 매개체 역할을 하는 데 사용될 수 있다. 또한, DNA 나노기술(DNA nanotechnology)를 결합시킬 경우, 나노포어 단일 분자 센싱의 한계점에 대한 돌파구 또한 마련될 수 있을 것으로 보인다.
본 동향리포트에서는 나노포어 단분자 탐지 기법과 이와 연관된 DNA 나노기술 분야 연구에 대한 것이며, 특히 저자는 DNA 나노기술과 나노포어 단분자 탐지기법이 결합된 최근의 연구를 중심으로 떠오르고 있는 핵심 테크닉 몇 가지에 대해 짚어보고자 한다.
2. 본문
2.1. 나노포어의 등장
유전 정보를 암호의 형태로 담고 있는 분자인 DNA는 가히 모든 생명체의 청사진(blueprint)이라 할 수 있다. 정확하고 빠른 DNA 염기서열 기술(sequencing technology, 이하 “seq. ”으로 지칭)이 향후 질병 치료, 특히 개인화 의학(personalized medicine)의 실현에 지대한 영향을 미칠 것임은 확실하다. 현재 상업적으로 판매되고 있는 비-나노포어(non-nanopore) DNA seq. 기술은 데이터 처리를 위해 많은 양의 샘플 준비와 복잡한 알고리즘이 필요하다.
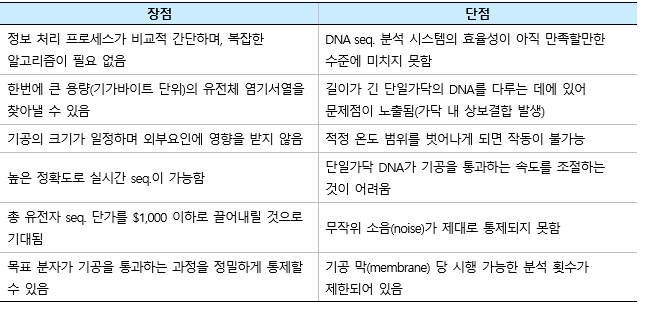
이 기존의 기술은 처리량이 적고, 상대적으로 많은 비용이 들며, 읽기 길이(read length)가 짧다는 등의 한계점을 지니고 있다 [7]. 다행히도 지금은 값싼 DNA seq. 분석기를 개발하기 위한 많은 학술적 및 상업적 발전이 이루어진 상태다. 3세대 seq. 기술 개발 이후 DNA seq. 기술은 이제 단분자 나노포어 기술 시대로 접어들고 있다. 1996년부터는 PNAS (Proceedings of the National Academy of Sciences of the United States of America)에 발표된 최초의 나노포어 논문 [8]을 시작으로 단일 분자에 대한 나노포어 기반 검출이 가장 강력한 seq. 기술 중 하나로 떠올랐다. 나노포어의 중요한 장점은 화학적인 라벨링(chemical labeling)이 필요 없고, 아주 긴 길이의 DNA를 읽는 것이 가능하며(104–106 basepairs), 정보 처리량이 월등히 높다는 것이다 [9](표 1).
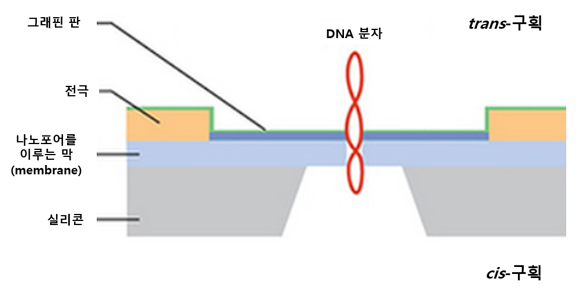
나노포어 기반 기술은 쿨터 카운터(Coulter counter)와 이온 채널(ion channels)에서 비롯되었다 [10]. 외부 전압을 적용하면, 기공 크기보다 약간 작은 크기의 입자가 기공을 통과할 수 있게 된다. 나노미터 크기의 기공은 생물학적 막(biological membrane)에 박혀 있거나, 고체 필름에 형성되어 있어 전도성 전해질을 함유한 저장소(reservoir)를 cis-구획과 trans-구획으로 분리한다 [11]. 전압을 가하면 전해질 용액(electrolyte solution)의 이온은 기공을 전기영동(electrophoresis)의 원리로 통과하게 되면서 이온 전류 신호를 발생시킨다. cis-구획에 형성된 음전하를 띄는(anionic) DNA 분자와 같은 분석 물질에 의해 기공이 막히면 나노포어를 통과하는 전류가 차단되어 현재 신호를 방해하게 된다. 이러한 일련의 과정에서 발생하는 과전류의 진폭과 반응 지속시간 등을 통계적으로 분석하면 목표물 분자의 물리적 및 화학적 특성을 파악할 수 있게 된다 (그림 1).
표 1. 차세대 시퀀싱 기법으로 각광받는 나노포어 seq.과 기존 seq. 방법과의 비교.
2.2. 나노포어의 종류
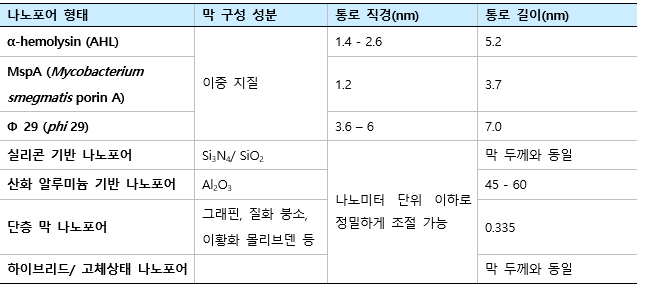
나노포어는 구성하는 재질에 따라 생물학적 나노포어(biological nanopores)와 고체 상태 나노포어(solid-sate 또는 SS nanopores)로 나눌 수 있다 [12]. 생물학적 나노포어는 Φ29 (phi29) [13]와 α-용혈단백(α-hemolysin, αHL) [14] 등과 같은 통로 단백질(channel proteins)로 구성된다 [15]. 굉장히 미세한 직경을 가진 통로(약 1–2 nm) 단백질로 이루어진 나노포어의 경우 단가닥 DNA(single-strand DNA, ssDNA)의 직경과 유사하기 때문에 DNA seq.에 있어 민감도(sensitivity)가 높으면서 정확한 전기 신호를 생성할 수 있다 (그림 2). 높은 재현성(reproducibility) 또한 생물학적 나노포어의 큰 장점이며 휴대성이 극대화된 상용 나노포어 진단 키트(nanopore diagnostic kits) 또한 활발히 개발 중에 있다 [16].
그러나, 생물학적 나노포어는 몇 가지 치명적인 단점이 있다. 예를 들어, 단백질 나노포어의 경우 연구자의 의도에 따라 포어(pore)의 크기와 전하(charge)를 조작하기가(manipulation) 용이하지 않다 [17]. 더욱이 단백질이 이용될 경우 온도나 수소이온 농도(pH: potentiel hydrogène)의 작은 변화조차도 생물학적 나노포어의 순응도(compliance)와 생물학적 활성도(biological activity)에 큰 영향을 미치기 때문에 외부 환경 조건에 더욱 민감할 수밖에 없다.
또 다른 부류의 나노포어인 SS 나노포어는 주로 무기질 재료(inorganic materials)를 이용해 합성하게 된다 [18]. SS 나노포어는 기공의 직경을 효과적으로 조절하여 DNA와 같은 초소분자(micromolecules)부터 분자량이 큰(macromolecules) 단백질까지 광범위한 분자 목표물들을 탐지해 낼 수 있다 (표 2). 그러나, SS 나노포어에 기반한 분자 검출법 역시 난제에 봉착해 있는데, 첫째는 비교적 큰 나노포어의 직경 때문에, 역설적이게도 분석 물질의 통과 속도가 지나치게 빨라 미세분자 검출에 있어 높은 일관도를 보이지 못한다는 점이다 [19].
이를 해결하기 위해 겔(gel) 제제로 이루어진 기질(substrates) [20], 분자 변형을 적용한 나노포어 [21], 나노비드(nanobeads)를 이용한 기공 차단(pore block) [22], 고염도 완충재(hyperosmolar buffers) [23] 등, 다양한 방법이 시도되고 있으나 많이 복잡하며 무작위 소음(random noise)가 증가한다는 것 그리고 일단 합성이 끝나면 수정하기가 극히 어렵다는 단점으로 인해 해당 분야 연구자들의 만족도가 그리 높은 편은 아니다. 또한, SS 나노포어의 막은 두께가 너무 두꺼워 DNA seq.에 사용 시 생물학적 나노포어에 비해 해상도(resolution)가 많이 떨어진다 [24].

표 2. 여러 가지 물질에 기반한 나노포어의 형태.
2.3. 나노포어 기술의 적용 분야
2.3.1. DNA 나노기술(DNA Nanotechnology)
최근 DNA 조립기술(DNA assembly)이 발달하면서 DNA 분자는 유전정보의 매개체 역할뿐만 아니라, 프로그래밍이 가능하다는 장점을 활용하여 통해 인공 나노시스템에서 핵심적인 역할을 수행하고 있다 [25]. 1980년대에 DNA는 컴퓨터 보조 분자 설계(computer-assisted molecular design), 즉 DNA 자가 조립을 통해 연구자가 원하는 나노 구조체(nano-structures)를 형성하여 특성을 파악하였다 [26]. 특정한 DNA 분자 인식을 활용하면 수소 결합(hydrogen bonds), 소수성 상호작용(hydrophobic interaction), 판 데르 발스 상호작용(van der Waals interaction)을 통해 안정적인 DNA 초분자 구조(DNA superstructure)가 자연적으로 생성될 수 있다 [27]( 그림 3).
DNA 구조를 생성하기 하는 데에는 크게 두 가지 방법이 있는데, 하나는 DNA 타일 조립(DNA tile assembly)에 기반하는 짧은 DNA 가닥을 이용하는 것이고 [28], 다른 하나는 긴 DNA 가닥(long DNA strands)을 이용한 DNA 접기 나노기술(DNA origami nanotechnology)이다 [29]. DNA 나노기술은 설계 가능한 나노스케일(nanoscale) 공학, 높은 제조 효율, 간편한 준비 과정 등 많은 장점을 가진 유망한 테크놀로지라 할 수 있다. 이 방법은 최근 생화학, 여러 임상의학 분야, 물질 과학(material science), 나노공학(nanoengineering), 분자 컴퓨팅(molecular computing) 등 다양한 분야에 적용되고 있는데, 특히 DNA 조립구조 기반의 검출/진단법은 상당한 관심을 끌고 있다. 생체공학적으로 설계된 DNA 조립 구조는 다양한 생분자(최대 64개)를 동시에 제어할 수 있으며, 복잡한 분자 정보 처리에 탁월한 성능을 발휘한다 [30].
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
-
이전글
- 고려대학교 의과대학 분자후성유전학 연구실
-
다음글
- 산화적 스트레스와 고혈압


 pdf_0003838.pdf
pdf_0003838.pdf