산업동향
통합구조생물학(Integrative Structural Biology)의 동향
- 등록일2021-03-18
- 조회수4716
- 분류산업동향 > 종합 > 종합
-
자료발간일
2021-03-18
-
출처
생물학연구정보센터(BRIC)
- 원문링크
-
키워드
#통합구조생물학#구조생물학#생물동향
- 첨부파일
통합구조생물학(Integrative Structural Biology)의 동향
◈요약문
통합구조생물학은 기존의 구조생물학 실험을 포함한 다양한 실험적 기법들과 계산 기법을 통합적으로 활용, 생물학적 타겟의 3D 구조 모델을 제시하는 분야이다. 본 동향 리포트에서는 통합구조생물학 모델 구축 방식에 대한 전반적인 내용 및 최근 연구 결과와 함께, 해당 분야에서 선결 과제로 인식되고 있는 부분들에 대해 다루었다.
키워드: 통합구조생물학, 구조생물학, X-ray crystallography, Electron Micros (EM), Mass Spectrometry, bioinformatics, , protein modelling
분야: Structural_Biology, Molecular_Biology, Biophysics
◈목차
1. 서론
1.1. 통합구조생물학의 정의
1.2. 통합구조생물학의 배경
2. 통합구조생물학적 모델 구축 방식
2.1. 구조생화학적 데이터 획득
2.1.1. 실험적 기법을 통한 개별 구조 획득
2.1.2. 실험적 기법을 통한 위치 정보 획득
2.1.3. 계산 기반의 구조 및 위치 정보 획득
2.2. 획득 데이터의 통합적 분석을 통한 모델 구축
2.2.1. 데이터 구성 요소의 ‘representation’
2.2.2. 획득 데이터를 이용한 scoring function 구성 및 평가
2.2.3. 획득 구조 모델의 평가
3. 통합구조생물학 분야의 이슈와 동향
3.1. Archiving 시스템 구축
3.1.1. 단일 구조 모델/ 실험 데이터 deposit을 위한 archiving 시스템 구축
3.1.2. 통합적 구조 데이터 deposit을 위한 archiving 시스템 구축
3.2. 통합적 구조 모델링용 소프트웨어 개발
3.2.1. Integrative Modeling Platform (IMP)
3.2.2. Rosetta
3.2.3. TADbit, PGS (population-based Genome structure)
3.3. 통합구조생물학용 데이터 시각화 및 분석용 프로그램 개발
3.3.1. ChimeraX
3.3.2. Visual Molecular Dynamics (VMD)
3.4. 데이터 형식별 representation, 평가, archiving 기준 정립
3.4.1. 개별적 실험 데이터용 기준 정립
3.4.2. 계산 방식 기준 정립
3.4.3. 통합 구조 모델 평가를 위한 기준 정립
4. 통합구조생물학을 이용해 규명된 구조의 예
4.1. Yeast 핵공복합체의 통합적 구조 규명(2018)
4.2. Genetic interaction mapping 데이터를 이용한 통합적 구조 모델 구축 (2020)
5. 결론
6. 참고문헌
◈본문
1. 서론
1.1. 통합구조생물학의 정의
“통합구조생물학”이란 구조적, 생화학적, 생물리학적 등 다양한 종류의 실험적 기법들과 모델링 등 계산 및 해석 기법을 통합적으로 활용, 원하는 생물학적 타겟의 3D 구조 모델을 제시하는 접근 방식이다.
1.2. 통합구조생물학의 배경
구조생물학은 생체 분자의 구조를 규명하는 학문으로, 구조 규명을 통해 분자의 기능과 메커니즘을 밝혀낼 수 있어 생명과학 분야 전반에 큰 정보를 제공해 왔다.
현재까지 NMR, X-ray 결정학, EM 등의 분석 기술이 발전하며 기존 구조생물학 실험 기법의 한계를 극복하거나, 서로 보완하며 구조생물학의 발전을 견인해 왔다.
그러나, 개별적인 구조생물학 기법에는 각각 한계가 있고, 특히 여러 subunit들이 결합해 있거나 다이나믹하고 heterogeneity가 높은 거대 분자의 구조 규명에는 어려움이 있어 왔다.
통합구조생물학은 개별적 실험 기법의 한계를 극복하고 다양한 종류의 데이터들을 통합, 단일 기술로 얻어낼 수 있는 데이터 및 분석 결과의 한계를 극복하고, 다이나믹한 복합체의 통합적이고 전체적인 모델을 얻어낼 수 있는 기법이다.
해당 모델링을 위해 우선 생화학적, 구조적, 생물리학적 실험 데이터들을 모아서 3D 구조 모델을 만들어내고, 이 모델들을 계산해 데이터에 부합하며 신뢰도가 가장 높은 최종 모델을 도출하게 된다.
이 방식은 전체 데이터셋을 통합하여 각 데이터에 모두 부합하는 모델을 구현하기 때문에 개별적 실험 기법이나 모델링 기법만을 이용한 구조 모델링보다 훨씬 정확도가 높으며, 보다 거시적이고 통합적인 관점에서 구조 모델을 제시할 수 있다.
최초의 통합적 구조 모델 중 하나로 double-strand DNA 구조를 들 수 있다. 이 구조 모델은 X-ray 회절 데이터, 화학적 구성 성분 정보, 뉴클레오타이드 간 결합비, 각 성분 간 stoichiometry 정보를 기반으로 제시되었는데, 각각의 데이터만으로는 DNA가 이중 나선이라고 주장하기에 충분하지 않지만, 이 데이터들을 통합적으로 분석하면 DNA가 이중나선 형태라는 모델에 도달할 수 있는 통합적 접근 방식의 좋은 예이다.
현대 생물학에서는 더 발달된 실험적 기법들과 계산 기법을 이용, 더 복잡하고 정교한 시스템을 통합구조생물학적으로 분석할 수 있게 되었다.
2. 통합구조생물학적 모델 구축 방식
2.1. 구조생화학적 데이터 획득
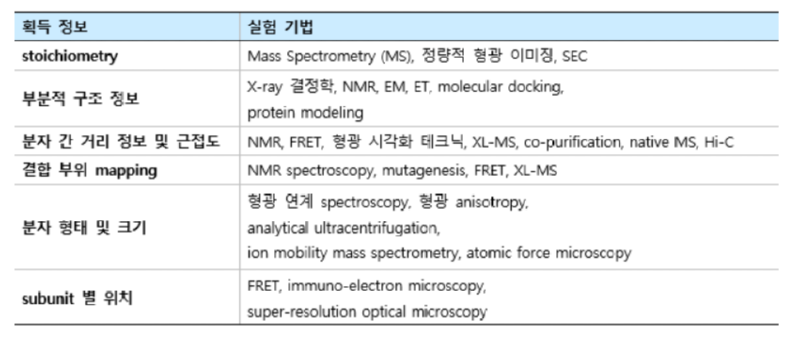
다양한 실험적 기법을 기반으로, 각 서브 유닛 별 구조 정보 및 위치 정보나 서브 유닛 간의 interaction에 대한 정보를 획득, 통합구조생물학적 모델 구축을 위한 기초 데이터로 활용한다 [1, 2].
2.1.1. 실험적 기법을 통한 개별 구조 획득
NMR, X-ray 결정학, EM 등을 이용해 생체 분자의 개별 구조를 획득한다. 각 기법별로 기술적인 한계나 장단점이 다르기 때문에 최종 목적에 부합하는 구조 데이터를 획득해내는 것이 통합구조생물학의 첫 단계라고 할 수 있다.
NMR의 경우 단백질 구조를 높은 resolution으로 규명할 수 있으나, 분석 가능한 단백질의 크기가 작다는 단점이 있으며, X-ray 결정학 역시 높은 resolution으로 다양한 크기의 단백질 구조를 규명할 수 있다는 장점이 있으나, heterogeneity가 높은 단백질 구조를 규명하기에는 기술적으로 한계가 있었다.
최근 급속도로 발전한 cryo-EM 기술의 경우 다양한 크기의 단백질 구조 규명이 가능하나, 규명된 구조의 resolution이 아직 낮고, 일정 크기 이하의 단백질은 구조 규명이 어려우며, 획득한 구조 내에서도 map 위치에 따라 resolution 차이가 크게 난다는 단점이 있다.
최근 기술의 발전으로 규명 가능한 resolution이 점점 높아지고 있으며, 단백질 정제 과정 없이 세포 내에 존재하는 단백질이나 세포 소기관, 혹은 세포 전체를 이미징할 수 있는 cryo-ET 등 새로운 응용 기술이 발전하며 구조생물학 전반에 기여하고 있다.
2.1.2. 실험적 기법을 통한 위치 정보 획득
개별 서브 유닛들이 세포 수준에서 어디에 위치하는지, 다른 서브 유닛들과 어떻게 interaction 하는지 등에 대한 정보를 얻기 위해 MS, SAXS, IP, labelling, co-localization 등의 실험 기법을 이용한다.
이와 관련된 실험 기법 및 획득 가능한 정보에 대해 표 1에 정리하여 나타내었다.
2.1.3. 계산 기반의 구조 및 위치 정보 획득
단백질 서열을 기반으로 한 2차 구조 예측, homology domain 탐색 등을 통해 각 서브 유닛 간의 interaction, 위치 등의 추가 정보를 탐색할 수 있다.
또한, de novo 모델링, homology based 모델링 등을 통해 각 서브 유닛의 구조 모델 정보 역시 추가로 획득할 수 있다.
표 1. 구조생화학적 데이터 획득을 위한 대표적 실험 기법.
2.2. 획득 데이터의 통합적 분석을 통한 모델 구축
2.2.1. 데이터 구성 요소의 ‘representation’
파트 2.1.에서 획득한 각 데이터들을 이용해 구축할 모델의 ‘representation’을 정의한다. 해당 ‘representation’은 모델의 구성 요소들을 명확하게 설명할 수 있으면서 전산 해석 역시 가능하도록 구성되어야 한다.
예를 들어, 구조 모델의 representation은 원자, 보다 덜 정교한 수준의 분자, 혹은 복합체 속 각 서브 유닛의 종류나 카피 넘버와 같은 구성 요소들을 명시하면서, 각 구성 요소들의 위치, 오리엔테이션, 형태 등의 정보를 잘 설명할 수 있어야 한다.
또, representation을 정의할 때 담긴 정보의 종류와 양 그리고 변수가 이용한 데이터의 종류와 구축 중인 모델에 알맞도록 정의해야만 한다.
또한, domain 정보, MS 데이터, density map이나 localization 정보 등의 데이터를 이용해 각 서브유닛의 위치나 interaction에 기반한 위치 정보 등을 공간적 제한 요건으로 정의, 모델 구축 시 해당 제한 요건을 충족하도록 하여 모델의 불확실성을 최소화할 수 있도록 한다.
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
관련정보


 pdf_0003739.pdf
pdf_0003739.pdf