BioINwatch
(BioIN + Issue + watch) : 바이오 이슈를 빠르게 포착하여 정보 제공4개 대륙, 180여 종 야생동물의 장내 마이크로바이옴 다양성과 기능 규명
- 등록일2021-04-29
- 조회수4843
- 분류생명 > 생명과학
-
발간일
2021-04-29
-
키워드
#동물#마이크로바이옴#animals#microbiome
- 첨부파일
-
차트+
?
차트+ 도움말
BioINwatch(BioIN+Issue+Watch): 21-30
4개 대륙, 180여 종 야생동물의 장내 마이크로바이옴 다양성과 기능 규명
◇ 인간 마이크로바이옴에 비해 연구가 미비한 동물 마이크로바이옴 관련하여 최근 네이처에 4개 대륙, 6개 지역에서 야생동물의 대변 샘플에서 그동안 알려지지 않은 약 1,200종의 미생물을 발견하고 5,000개 이상의 유전체 데이터베이스를 구축한 논문을 발표
▸주요 출처 : Nature, Diversity and functional landscapes in the microbiota of animals in the wild, 2021.04.
▣ 광범위한 인간 마이크로바이옴 특성 규명에 비해 야생동물에 대한 마이크로바이옴 특성 연구는 미흡한 상황
○ 인간 마이크로바이옴(Human Microbiome)은 지난 20년간 전 세계적으로 다양한 공동연구를 통해 인체에 서식하는 미생물을 파악
- 미국 중심의 ‘Human Microbiome Project(HMP)’ 및 EU(Metagenomics of the Human Intestinal Tract, MetaHIT), 캐나다(Canadian Microbiome Initiative), 일본(Human Metagenome consortium) 등에서 인간 마이크로바이옴 관련 국제 컨소시움 및 개별국가 차원의 다양한 프로젝트를 추진
○ 인간과 밀접한 관계가 있는 야생의 동물은 썩은 고기나 병원균에 감염된 독성 음식으로도 생존할 수 있으며 다양한 질병에 대한 면역력을 지녀 이런 특성들은 마이크로바이옴 때문일 수 있지만 우리는 아직 야생동물 마이크로바이옴의 다양성과 기능에 대한 이해가 부족한 상황
※ 그리폰 독수리(griffon vultures)의 장에서 썩은 먹이에 존재할 수 있는 독소를 분해할 수 있는 이전에 설명되지 않은 프로테아제를 확인
▣ 이스라엘과 미국 연구자들을 주축으로 야생동물의 장내 미생물 다양성을 조사하기 위해 4개 대륙, 6개 지역에서 다양한 샘플을 채취
○ 이스라엘 와이즈만연구소와 미국 스탠포드대 연구자들은 어류, 파충류, 조류, 포유류를 포함한 184종의 야생동물에서 406개 대변 샘플을 채취하고, metagenomics로 야생동물의 장내 마이크로바이옴을 분석
- de novo 메타게놈 어셈블리를 사용하여 이 중 75%가 이전에 알려지지 않은 박테리아 종으로서 1,209개의 박테리아 종으로부터 5,000개 이상의 게놈 데이터베이스를 구축
※ 동물은 주로 육상 척추동물이지만 다양한 먹이 패턴과 행동 특성을 보유
< 1,209개 게놈 정렬 기반의 계통 발생 트리 >
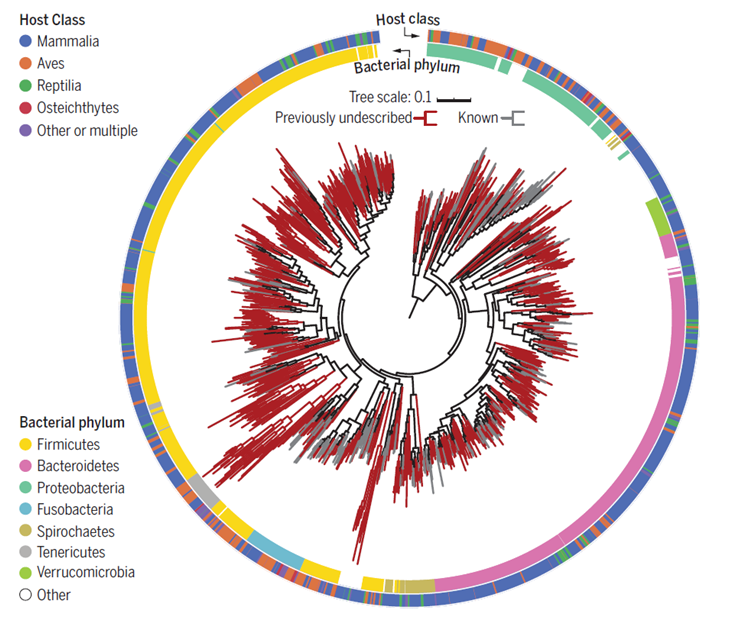
¶ 내부/외부 색상 고리는 각각 세균 문 및 숙주 등급을 나타내며 이전에 확인되지 않은 게놈은 진한 빨간색으로 표시
출처 : Nature, Diversity and functional landscapes in the microbiota of animals in the wild, 2021.04.
▣ 연구진들은 전체 메타게놈 서열분석을 사용하여 다양한 숙주종에 대한 장내 미생물을 조사하는 등 샘플수집 및 분석전략에 3가지 사항 적용
○ 첫 번째로는 포획이 여러 동물 종의 미생물 군집을 변화시키는 것으로 나타나 이러한 샘플링이 제기하는 많은 어려움에도 불구하고 야생동물의 샘플링에 집중
○ 두 번째로는 야생동물의 광범위한 다양성을 확보하기 위해 4개 대륙에서 다양한 특성과 먹이 패턴을 가진 다양한 동물을 샘플링
- 이를 통해 활동 시간 및 사회 구조 등을 포함한 각 종의 특성을 직접 선별하여 미생물 구성과 숙주 표현형 간의 관계를 체계적으로 연구하는 것이 가능
○ 세 번째로는 메타게놈 어셈블리 파이프라인을 조정하고 조립된 게놈에 분류학적 및 기능적으로 주석을 달아 야생생물의 미생물 환경을 나타내는 광범위한 게놈 컬렉션을 생성
< 코호트 연구 및 설계 >
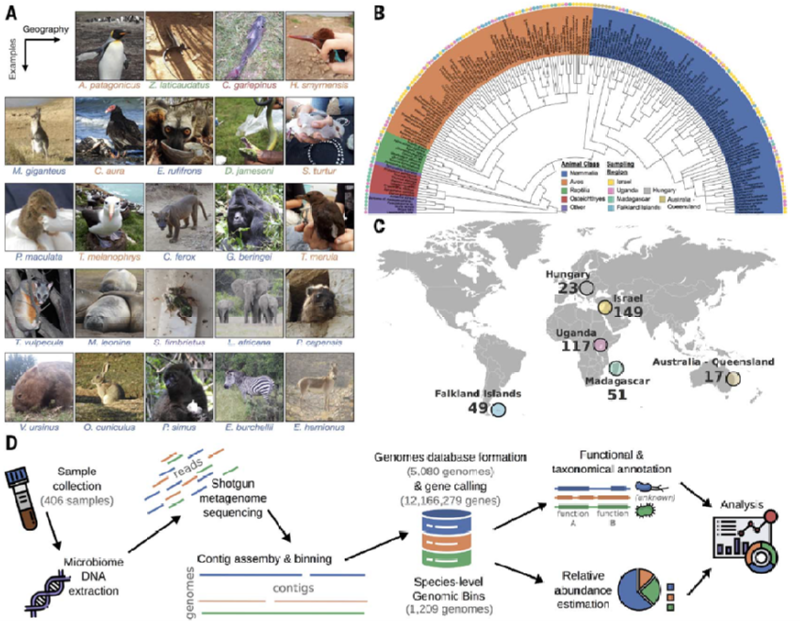
출처 : Nature, Diversity and functional landscapes in the microbiota of animals in the wild, 2021.04.
○ 전체 메타게놈 시퀀싱은 16S 리보솜 RNA 유전자와 같이 특정 조각을 선택하는 대신 샘플에 존재하는 모든 DNA 시퀀싱을 포함한다는 점이 특징
- 이러한 접근방식의 주요 이점은 메타게놈 조립 게놈(Metagenome Assembly Genome, MAG)이라고 하는 전체 또는 부분 게놈 시퀀싱 판독 자체를 정렬 및 연결로 재구성할 수 있어 이 과정을 ‘조립 및 비닝(binning)’이라고 함
▣ 동물의 미생물 군집은 항생제, 산업용 효소 및 면역 조절제를 포함,바이오산업에 영향을 미칠 수 있는 풍부한 원천 자원
○ 야생동물의 미생물군은 동물과 인간 모두 병원체의 자연 저장소로서,
- 야생동물의 병원체 저장소를 매핑한 최근 연구에서 인간 병원체 역학 감시 및 예측에서 이 접근법의 잠재력을 입증
○ 또한 이 매핑을 통해 현재 코로나19 대유행과 같이 인간 집단으로 전파되는 시기와 경로를 설명할 수 있을 것이며,
- 더불어 야생동물의 미생물군을 매핑하는 것은 생물종 보존 노력에 도움이 될 것으로 기대
○ 무엇보다 본 연구의 가장 큰 기여는 체계적으로 분석·생성된 야생동물의 메타게놈 데이터
- 본 연구는 야생동물의 장내에서 추출한, 주로 알려지지 않은 종의 대규모 주석이 달린 박테리아 게놈 데이터베이스를 제시하였으며,
- 동물의 특성 및 분류에 다양한 미생물 패턴을 식별하고 새로운 산업용 효소와 치료제의 발견을 위한 미개척 자원으로 그 잠재력을 강조
- 이러한 메타게놈으로 이루어진 발견에 힘입어 미생물 보존과 항생제 내성 등 다양한 분야에서 새로운 돌파구가 마련될 수 있음을 언급
▣ 연구진은 위와 같은 가능성과 동시에 어셈블리 기반 접근방식의 한계를 언급하며, 방대한 데이터에 대한 가설을 테스트하기 위해 많은 계산, 해석 및 실험적 검증이 필요하다고 강조
○ 어셈블리 기반 접근방식은 ‘조립 및 비닝’에 의해 소량의 미생물과 사이즈가 큰 게놈을 가진 미생물은 과소 표시될 가능성이 있는 등 포괄범위에 편차가 있고, 제시된 야생동물 집단은 동물 생명나무 전체를 균일하게 커버하지 못하는 등의 한계 존재
- 또한 짧은 읽기(short reading) 기반 접근방식은 비교적 덜 복잡한 게놈 영역을 재구성하는 데 한계가 있었음을 언급
○ 이에 연구진은 대부분의 특성화되지 않은 미생물에 대한 미래 연구 방향을 제시하고, 다양한 실험적 검증이 필요하다고 강조
- 흥미로운 미래 연구로 야생동물의 미생물이 독성물질을 분해하는 데 어떻게 도움이 되는지, 먹이 내 병원균으로부터 어떻게 방어하는지, 다양한 영양 공급원에서 영양분을 추출하는 방법에 대한 연구를 제시
- 또한 숙주에서 어떻게 작용하고 있는지, 일시적으로 위장관을 통과하는지 여부 등에 대해서도 연구의 필요성을 언급
지식
- BioINpro [KRIBB 워킹그룹] 마이크로바이옴 기반 장 면역 조절 연구 동향 2024-12-16
- BioINpro [KRIBB 워킹그룹] 첨단의약품 개발을 위한 오가노이드 기술 2024-11-07
- BioINwatch 동물 감염병 및 인수공통감염병 제어를 위한 자가 전파형 백신(Transmissible Vaccines) 2024-10-24
- BioINpro [2024 바이오 미래유망기술(상)] 마이크로바이옴 표적 항암백신 전략 2024-05-13
- BioINwatch 숙주-미생물 상호작용체(interactome) 아틀라스 2024-04-17
동향


 BioINwatch21-30(4.29)●4개 대륙 180여 종 야생동물의 마이크로바...
BioINwatch21-30(4.29)●4개 대륙 180여 종 야생동물의 마이크로바...
 1,209개 게놈 정렬 기반의 계통 발생 트리
1,209개 게놈 정렬 기반의 계통 발생 트리