BioINpro
(BioIN + Professional) : 전문가의 시각에서 집필한 보고서 제공미생물 유전체 연구동향
- 등록일2016-01-26
- 조회수20788
- 분류생명 > 생명과학, 플랫폼바이오 > 바이오기반기술
-
저자/소속
김지현 부교수, 미생물유전체전략연구사업단 단장, 송주연, 연구교수/연세대학교 생명시스템대학
-
발간일
2016-01-26
-
키워드
#유전체
- 첨부파일
-
차트+
?
차트+ 도움말

1. 미생물 유전체 연구의 중요성
미생물의 존재가 인식되고 난 이래, 미생물에 대한 연구를 통해 질병, 대사 등 지구 환경에서 미생물들의 역할들이 알려지고 있지만, 아마도 현재까지 인간이 알고 있는 미생물에 대한 지식수준은 눈에 보이지 않는 미생물의 크기보다도 미미할 것으로 예상된다. 세균의 경우, 지구상에 약 5×1030 개의 세포들이 존재하며, 세포 구성 탄소량은 350-550 Pg(1 Petagram = 1015 g)에 이르는 것으로 추정되므로(Whitman et al., 1998), 보이지 않는 미생물의 존재감 및 영향력을 가늠할 수 있다. 이러한 미생물들은 대사 작용을 통해 지구 환경 생태 및 대사 순환에 이르기까지 영향을 주고 있으며, 다른 유기체와 공생, 기생, 협력, 경쟁 등을 통하여 긴밀한 상호작용을 하고 있다. 또한 다양한 대사체 및 효소 활성은 산업적으로도 중요성을 지닌다.
한편, DNA 및 RNA와 같은 세포 내 유전물질들이 사람의 손에서 다뤄질 수 있게 되고 유전자 기능 연구가 가능해지면서 유전자 정보는 모든 생물 연구와 활용에 가장 기초가 되는 자료가 되었다. 또한, 유전학적 기술의 진보와 함께 한 생명체 내에 모든 유전 정보 총체 즉, 유전체의 전 염기서열 정보를 해독하고 그 안에 포함된 각각의 유전 정보들의 수집이 가능한 기술들이 개발되면서 모든 생물체 및 생명현상 연구에 획기적인 기회를 가져다주었다. 특히, 세균의 유전체는 다른 고등생물 및 식물 유전체에 비해 작은 크기로 존재하기 때문에 생명 연구의 모델이었을 뿐 아니라, 유전체 해독 기술 또는 분석 기술의 개발의 전환 시 연구의 모델로 제시되어왔다.
미생물을 대상으로 한 연구는, 생명 현상 및 기작에 대한 이해를 돕고, 보건, 의학, 환경, 산업 분야에의 활용으로 이어지므로, 미생물의 유전체 분석을 통해 각각의 유전자의 기능들을 파악하는 연구가 많이 진행되어 왔다. 이를 위해 미생물 유전체 연구는 단일 유전체, 즉 하나의 클론(clone)에서 나온 미생물 세포의 유전체 해독 연구들이 주로 이루어졌다. 이후, 메타유전체(metagenome; 메타지놈 또는 메타게놈이라고도 함)의 개념이 도입되면서 광범위한 염기서열 해독을 통한 특정 환경 내의 모든 미생물 유전체 서열 정보를 확보하기 위한 서열 기반의 메타유전체 연구가 이뤄지고 있다. 특히, 최근 차세대 염기서열 해독 기술(next-generation sequencing, NGS)의 발전으로 인해 이러한 연구들이 가속화되었으며(그림 1), 미생물 연구의 새로운 전성기가 시작되었다고 해도 과언이 아니다. 미생물의 유전체 및 활용 연구는 전 세계적으로 지속, 확대될 것이 예상되고 있다.
미생물 단일 유전체 및 메타유전체 연구로부터 얻어진 기능성 유전자의 확보 및 생명현상 원리의 이해는 학문적, 산업적으로 매우 중요하다. 병원성 미생물 유전체 연구를 통해 의약학적 질병 진단 및 제어의 지식 기반이 되며, 질병 마커 유전자 개발을 통한 산업적 중요성이 있다. 또한 특이적 대사체 및 효소 개발을 통해 바이오 신소재를 발굴하며, 환경 생태학적 모니터링을 위한 자료를 수집하고, 기후변화 및 생태계 변화 등에 대처하기 위한 방안을 위한 기반을 구축할 수 있다. 이러한 개발 가능성은 과학 선진국 및 신흥 국가들이 중요성을 인지하고, 개발에 막대한 투자를 실행하고 있다. 연구 기술 발전 도모 및 소재 확보를 위해 유전체를 통해 얻어진 정보를 이용하여 개발된 생산물들을 각종 바이오 시장 선점하기 위함이다. 더구나 미생물 유전체 정보로부터 새로운 기능 유전자 발굴 및 유전자간 상관관계를 연구하기 위한 기반이 빠르게 변하고 있다. 특히, 대용량의 유전체 정보를 효율적으로 처리하는 기술은 빠른 소재 발굴 기회 확보로 이어질 수 있으며, 이는 학문적 성과 뿐 아니라 산업적 진보에 긍정적으로 영향을 미칠 수 있다. 따라서 국내외 연구 동향에 대해 분석하고 미생물 유전체 및 메타유전체 연구를 위한 방안 및 대처를 모색해보고자 한다.
(A)
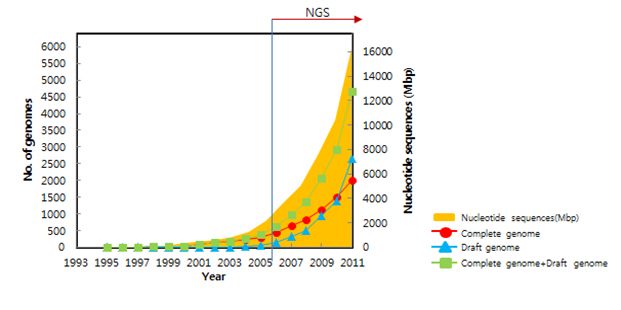
(B)
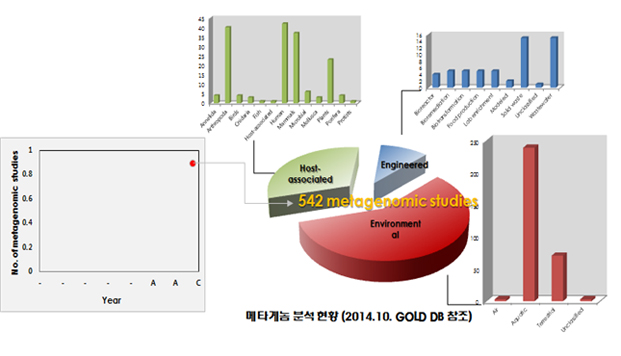
[그림 1. 전 세계적인 미생물 유전체 및 메타유전체 해독 건수 및 분석 경향 (updated form Song et al., 2012)]
2. 연구 동향
(1) 염기서열 해독기 개발 및 유전체 연구
1970년대에 개발된 프래드 생어에 의한 DNA 서열 해석 기법 개발과 자동화기기의 개발은 유전학 연구 및 인간 유전체 염기서열 해독 프로젝트까지 생물학 분야에 큰 기여를 하였다. 인간 유전체 염기서열 해독 프로젝트를 통한 연구 개발에 막대한 투자와 관심은 유전체 서열 조각을 조립할 수 있는 알고리즘 및 프로그램들의 개발과 새로운 염기서열 분석기기가 개발되는 것으로 이어졌다.
제 2세대 염기서열 분석은 대규모 병렬 염기서열 해독(massively parallel sequencing)으로써, 나노사이즈의 공간에 증폭된 DNA의 서열 신호를 읽을 수 있는 방법이었고, pyrosequencing 기술을 적용한 454 Life Sicence의 GS20 기기가 첫 번째 상용화되어 출시되었다. 그 이후, Illumina/Solexa의 GA 및 HiSeq series, Life technologies SOLiD와 Ion Torrent 들의 기기들이 출시되어 유전체 및 전사체 등의 분석에 활용되어왔다. 이후, 증폭을 거치지 않는 단분자실시간시퀀싱(single- molecule real-time sequencing) 기술이 개발되면서 DNA 중합효소와 시퀀싱 기술을 접목한 Pacific Bioscience의 PacBio RS 시스템이 출시되었으며, 최근 본격적인 상용화는 되지 않았지만 Oxford Nanopore Technologies 사의 Nanopore가 가장 긴 sequencing read를 생산하는 서열분석기로 소개되었다.
제 3세대 염기서열 분석기기 시대가 되면서 long read의 생산이 가능해지게 되었다. 아직 제 3세대 기기들이 데이터 생산량이 제 2세대 기기들에 못 미치므로, 메타유전체 해독과 같은 큰 사이즈의 유전체에는 바로 적용을 못하고 있지만, 단일 미생물 유전체 해독에 많은 진보를 이루었고, 제 2세대에 의한 해독 서열의 scaffolding 및 hybrid assembly에 활용되고 있다(Boetzer M et al., 2014). 이러한 대용량 유전체 서열 해독기기와 소프트웨어의 개발 및 데이터 처리 최적화로 미생물 단일 유전체 해독률 및 정확도가 높아짐에 따라 과거 유전체 해독의 병목이었던 미생물 유전체 해독 및 조립 과정이 단순화되고, 기능성 유전자의 발굴은 해독 대상 유전체의 선정 및 분석 기술에 달리게 되었다.
이를 위해서는 빅데이터 분석 및 정보 처리 기술 즉, 생물정보학적 기술이 중요시 되었다. 특히 메타유전체와 같은 대용량 서열이 생산되는 연구 분야에서는 이러한 연구자의 연구 역량과 함께 컴퓨팅 시스템과 같은 인프라가 중요한 연구 요소로 여겨지게 되었다.
(2) 미생물 유전체 연구
미생물 유전체 분석 결과를 통해 유전체의 특성을 분석하고 생리학적 특성을 서열 정보 수준에서 이해할 수 있다. 이 과정에서 유전자의 기능이 유추되는 한편, 기능성 유전자, 병원성 유전자 등 균주 또는 종 특이적 유전자를 선별하여 서열이 해독된 미생물의 특성을 파악할 수 있게 된다. 이러한 서열 분석 결과는 유전자의 세포내 분자 수준의 기능 연구 및 새로운 기능 유전자 발굴에 이용될 수 있다. 이러한 분석 결과는 유전체 비교 분석을 통해서 도출될 수 있는데 미생물 특정 그룹이 공통적으로 가지고 있는 코어 유전체와 전체 유전자 풀을 포함하는 팬-유전체 분석 방법이 많이 이용되고 있다. 코어 유전체와 판-유전체의 개념은 2005년 Tettelin 등에 의해서 제시되었고(Tettelin et al. 2005), 이러한 분석은 추후 PGAP, PanSeq, OrthoMCL과 같은 유전체 비교 분석 프로그램을 이용하여 분석되고 있으며 그 결과는 종, 또는 그룹 특이 유전자를 분류하는데 유용하다.
기능적 분석 외에 유전체 서열 분석을 통해 유전적인 진화 관계를 설명할 수 있다. 세균의 경우, 16S rRNA 서열이 종을 분류하는데 많이 이용되나, 특정 균주, 종, 그룹 단위의 유전체 수준에서 계통유전체학적 분석을 수행할 수 있다. 전 유전체 서열이 이용 가능한 경우, multilocus sequence typing(MLST)을 이용하거나 코어 유전체의 유전자를 이용하여 계통학적 분석을 수행할 수 있다. 이러한 분석 결과는 유전체학적 진화 경로를 알아내는데 사용된다(Kwak MJ et al., 2015).
최근 미생물 비교 유전체 연구는 같은 종 내에서 균주 단위의 resequencing 방법을 이용하여 이뤄지기도 하며, 조립된 유전체 서열 사이의 단일염기 다형성, 도입, 결실 유전자를 찾거나, sequencing read를 가지고 참조 유전체 서열에 대해 mapping을 수행함으로써 변이 위치 및 변이 서열을 탐색하여 유전자 기능의 변화를 추적할 수 있다(Marvig RL et al., 2015).
유전체 서열은 유전자들의 기능적 상관관계 및 네트워크를 분석하기 위한 자료로 많이 사용된다. 유전체 서열은 유전자 기능 연구를 위한 가이드가 되기도 하는데 최근 전사체를 비롯한 각종 오믹스 연구가 NGS를 통한 분석 기술들이 개발 되었으며, 따라서 전사체, 대사체, 단백질체 분석 등 오믹스 분석 기반으로 활용될 수 있다.
이러한 미생물 유전체 연구들은 주로 비교 유전체를 중심으로 연구되고 있으며, 그 분석대상은 병원성 미생물부터 산업적 응용이 가능한 미생물, 그리고 환경 생태학적으로 중요한 미생물까지 다양해졌다. 병원성 미생물의 경우 유전자 변이 및 신규 병원성 유전자의 발굴 분석이 대상이 될 수 있으며, 이러한 결과는 병원성 미생물 및 유전자의 새로운 병원성 기작을 알아내거나 이를 제어하기 위한 연구에 활용될 수 있다. 유전체 서열 발굴 및 균주 특이적 유전자의 발굴은 산업적 유용 미생물의 연구에도 활용될 수 있는데, 의약학적으로 활용 가능한 신규 화합물 생산 유전자 발굴 및 식품미생물의 활용에 가능한 유용 기능성 유전자의 발굴을 통한 생물자원 확보(유산균, 식품 발효 효모 등) 등을 통해 다양한 고부가가치 산업과 연결시킬 수 있다. 특정 기주와 공생하는 미생물의 경우 생물 종간 상호작용의 연구 모델이 되거나 생태 또는 생리학적으로 중요한 학문적 자료가 될 수 있다.
...................(계속)
* 로그인 하셔야 자세한 정보를 모두 보실 수 있습니다.
-
이전글
- 유전체 교정 기술
-
다음글
- 뇌-컴퓨터 인터페이스 기술 연구동향
지식
- BioINwatch 최대 규모의 오픈소스 Evo2를 활용한 유전체 모델링 및 설계 2025-03-21
- BioINwatch 후성 유전체 비트(Epi-bits)를 활용한 DNA 기반의 데이터 저장 기술 2025-03-18
- BioINwatch 작물 중개 유전체학에 거는 기대와 도전 과제 2025-01-24
- BioINregulation 유전체 데이터 2차 활용 관련 국가별 제도 비교 2025-01-24
- BioINwatch 시행 가능성이 높아진 미국의 생물보안법(US Biosecure Act), 바이오산업 공급망 재편 압박 2024-12-04
동향


 미생물 유전체 연구동향.pdf
미생물 유전체 연구동향.pdf
 전 세계적인 미생물 유전체 및 메타유전체 해독 건수 및 분석 경향
전 세계적인 미생물 유전체 및 메타유전체 해독 건수 및 분석 경향