BioINpro
(BioIN + Professional) : 전문가의 시각에서 집필한 보고서 제공유전체 편집 기반 양적 형질 조절기술 (Quantitative trait loci(QTL) Engineering)
- 등록일2018-05-29
- 조회수13650
- 분류플랫폼바이오 > 바이오기반기술
-
저자/소속
박순주 조교수/원광대학교 생명과학부
-
발간일
2018-05-29
-
키워드
#유전체 편집
- 첨부파일
-
차트+
?
차트+ 도움말

1. 기술의 정의
유전체 편집기술의 발전은 가축과 농작물에서 양적형질 활용의 폭을 넓힐 수 있는 탁월한 도구가 되어 미래 인류를 위해 먹거리 혁명을 이끌 것으로 기대된다. ‘유전가 가위’로 알려진 ‘CRISPR/Cas9 시스템’은 박테리아가 진화적으로 획득한 항바이러스 면역시스템의 원리를 이해하고 응용하여 다른 생물체에 유전공학적으로 유전체 편집이 가능하도록 개발된 최신 유전체 편집기술이다[1]. 이 기술의 활용범위는 형질전환이 가능한 대부분의 농작물과 가축의 유전체에 이르며, 이 기술의 기능은 유전체 내부 유전자에 염기서열 결실 또는 염기서열 삽입 등을 아주 정교하고 정확하게 유발할 수 있도록 특성화 되고 있다. 최근에는 유전자 가위로 특정 염기를 정확히 치환시킬 수 있는 기능이 개발되어 보고되고 있다. 이 기술은 마치 성능과 속도가 매우 뛰어난 컴퓨터 하드웨어와 같아서, 어떤 소프트웨어를 사용하는가에 따라 엄청난 효과를 발휘할 수 있다. 여기서 인간이 길게는 수십년 동안 짧게는 수년 동안 연구해 온 양적형질인자 개발과 양적형질인자 기능연구가 엄청난 효과를 나타낼 소프트웨어가 될 수 있음을 제시하고자 한다.
인간은 자연에서 만들어진 야생유전자원으로 부터 다양한 양적형질과 질적형질을 활용해 왔다. 이 형질들의 개선은 인간 삶에 다양한 풍요로움을 제공할 수 있다. 즉, 우유, 고기, 곡식 등에서 지속적인 양적/질적형질을 개선함으로써 인간사회에서 삶의 풍요를 제공할 것이다. 인간이 활용해온 유용형질은 크게 질적형질(Qualitative traits)과 양적형질(Quantitative traits)로 구분할 수 있는데, 질적형질은 유전적으로 명확히 구분되는 자손형질 즉, 불연속된 표현형질을 말하며, 이는 소수의 유전자의 변이에 기인하므로, 단순한 유전법칙들을 적용하여 관련 유전변이를 확인할 수 있다. 반면, 양적형질은 환경적, 유전적 요소에서 여러 변이들이 특정형질에 부분적으로 작용하여 나타나는 연속적이고 계량적으로 표현되는 형질을 말한다. 다수의 유전자에 발생된 변이들은 크고 작은 표현형적 효과를 나타내며, 유전적으로는 복합적인 유전효과가 중복되어 있고, 환경적인 영향도 함께 나타나게 된다. 따라서 양적형질의 유전연구를 효과적으로 수행하고 활용하기 위해서는 환경적 영향을 제거하고 복합적인 유전효과를 유전체에서 유전자 범위로 단순히 나타내는 방법들이 필요한데, 양적형질 매핑(Quantitative Traits Locus Mapping/QTL mapping)과 Genome-Wide Association Study(GWAS)등이 양적형질 유전자들을 분리하는데 중요한 도구가 되고 있다[2, 3].
양적형질 매핑(QTL mapping)은 20세기 말에 유전체 범위로 염기서열 정보를 얻을 수 있게 되면서 염색체 위치별 마커들을 이용하여 표현형질과 마커들 간의 분리비를 통계적으로 분석할 수 있었고, 이를 통해 양적형질의 유전자 좌위를 대략적으로 확인할 수 있게 되었다. 즉, 양적형질을 나타내는 유전자들은 매핑집단에서 마커기반 linkage mapping결과로 염색체 부분을 지정할 수 있으며, 양적형질은 나타내는 F2개체와 여교배를 수행하여 양적형질 유전인자를 단순화 시킨 Recombinant Inbreeding Lines(RIL) 또는 Introgression Lines(IL)을 육성함으로써 양적형질 변이인자를 유전자 범위로 정확히 분리할 수 있다. 이들 변이인자들은 다른 양적/질적인자들과 유전공학적으로 병합하여 양적형질들을 인위적으로 유전적으로 증진시키고자 하는 QTL 피라미딩을 수행하는데 중요한 유전재료가 된다(그림 1).
GWAS(Genome-Wide Association Study)는 글로벌 규모로 수집된 야생종, 육성품종들의 표현형질들과 유전체를 직접비교 분석하여 양적형질인자를 연구할 수 있는 길을 열었다. 차세대 염기서열 분석기술(Next Generation Sequencing/NGS)은 GWAS를 양적형질인자 분리 연구에 있어서 최고의 도구로 만들었다. 야생유전자원과 재배종들이 표현형질들과 유전체 범위에서 존재하는 단일염기치환 (Single nucleotide polymorphism; SNP)들이 얼마나 연관되어 있는가는 통계적인 SNP 빈도차이로 나타낼 수 있으며, 유전체에서 염색체범위로 SNP 빈도의 변화 경향을 추론하여 양적형질변이와 연관된 염색체 부위를 맵핑할 수 있는 것이다. 이들 맵핑위치 범위에서 연관불평형(linkage disequilibrium; LD) 상태에 있는 유전자들 중에 하나가 직접적인 양적형질변화의 유전적 원인이 된다. GWAS로 분리된 양적 형질 유전자 좌위들은 QTL인자와 같이 유전자 가위와 같은 유전공학적 도구를 이용하여 기능적, 유전학적으로 양적형질들의 피라미딩이 가능할 것이다(그림 1).
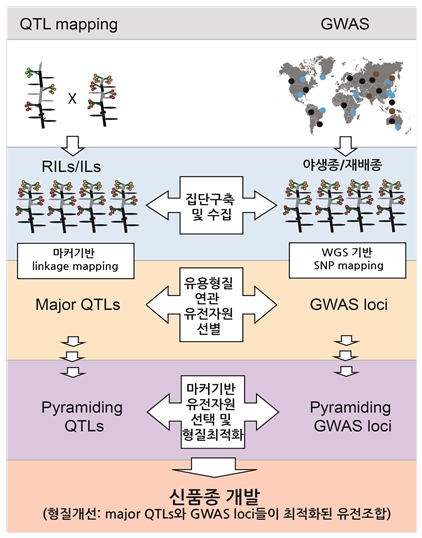
[그림 1. QTL mapping과 GWAS 연구를 통한 양적형질 유전자원 분리 및 유전자원 활용 전략을 나타낸 모식도]
유전자 가위 기술은 양적형질 유전변이를 다른 품종에 도입하거나, 양적형질을 유전공학적으로 더 강화 시키는데 매우 중요한 도구가 되고 있다. 유전자 가위 기술은 유전체에서 특정 염기서열에 결합하도록 제작된 single guide RNA(sgRNA)가 목적 DNA서열에 결합한 후, 핵산내부가수분해효소(Cas9)와 복합체를 이루어 double-strand breaks(DSB)를 유발하고, 절단된 DNA 이중가닥 위치에서 유도되는 non-homologous end-joining(NHEJ)으로 짧은 염기서열의 결실 또는 삽입돌연변이를 유발시킬 수 있다. 또는 인공적으로 유도된 염기서열 삽입을 위해 절단된 DNA 이중가닥 위치에 donor DNA를 주입하여 homology-directed repair(HDR) 또는 homologous recombination(HR)를 유도할 수 있다. 따라서 양적형질이 개선된 새로운 품종을 만들기 위해 유전자 가위 기술을 이용하여 양적형질개선 돌연변이와 양적형질개선인자 도입변이 유발이 가능하다(그림 2).
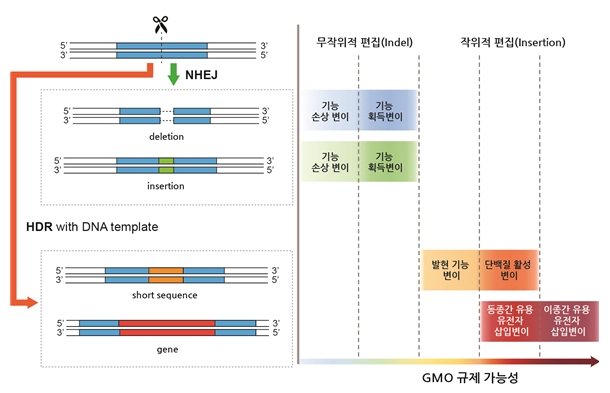
[그림 2. 유전체 편집 기술 및 작물 돌연변이체 개발과 GMO 규제에 관한 모식도]
출처 : Araki et al., 2015 Trends Plant Sci의 그림을 수정함
2. 국내외 동향
1) 야생자원 확보와 양적형질연구
야생유전자원들의 다양성은 인간에 필요한 유용형질들을 제공해주는 유전자원의 보고이며, 미래 농작물 개발에 중요한 자원이기 때문에 글로벌 규모의 유전자원의 활용과 이익공유를 위한 협약으로 ‘나고야 의정서’의 생물다양성 협약이 체결되었다. 따라서 글로벌 규모로 야생유전자원들은 수집되어 서로 공유하고 있으며, 이들 자원을 이용하여 유용형질 유전자원을 발굴하는 것은 야생유전자원 제공자와 유전자원 개발자 모두에게 이익이 돌아가도록 국제적으로 약속하고 있다.
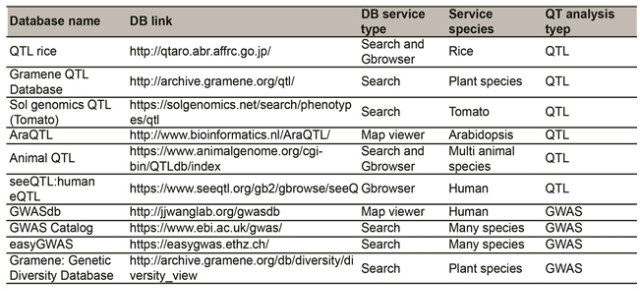
야생유전자원들을 이용하여 농작물의 양적형질연구가 활발히 진행되고 있으며 간단히 소개하자면 개화시기가 다른 야생종간 교배를 통해 QTLs 지도 기반 클로닝이 벼에서 Hd6, Ghd7, 토마토에서 SP5G 등의 유전자 기능을 밝혔다[4,5,6]. 특히 유전자 편집된 cr-sp5g변이는 효율적으로 재배기간을 조절하여 수확성을 증진시키는 유전자원을 개발했다[6]. 벼에서 야생/재배유전자원들의 GWAS분석을 통해 Hd3a, TAC 등의 양적변이들이 수확성과 연관된 잡종강세를 유발함을 밝혔다[3]. 이와 같은 양적형질 연구결과들은 앞으로 양적형질을 더 증진시키는데 중요한 유전자원이 될 것으로 생각된다. 최근에는 QTL/GWAS 연구로 분리된 양적형질인자들을 잘 정리하여 유전공학적으로 응용하고자 기초자료를 축적하고 있다. QTL map, GWAS map 등의 데이터들이 유전자 정보, 발현체 정보 등과 함께 정리하되 유전체 정보 브라우저에 함께 공유하거나, 연구자가 직접 결과를 공유하기위해 QTL/GWAS 공유사이트에 업로드 하는 방식으로 양적형질인자 연구정보가 데이터베이스화 되고 있다(표 1).
[표 1. QTL mapping 또는 GWAS 결과들을 정리해 놓은 데이터베이스 정보]
2) 개발된 양적형질유전변이의 특징
농작물의 재배종이 나타내는 수량성, 식물체형, 열매색깔, 질병저항성 개화시기 등의 양적형질들은 양적형질변이들과 연관되어 있음이 보고되고 있다. 야생종으로부터 새로운 유전자를 획득하거나 불필요한 유전자에 결실을 유발하여 명확하게 양적형질이 개선됨이 알려지고 있다(예, 벼의 Sub1A, 토마토의 SP5G)[6,7]. 특히, 발생학적으로 최적의 열매를 생산하는 형질을 획득하는데 식물조직 발생시기와 조직내부에서 발현차이를 보이는 변이, 발현강도 차이를 보이는 변이, 미세한 기능 소실 및 기능 획득 변이, 발현 유전자의 배수성이 증가된 변이들이 수량성과 같은 양적형질개선에 큰영향을 미침이 알려지고 있다(예, 벼의 OsSPL14, 토마토의 LC)[8]. 게다가, 양적형질은 유전변이들의 종합적인 효과에 기인하는데 복수의 유전인자들 간의 복합적인 유전효과가 단일 표현형에 미치는 영향에 관한 연구가 진행되고 있다(예, Hd3a, Ehd1, Hd1 등의 복합적 유전연구)[9]. 이와 같은 양적형질 변이들이 기인하는 유전정보는 유전자 가위를 이용한 해당형질 도입을 위해 매우 중요한 정보이며, 유전자 도입(Knock in), 유전자 기능 결실돌연변이 유발(Knock out), cis-regulatory elements(CREs)변이 등은 현재의 유전자 가위 기술로 양적형질 도입이 가능하다.
3) 유전자 편집연구기반 QT 개선 연구
상기에 설명된 양적형질변이는 유전자 가위로 유발된 NHEJ와 HDR 기능으로 신품종에 양적형질 개선 돌연변이와 양적형질인자 도입 변이 개발이 가능하다. 식물세포에서 양적형질인자 도입을 위해 HDR 또는 HR을 유발하기 위해 유전자 가위로 DSB를 유발함과 동시에 DNA 바이러스 replicon을 이용한 donor DNA 증폭효율을 높이는 연구가 활발히 진행되고 있으며, 밀과 토마토 등과 같은 작물에서 성공적인 HR의 결과들이 보고되고 있다[10]. 최근에는 단일염기편집 기술이 쥐의 배와 같은 동물세포에서 성공적으로 도입 되었으며 농작물과 같은 식물세포에서도 그 가능성이 점차 현실화 되고 있다. 단일염기편집기술은 nCas9(nickase Cas9)에 시토신 또는 아데닌 염기로부터 아미노기를 떼어내는 기능단백질을 결합하여 형질전환 시키면 sgRNA로 nCas9이 특정위치에 고정되고 디아미네이션과 단일 DNA가닥 절단, DNA 손상회복 등의 과정으로 시토신(C)을 티민(T)으로 아데닌(A)을 구아닌(G)으로 염기로 치환하게 하는 기술이다[11]. 이 기술은 단일염기 치환으로 유발되는 무수히 많은 양적형질변이들을 신품종에 도입할 수 있는 기술으로 기대되고 있으며, 농작물에 양적형질 도입연구가 활발히 진행되고 있다(그림 3).

[그림 3. 유전자 가위 기반 DNA 염기편집 메카니즘]
출처 : Kim, 2018 Nature plant의 그림을 수정 편집함
최근에는 유전자 가위를 이용한 양적형질변이 도입의 새로운 방법으로 특정형질을 가지는 야생종 또는 재배종과 교배로 만든 이형접합체에 유전자 가위를 이용한 DSB-induced HR repair로 형질변이도입의 새로운 가능성이 제시되었다. 이는 형질변이 도입을 위해 linkage 연관 유전자들을 치환시키는 단점을 제거 시킬 수 있는 길을 보여주고 있다[12].
4) 유전자 가위 기반 양적형질개선 기술
양적형질을 결정하는 유전자의 기능을 유전자 가위 유발변이로 조절하여 양적형질의 최적화를 이루는 방법들이 최근에 제시되고 있다. 첫째로는 양적형질의 주된 결정인자의 발현을 조절하거나 단백질 아미노산 변이로 그 활성도를 조절하여 양적형질의 최적점을 찾는 연구방법이 있다. 이 방법은 프로모터에 무작위적인 유전자 가위 유발 결실변이를 유발하여 양적형질 결정인자의 발현범위가 다양하게 나타내는 개체집단을 구축하고 양적형질의 강약을 스크리닝하여 양적형질의 최적점을 나타내는 유전자 가위 유발변이를 찾아내는 방법이다. 이방법은 토마토의 꽃대구조, 열매크기, 줄기성장 인자들의 프로모터 변이 유발과 구조의 다양성을 나타내는 연구결과로 그 가능성이 부각되었다(그림 4)[13].
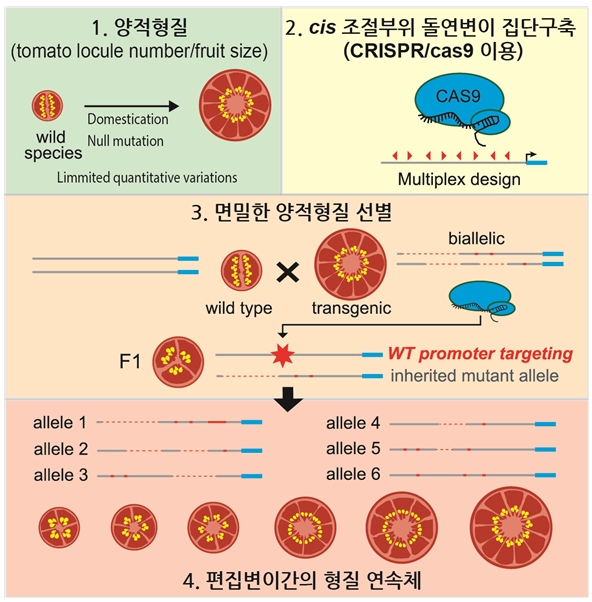
[그림 4. 양적형질결정인자 프로모터 편집을 통한 양적형질최적점 스크리닝 모델]
출처 : Rodrigues et al., 2017 Cell의 그림을 수정 편집함
둘째로는 양적형질을 결정하는 단백질 기능복합체의 유전변이조합을 이용한 기능활성 투여량 효과(dosage effect)를 분석하여 양적형질의 최적점의 변이조합을 선별하는 방법이다(그림 5). 양적형질을 결정하는 중요인자가 그 기능을 수행하기 위해서는 복수의 단백질이 서로 결합하여 기능적 복합체를 이루어서 최상의 기능을 수행하게 된다. 기능적 복합체를 구성하는 단백질에 유전자 편집 돌연변이를 유발하고 각 복합체의 돌연변이에 동형접합 또는 이형접합의 변이들의 유전자형 조합을 양적형질과 연관하여 표현형을 분석하면 특정유전자형 조합에서 최적의 양적형질을 나타냄을 확인할 수 있음을 토마토 개화인자, 꽃대발생인자 연구와 야자 열매유 잡종강세 연구에서 밝혔다[14, 15].
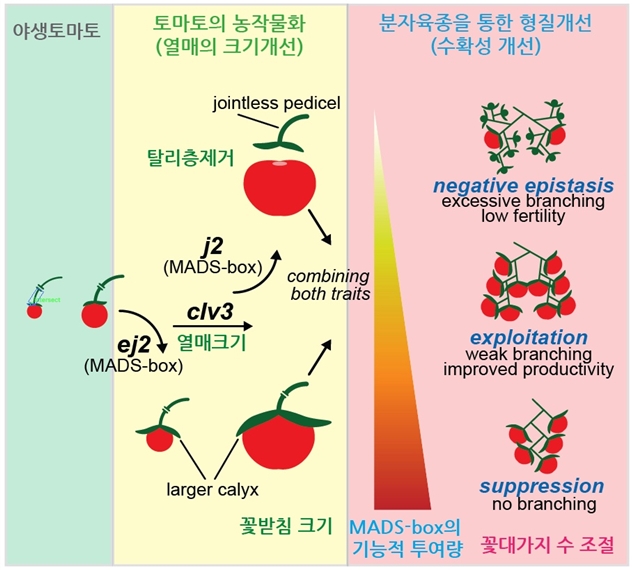
[그림 5. 토마토 꽃대가지 수 결정인자인 MADS-box 유전자 복합체의 기능적 투여량 효과 분석으로
최적의 양적효과 스크리닝 모델]
출처 : Sebastian et al., 2017 Cell의 그림을 수정 편집함
...................(계속)
* 로그인 하셔야 자세한 정보를 모두 보실 수 있습니다.
-
이전글
- 식품 유해성분 동시검출 센서 기술 동향(Simultaneous detecting sensor)
-
다음글
- 오가노이드 기반 생체모사기술 (Organoids-based biomimetics)
지식


 유전체 편집 기반 양적 형질 조절기술.pdf
유전체 편집 기반 양적 형질 조절기술.pdf
 토마토 꽃대가지 수 결정인자인 MADS-box 유전자 복합체의 기능적 투여량 효과 분석으로 최적의 양적효과 스크리닝 모델
토마토 꽃대가지 수 결정인자인 MADS-box 유전자 복합체의 기능적 투여량 효과 분석으로 최적의 양적효과 스크리닝 모델