BioINpro
(BioIN + Professional) : 전문가의 시각에서 집필한 보고서 제공[KRIBB 워킹그룹] de novo 단백질 디자인 기술의 발전 현황 II
- 등록일2023-11-29
- 조회수2711
- 분류생명 > 생명과학, 생명 > 생물공학, 플랫폼바이오 > 바이오융합기술
-
저자/소속
안우찬 외 2명/한국생명공학연구원 중대질환진단융합연구단
-
발간일
2023-11-29
-
키워드
#de novo 단백질 디자인 기술#인공지능 기반의 단백질 설계#단백질 기능에 따른 디자인 전략 개발#단백질 디자인 기술 개발
- 첨부파일
-
차트+
?
차트+ 도움말
de novo 단백질 디자인 기술의 발전 현황 II
2023년도 KRIBB 워킹그룹이슈페이퍼 제 9호
◈ 목차
1. 개요
2. 주요 동향 및 이슈
2.1 단백질 디자인 기술 개발 동향
2.2 인공지능 기반의 단백질 설계 전략
2.3 단백질 기능에 따른 디자인 전략 개발 동향
2.4 국내 연구진 디자인 기술 개발 동향
2.5 인공단백질 디자인 기반 산업계와 학회 동향
3. 결론 및 시사점
◈본문
1. 개요
□ 단백질 구조 예측과 단백질 설계 분야 흐름의 변화, 인공지능
○에너지 계산을 기반으로 하는 전통적인 방식의 단백질 구조 예측과 단백질 설계
╶︎원래 구조 예측과 단백질 설계는 기본 물리 화학적 에너지 계산이나, 유사한 서열을 가진 단백질의 구조를 기반으로 이루어짐
╶︎에너지 계산 방식으로 가장 대표적인 것은 로제타로, 반복적인 에너지 계산을 통해서 가장 안정적인 구조와 서열을 찾아내는 방식임
○알파폴드의 등장과 함께 나타난 지각변동
╶︎2020년 알파폴드가 처음 등장하고 기존의 단백질 구조 예측 방법보다 월등한 성능을 보임
╶︎’21년 알파폴드 코드 공개 및 로제타폴드(RoseTTAFold)가 개발되면서 바야흐로 인공지능 기반의 단백질 구조 예측의 시대가 열리고 과학계를 넘어서는 관심을 받음
╶︎인공지능 기반의 구조 예측 방법의 성공 뒤를 이어 인공지능 기반의 단백질 설계 방법들이 개발, 공개되기 시작
□ 단백질 디자인과 단백질 구조 예측
○단백질 구조 예측 연구는 1차원의 아미노산 서열 정보를 이용해서 단백질의 3차 구조를 예측하는 연구 분야임
○단백질 디자인은 단백질 구조 예측의 역순으로, 특정한 3차원 구조를 형성하기 위해서 어떤 아미노산 서열이 필요한지 추적하는 연구임
○단백질 구조 예측과 단백질 디자인의 관계는 상호보완적으로, 단백질 구조 예측 기술의 성장은 단백질 디자인 기술의 성장과 비례함
|
출처 : 워싱턴 대학교 단백질 디자인 연구소 (UW Institute for Protein Design) |
[그림 1] 단백질 디자인과 단백질 구조 예측의 관계 |
2. 주요 동향 및 이슈
2.1 단백질 디자인 기술 개발 동향
□ 알고리즘 기반의 디자인 기술의 대두
○다양한 인공지능 모델을 기반으로 한 단백질 디자인 기술이 앞다투어 나오는 상황
╶︎대표적으로 ProteinMPNN, RFdesign, RFdiffusion 등이 있음 [표 1]
○인공지능 기반 디자인 기술들은 사용법이 비교적 간단하여 진입장벽을 크게 낮추었음
╶︎깃허브(Githhub) 등을 통해서 쉽게 설치가 가능하며, 구글 코랩(Colab)과 같은 클라우드 서비스를 이용하면 고성능의 그래픽 카드를 보유하지 않아도 사용 가능해짐
╶︎에너지 기반 단백질 디자인 프로그램 중 가장 대표적인 로제타(Rosetta Suite)과 같은 스크립트(Script) 기반의 프로그램보다 접근성이 매우 높아짐
[표 1] 주요 단백질 디자인 인공지능 모델
모델 이름 | 접근법 | 주요 기능 및 특징 |
ProteinMPNN | 그래프 인공 신경망 메시지 전달 인공 신경망 |
|
MaSIF | 기하학 기반 귀납적 편향 합성곱 신경망 |
|
Hallucination | 구조적, 구조 안정성 오차 추적 |
|
DiffAb | 확산 모델 |
|
RFdiffusion | 확산 모델 |
|
Chroma | 확산 모델 |
|
ProGen | 언어 모델 |
|
출처: Cell Systems (2023)
□ 그래프 인공 신경망 기반 디자인 기술
○그래프 인공 신경망은 노드(원자 혹은 아미노산)와 노드 사이의 관계인 엣지(원자 간 결합 혹은 공간적 상호작용)를 부여하여 단백질의 구조를 그래프 형태의 데이터로 적용하여 분석
╶︎그래프 인공 신경망 종류중 하나인 MPNN은 메시지 전달 인공 신경망(Message Passing Neural Network). 이 인공 신경망의 핵심은 각 노드(아미노산)들이 주변의 노드(아미노산)의 영향을 계산하여 자신을 수정하는 ‘메세지 전달’이고, 하나의 아미노산의 변화가 자신 서열의 앞, 뒤, 공간적으로 주변의 잔기들의 영향을 주고 받기 때문에 구조적 데이터 연구에 적합
╶︎가장 대표적인 기술로 David Baker 연구팀이 개발한 ProteinMPNN이 있음 (22’ Science)
|
출처 : Science (2022) |
[그림 2] 그래프 인공 신경망 기반 단백질 디자인 기술 ProteinMPNN |
□ 확산 모델 기반 디자인 기술 RoseTTAFold diffusion(RFdiffusion)
○가장 대표적인 확산 모델 기반 디자인 기술인 RFdiffusion은 2022년 말 BioRxiv에 공개되면서 이미 큰 주목을 받고, 2023년 3월 코드가 공개되고 이어서 7월 네이처지에 출판(23‘ Nature)
╶︎인공지능 확산 모델은 입력에서 노이즈를 제거하도록 훈련된 신경망으로, 임의의 픽셀 혼란을 주면 이를 인식 가능한 이미지로 바꾸어 줌
╶︎RFdiffusion은 무작위적인 입력값에서부터, 노이즈를 조금씩 제거하며 예측을 진행하는 과정을 반복하여, 설계 가능한 단백질 골격을 만들어감. 이 단백질 골격에 ProteinMPNN을 이용하여 골격 단백질이 유지 가능한 서열을 생성하여 신규 단백질을 설계
╶︎RFdiffusion의 단백질 디자인 성능은 현재까지 보고된 기술 중 가장 높은 디자인 성공률을 보임
※︎RFdiffusion 논문에서 보고한 결합 단백질 성공률을 10% 이상으로 기존 방법론이 1% 미만의 성공률에서 큰 도약을 함
|
출처 : Nature (2023) |
[그림 3] RFdiffusion이 작동 방식과 이를 이용해 디자인된 예시 |
○아미노산 뿐 아니라 핵산, 저분자, 금속 등을 고려할 수 있는 구조 예측 모델 RoseTTAFold all-atom(RFAA)와 생성 모델 RFdiffusion All-Atom(RFdiffusionAA)의 등장
╶︎알파폴드나 로제타폴드의 한계점 중 하나는 단백질 서열만 예측 가능하고, 저분자 등 단백질이 아닌 구성요소는 추가로 도킹(docking)과 같은 독립적인 과정을 진행해야 한다는 것임
╶︎복합체 구조를 포함하는 학습을 통해 개발된 RFAA는 알파폴드2와 유사한 수준의 정확성을 보임
╶︎RFAA를 기반으로 한 RFdiffusionAA는 치료용 의약품, 효소 보조인자(cofactor)와 광합성 관련 저분자를 결합할 수 있는 신규 단백질을 설계하고 실험적으로 검증하여, RFdiffusionAA의 높은 활용도와 정확도를 제시함
○제너레이트 바이오메디슨 사에서 개발한 디퓨젼 모델인 크로마가 2023년 9월 네이처에 공개되고, 이어서 11월 모델을 공개(23’ Nature)
╶︎RFdiffusion과 유사하게 확산모델을 사용하여 구조를 생성하는 모델
╶︎conditioner라는 이름으로 다양한 제한을 두어 대칭구조, 특정 모티프, 특정 모티브 유지 등의 다양한 기능을 탑재
╶︎눈에 띄는 점으로 자연어를 이해하여 구조를 생성하는 기능이 있음. 이는 최근 이미지 생성형 인공지능이 자연어를 이해하고 그에 맞는 이미지를 생성해주는 것과 같은 기능임
╶︎제너레이트 바이오메디슨은 암젠과 연구 제휴를 통해서, 5가지 임상 표적에 대한 단백질 치료제 개발 제휴를 맺고, 결과에 따라 최대 19억 달러를 지급하기로 한 바 있음
|
출처 : Nature (2023) |
[그림 4] 제너레이드 바이오메디슨 사의 Chroma로 생성된 다양한 단백질 구조 |
□ 로제타폴드(Rosettafold) 기반 디자인 프로그램 Rosettafold design(RFdesign)
○로제타폴드를 개발한 David Baker그룹에서는 로제타폴드 기반의 단백질 디자인 기술인 RFdesign을 개발
╶︎RFdesign은 결합이나 촉매 활성에 필요한 기능성 모티프를 입력(input)으로 하여, 원하는 활성과 특성을 가진 단백질을 설계할 수 있음
○RFdesign은 기능성 단백질 설계를 위해서 Hallucination과 inpaint라는 두 가지 전략을 사용함
╶︎Hallucination은 구조 예측과 아미노산 서열 변이를 수천 번 반복하여, 높은 신뢰도를 갖는 구조와 서열을 도출함
╶︎inpaint는 구조예측 인공지능 모델을 구조와 서열이 일부 소실된 데이터를 복구하도록 추가적으로 학습시켜, 모티프 사이를 연결할 수 있음
|
출처 : Science (2022) |
[그림 5] RFdesign 기술(Hallucination/inpaint)의 개요 |
□ 인공지능 기반 에너지항 계산을 통한 단백질 디자인 SCUBA
○Haiyan Liu 그룹에서 개발한 SCUBA(Side chain-Unknown Backbone Arrangement) 모델은 신경망 형태의 에너지항(Energy term)을 사용하여 주형(template)이 필요 없는 de novo 단백질을 설계할 수 있음(22‘ Nature)
╶︎SCUBA는 기존의 단백질 디자인 기술이 이미 알려지거나 만들어진 골격 단백질에 의존해야 하는 제한점을 극복하고자 시도됨
╶︎SCUBA는 아미노산의 곁사슬을 배제하고, 골격 중심으로 학습된 모델로, 신경망 형태로 에너지항을 계산하여 새로운 형태의 골격 단백질을 생성함. 이후 생성된 골격 유지를 위한 아미노산 서열을 ABACUS2(20’ Bioinformatics)라는 서열 생성 모델을 통해 도출함
╶︎해당 모델을 이용하여 디자인한 새로운 구조의 단백질 9개 결정 구조로 디자인 성능을 검증
□ 언어모델 기반 단백질 디자인 기술 ProGen
○단백질의 서열은 20개의 아미노산이 일정한 규칙에 따라서 순서대로 나열되어 있는 것이 언어와 유사성이 있고, 이러한 배경으로 다양한 언어모델을 통해서 단백질 서열을 학습시킴
╶︎언어모델을 방대한 양의 텍스트를 통해서 숨어있는 규칙을 인공지능이 비지도 학습으로 학습하게 되는 것인데, 염기 서열 분석 기술과 프로테오믹스의 발달로 보유하게 된 방대한 양의 단백질 서열 데이터를 기반으로 언어모델을 학습시켜 단백질 언어모델(Protein language model, PLN)들을 개발함
○Salesforce Research사에서 개발한 ProGen은 생성형 언어모델 기반의 단백질 디자인 기술로, 원하는 단백질의 특성을 입력하면 해당 특성을 가진 새로운 단백질 서열을 생성함(23’ Nature)
╶︎2.8억 개의 단백질 서열을 1만 9천 개의 단백질 패밀리(Protein family)를 기반으로 학습된 이 언어모델은 원하는 단백질 패밀리나 특성을 입력하면 해당 패밀리에 속할 수 있는 새로운 아미노산 서열을 출력함
|
출처 : Nature (2023) |
[그림 6] 언어 생성 모델 기반의 단백질 디자인 프로그램 Progen |
2.2 인공지능 기반의 단백질 설계 전략
□ 단백질의 기능-구조-서열의 관계와 디자인 전략
○단백질 설계는 결국 특정한 기능을 하기 위한 단백질을 만들고자 하는 것이고, 최종적 산물은 단백질의 서열. 즉, 단백질의 기능에서 시작하여 최종적인 서열을 획득하는 것을 목표로 함
○단백질의 기능, 구조와 서열 세 가지 요소의 관계로 인하여, 단백질 설계 전략은 3가지 중 한 가지를 취하게 됨
╶︎단백질의 기능을 위한 구조 설계
╶︎단백질의 구조를 위한 서열 예측
╶︎단백질의 기능을 위한 서열 예측
|
출처 : 집필진 작성 |
[그림 7] 단백질 기능-구조-서열의 관계와 단백질 디자인 전략 유형 |
□ 단백질 기능을 위한 구조 설계
○단백질 기능을 위한 구조를 설계할 경우 표적 결합 단백질, 올리고머 단백질, 효소 단백질 등 목표로 하는 단백질의 기능에 따라서 적합한 골격을 도출해야 함
╶︎표적 결합이나 효소 단백질의 경우, 결합 모티프나 활성 모티프를 유지할 수 있는 적절한 골격을 생성해야 하고, 대칭 올리고머 단백질의 경우 반복 순환적인 골격 구조를 생성할 수 있어야 함. 동시에, 단백질 접힘 원리도 고려해야 함
○대표적인 디자인 방법은 할루시네이션과 확산 모델 기반의 기술
╶︎할루시네이션이 경우, 무작위적인 돌연변이와 구조 예측을 여러 차례 반복하여 구조 예측 알고리즘에 의해서 성립 가능한 구조를 찾아 나가는 방식임. 자연계에 존재할 것 같지만, 다시 말하면 단백질의 접힘 원리를 따르지만, 존재하지 않는 새로운 구조와 서열을 동시에 생성함. 본래 기능은 구조와 서열을 동시에 생성하지만, 많은 경우 서열 생성을 다시 진행
╶︎RFdiffusion은 단백질의 모든 원자를 사용하지 않고, 골격에 사용되는 N, Ca, C만을 사용하여 골격을 형성함. 대칭 올리고머 디자인, 결합체 디자인, 모티프나 효소 활성 부위 고정을 위한 골격 설계 등이 가능하며, 골격 생성 이후에 서열 디자인을 필요로 함
□ 단백질 구조를 위한 서열 예측
○단백질 구조를 위한 서열예측은 원하는 구조를 유지하기 위한 아미노산 서열을 생성하는 전략임
○이 부분에서는 현재로는 ProteinMPNN이 가장 대표적이고 압도적인 성과를 내고 있음
╶︎ProteinMPNN을 사용하여 디자인된 단백질의 성공 사례가 이미 다수 보고되었고, 특히 할루시네이션과 확산 모델 기반 기술과 접목되면서 활용도가 높아지고 있음
╶︎따라서 실험적으로도 가장 많이 검증된 디자인 전략 중 하나임
╶︎상대적으로 간단한 사용법과 빠른 생성 속도 또한 큰 강점임
□ 단백질 기능을 위한 서열 예측
○단백질 기능을 위한 서열예측은 특정한 기능을 가지는 단백질을 구조를 생성하는 단계를 거치지 않고 서열을 생성하는 방식임
○주로 특정한 기능을 하거나 할 것으로 예상되는 단백질 서열을 학습하고, 그와 유사한 서열을 생성하게 하는 전략을 취함
╶︎루시퍼레이즈 유사 단백질 서열들로 학습된 모델이 루시퍼레이즈 유사 단백질 서열을 생성하거나, 특정 항원에 결합 수 있는 항체의 서열을 기반으로 학습하여 해당 항원에 결합할 수 있는 새로운 항체 서열을 생성하는 사례
╶︎최근에는 기능별로 새로운 학습을 하는 것이 아닌, 하나의 모델이 다양한 기능의 단백질 서열을 생성하는 ProGen과 같은 기술이 개발되고 있음
2.3 단백질 기능에 따른 디자인 전략 개발 동향
□ 효소 단백질
○효소는 특정 생화학적 반응을 촉진하는 단백질로 활성 잔기, 결합 부위 등이 구조적으로 필요한 위치에 정확하게 존재해야 하므로, 인공적으로 설계하는 데 제약이 많음
○De novo 루시퍼레이즈(luciferase) 효소 단백질 개발(23’ Nature)
╶︎인공단백질 디자인의 도전적인 과제 중 하나였던, de novo 효소 단백질 디자인으로 이 성과는 단백질 디자인 분야의 큰 도약을 의미함
╶︎RFdesign의 할루시네이션(hallusination) 기술을 활용하여 원하는 결합부위를 가진 스캐폴드단백질을 생성하고, 기존에 알려진 활성 잔기들을 이식(grafting)하는 방법으로 설계하였음
╶︎이 de novo 효소 단백질은 낮은 크기와 높은 안정성을 가지며, 자연형보다 높은 기질 특이성을 가짐
|
출처 : Nature(2023) |
[그림 8] De novo 루시퍼레이즈 설계 전략 |
○저분자 리간드 결합을 위한 배럴형 단백질 스캐폴드 생성 기술 개발 (23’ Nature)
╶︎미국 워싱턴대학교 David Baker 실험실의 Linna An 외 연구진은 TIM 배럴, 베타 배럴과 같은 유사순환(pseudocyclic) 구조 스캐폴드를 생성할 수 있는 딥러닝 기반 기술을 개발함
╶︎할루시네이션 모델을 기반으로 하는 이 기술을 2~7개의 반복 단위를 사용하여 최대 156개 아미노산 길이의 배럴 형태의 스캐폴드를 생성함
╶︎배럴 형태의 스캐폴드의 중앙에 나타나는 공간은 다양한 저분자를 품을 수 있는 크기이기 때문에, 이 스캐폴드 생성 기술은 추후에 저분자 리간드 결합 단백질이나 효소 단백질 디자인 기술 발전 활용될 수 있음
○RFdiffusion All-atom 기반 저분자 결합 단백질 디자인(23’ BioRxiv)
╶︎최고 보고된 RFdiffusion All-atom은 단백질 뿐 아니라 이와 상호작용하는 다양한 생화학적 물질들까지 고려하는 단백질 구조 생성 모델임
╶︎이 기술은 본격적으로 다양한 저분자, 핵산 등과 결합 및 효소 활성을 보일 수 있는 성능을 가짐
╶︎대표적으로 실험적 검증을 진행한 성과는 헴(Heme)결합 단백질 38종, 광합성에 연관된 색소인 빌린(Bilin) 결합 단백질을 3종, 심장 질환 관련 저분자 의약품(Digoxigenin) 결합 단백질 종을 디자인하고 결합을 검증하였음
|
출처 : BioRxiv(2023) |
[그림 9] RFdiffusion All-atom 기반 저분자 결합 단백질 디자인 |
□ 센서 단백질 (Biosensor protein)
○저분자 결합 단백질 설계 방법론 제시(23’ BioRxiv)
╶︎미국 워싱턴대학교 David Baker 실험실의 이규리 박사 외 연구진은 저분자 결합 단백질 및 센서를 설계하는 방법론을 제시하고, 이를 활용한 6가지 이상의 저분자 타깃에 대한 결합 단백질로 검증함
╶︎이 저분자 결합 단백질을 기반으로 저분자를 인식할 수 있는 단백질A와, 저분자와 단백질A의 복합체를 인식할 수 있는 단백질 B를 설계하여, 저분자 센서 플랫폼을 제시함. 본 논문에서는 예시로 코르티솔(cortisol)을 타깃으로 한 바이오센서를 개발함
|
출처 : BioRxiv(2023) |
[그림 10] 저분자 센서 단백질 설계 모식도 |
□ 플랫폼 기술 단백질 (Platform technology protein)
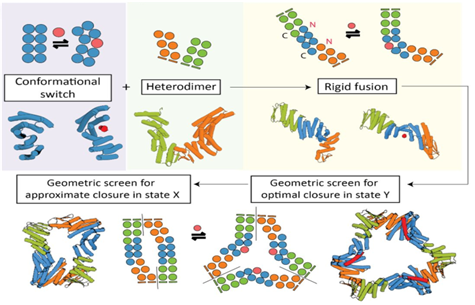
○신호에 따라서 2가지 구조를 형성할 수 있는 경첩(hinge) 단백질과 이를 활용한 다량체를 개발(23‘Science, 23’ BioRxiv)
╶︎Florian Preatorius 외 연구진(미국 워싱턴 대학교 David Baker 교수 실험실)은 인공적인 신호 단백질의 유무에 따라서 2가지 안정적인 구조를 형성할 수 있는 인공 경첩 단백질을 개발함
╶︎이 경첩 단백질은 8개의 나선 다발로 구성되었는데, switch 단백질과 결합하여 9개의 나선 다발을 형성하여, 직선형에서 꺾인형으로 구조적 변화를 겪음
╶︎개발된 경첩 단백질 기술을 기반으로 상황에 따라서 다양한 다량체로 변화할 수 있는 조립체(assembly)들을 개발함
|
출처 : BioRxiv(2023) |
[그림 11] 정해진 자극에 따라서 올리고머 상태 변화가 가능한 단백질 설계 |
○나노 입자를 고정할 수 인공 결정화 단백질 개발(23’ Nature Materials)
╶︎최근 Zhe Li 외 연구진(미국 워싱턴 대학교 David Baker 교수 실험실)은 인공적인 결정화를 유도할 수 있는 단백질은 디자인함. 95도 이상의 고온 등 가혹조건에서도 결정상태를 유지
╶︎수화(hydration)와 건조(dry)를 통해 수축 팽창이 가능하며, 이 과정으로 금입자(Gold nanoparticle, AuNP)를 초격자(superlattice)화 할 수 있음
|
출처 : Nature Materials(2023) |
[그림 12] 결정화 단백질 설계 전력과 금입자 초격자화 |
○서열특이적 DNA 결합 단백질 개발(23‘ Biorxiv)
╶︎5~6개의 염기서열을 인식해서 이중가닥 DNA의 굴곡에 결합할 수 있는 de novo 단백질이 RIFdock, MotifGrafting과 ProteinMPNN 기술을 활용하여 개발됨
╶︎30-100 nM 수준의 결합력을 가지며, 이 신규 단백질을 기능성 단백질들과 재조합하여 만든 전사조절 단백질은 대장균과 동물세포 내에서 작동하는 것을 확인함
□ 펩타이드와 펩타이드 결합 단백질 (Peptide & peptide binding protein)
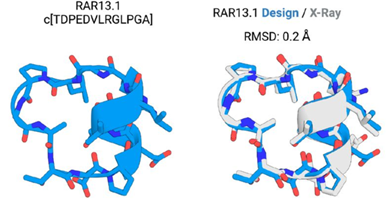
○순환 펩타이드 구조 예측 및 디자인 기술 개발(23‘ Biorxiv)
╶︎하버드대학교 Sergey Ovchinnikov 교수와 워싱턴대학교 Gaurav Bhardwaj 교수 연구팀의 협업으로 개발
╶︎순환 펩타이드는 펩타이드의 N-말단과 C-말단 간을 공유결합 등으로 연결하여 안정성을 높이는 전략
╶︎알파폴드 모델을 기반으로 하여 순환 펩타이드의 구조 예측에 특화된 구조 예측 모델과, 특정 순환 펩타이드의 골격을 형성하기 위한 아미노산 서열을 최적화하여 de novo 순환 펩타이드 디자인 방법론을 제시함
╶︎7~13개 아미노산 길이의 다양한 구조의 순환 펩타이드 골격에 대해서 서열 최적화를 한 경우, 높은 구조 예측 신뢰도를 보임. 7종의 경우 결정화를 통해 규명과 구조와 모델이 RMSD 1.0 Å이하의 높은 구조적 유사성을 보임
|
출처 : BioRxiv(2023) |
[그림 13] 순환 펩타이드 구조 디자인 |
○만성 통층 치료용 펩타이드 개발(23‘ eLife)
╶︎Valdimir Yarov-Yarovoy 교수 (미국 University of California, Davis) 연구팀은 만성 통증 질환의 핵심 표적인 Nav1.7 채널선택적 펩타이드를 개발하는 데 성공
╶︎타란툴라 거미 독의 주요 독소인 ProTx-II를 주형으로 시작하여, 로제타를 이용한 디자인을 통하여 NaV1.7에 대한 결합력과 선택성을 극적으로 높임
╶︎마우스와 인간 감각 뉴런 세포에서 전류를 억제하였고, 마우스 모델에서 활성을 확인
○아밀로이드 형성 펩타이드 저해제 개발(23‘ Biorxiv)
╶︎아밀로이드는 여러 작은 단백질들이 반복적으로 결합하는 섬유화로 인해 발생하는 단백질 응집체로 대표적으로 알츠하이머병을 유발하는 아밀로이드 베타(amyloid-beta)나 타우(tau) 단백질 펩타이드들 있음
╶︎이 펩타이드들이 섬유화되면서 베타-병풍 구조(beta-sheet)를 형성하는 점을 착안하여, 네덜란드 휘브레흐트 연구소의 Danny Sahtoe 교수는 타우 단백질 펩타이드를 포함하는 4종의 아밀로이드 펩타이드를 특이적으로 결합할 수 있는 단백질을 설계
╶︎이 단백질들은 자신들 골격의 베타-병풍 구조 사이에 이 아밀로이트 펩타이드를 삽입할 수 있도록 설계됨
|
출처 : BioRxiv(2023) |
[그림 14] 아밀로이드 펩타이드 결합 단백질 디자인 전략 |
2.4 국내 연구진 디자인 기술 개발 동향
□ 효소 디자인 기술로 감미료 생산 효소 개발
○한국생명공학연구원 우의전 박사 연구팀은 로제타와 PSSM 알고리즘을 활용한 효소 디자인 기술로 설탕 대체 감미물질인 레바우디오사이드를 생산할 수 있는 UGT 효소의 안정성과 활성을 증대시킴 (23’ Comm. Chem)
╶︎중요 설탕 대체제인 레바우디오사이드 생산 효소 활성 개선을 위하여 효소의 활성 부위가 아닌 주변 잔기에 돌연변이를 가해 간접적으로 효소의 활성을 증대시키고 안정성을 향상시키는 전략을 사용
╶︎UGT효소의 당전이 활성이 2.6배 증대되고, 내열성 지표인 Tm을 9도 상승한 변이체 1종과, 당전이 활성이 1.9배 증대되고 55도씨에서도 최대 활성을 유지하는 변이체 1종을 개발
□ SARS-CoV-2 돌연변이 회피 항체를 단백질 계산을 통해 선제 개발
○KAIST 오병하 교수 연구팀은 팬더믹 초기에 개발된 SARS-CoV-2 표적 단일클론항체들이 변이 바이러스 발생으로 인하여 결합력이 저하되거나 없어지는 점을 고려하여, 보고된 모든 코로나 변이에 결합 가능한 항체를 개발 (22’ mAbs)
╶︎컴퓨터를 이용한 계산과 실험적 결합력 증대 방법, 구조적 분석을 통하여 피코몰(pM)수준의 강력한 결합력과 중화 활성을 가진 신규 항체 개발
□ 단백질 구조 예측 모델 데이터베이스의 구조 기반 클러스터링 기술 개발
○서울대학교 Martin Steinegger 교수 연구팀은 거대 구조 모델 데이터베이스에서 유사 구조를 빠른 속도로 군집화할 수 있는 FoldSeek 기술을 개발(23’ Nat. Biotechnol.)
╶︎구조적으로 근접한 아미노산 간의 관계를 일종의 알파벳 형식으로 치환하는 방법을 통해 구조적 유사성을 빠르게 계산할 수 있는 방법론으로 기존의 방식인 TM-align보다 4천 배, Dali보다 2만 배 빠른 성능
○Martin Steinegger 교수 연구팀은 또한, 이 FoldSeek 기술로 3억 개에 달하는 알파폴드 데이터베이스를 분류, 단백질 구조와 서열, 기능 간의 새로운 통찰을 제시(23‘ Nature)
╶︎전통적인 단백질 분류 방법인 아미노산 서열 상동성 기반의 방법에서는 분류할 수 없던 단백질들이 구조적 기준으로 분류가 가능해졌고, 기능을 유추할 수 있게 됨
╶︎이는 서열-구조-기능 간의 관계도를 보완하여 거대 언어모델 기반 단백질 디자인 등 다양한 구조 예측과 단백질 설계 분야에 큰 영향을 기대하게 함
2.5 인공단백질 디자인 기반 산업계와 학회 동향
□ 국외 단백질 디자인 기반의 스타트업
○단백질 설계 기술 기반의 스타트업의 경우 크게 개발된 신약 파이프라인을 기반으로 하거나, 디자인 기술 자체를 기반으로 하는 2가지 전략을 취하는 것이 대표적이나, 최근에는 특정 분야에 특화된 기술을 보유한 기업들이 주목받고 있음
╶︎세포치료제 관련 기술의 Outpace Bio, 항체 개발 중심의 absci, 효모 기반 스크리닝 기술 중심의 A-alpha bio 등이 각 분야에서 특화된 기술을 보유하고 있으며, 독자적인 개발과 파트너십을 통한 협력을 동시에 진행함
○A-alpha bio (https://www.aalphabio.com/)
╶︎시애틀에 본사를 둔 A-alpha Bio는 효모를 이용하여 단백질 간 상호작용을 대량으로 스크리닝하고, 이를 기반으로 머신러닝 플랫폼을 기술을 보유한 회사임
╶︎본 회사는 자신들이 기술을 AlphaSeq과 AlphaBind라는 이름의 두 가지 기술로 명명함
╶︎표적 단백질에 상호작용 할 수 있는 후보를 스크리닝하는 AlphaSeq과 수집된 데이터와 기존의 데이터베이스를 기반으로 더 강력하거나 원하는 결합 성질을 가진 단백질을 디자인하는 AlphaBind 기술을 통하여 결합력과 선택성을 조절할 수 있다고 함
╶︎워싱턴 대학교의 단백질 디자인 연구소의 스핀오프로 시작된 A-alpha Bio는 2021년 시리즈A에서 2,000만 달러를 유치한 후 빌 게이츠 재단 등의 지속적인 투자를 통해 2023년 7월 기준으로 총 자금 5억 1,000만 달러를 확보한 것으로 알려짐
|
출처 : A-Aplha Bio사 홈페이지 (https://www.aalphabio.com/) |
[그림 15] A-Alpha Bio사의 플랫폼 기술 AlphaSeq |
○Outpace Bio (https://www.outpacebio.com/)
╶︎Lyell Immunopharma사의 스핀오프인 Outpace Bio는 단백질 디자인 기술을 통해 효과적이고 안전한 세포치료법을 개발하는 데 초점을 맞추고 있으며, 특히 다양한 고형암 치료를 목적으로 함
╶︎공개된 Outpcae Bio의 파이프라인 중 하나인 OPB-101은 췌장암, 난소암 등의 암에서 과발현되는 바이오마커인 메조틸린(mesothelin, MSLN)을 표적으로 하는 세포치료법을 단백질 디자인 기술로 효율과 안전성을 높였다고 보고하고 있음
╶︎2021년 시리즈A를 통해 3,000만 달러를 투자 유치에 성공함
출처 : Outpace Bio사 홈페이지 (https://www.outpacebio.com) |
[그림 16] Outpace Bio사의 세포치료제 관련 기술들 |
○Evozyne (https://www.evozyne.com/)
╶︎미국 시카고 기반의 단백질 디자인 회사로 진화 기반(evolution-based) 단백질 디자인 기술을 보유
╶︎유전자 편집 단백질 기술 개발 연구를 진행 중이며, 이 기술을 기반으로 대형 제약사인 타케다(Takeda Pharmaceutical)와 전략적 협력과 4,000만 달러 규모의 계약을 맺음
╶︎2023년 시리즈B를 통해서 8,100만 달러 규모의 투자 유치. 엔비디아가 참여한 이번 투자에서는 Evozyme이 기존의 치료용 단백질 디자인뿐만 아니라, 면역반응 조절을 위한 유전자 편집 단백질과 효소, 탄소 포집 분야로 영역을 넓혀갈 것으로 제시함
○Absci (https://www.absci.com/)
╶︎2023년 새로운 항체 생성 인공지능 모델을 발표한 Absci는 표적 에피톱(epitope)의 구조만을 입력값(input)으로 하여 새로운 CDR 서열을 생성하는 기술을 개발. HER2 등 세 가지 표적에 대한 항체를 생성, 검증하여 보고함
╶︎HER2 표적에 대해서 추가적인 최적화 과정 없이, 모델을 통해서 생성된 서열 중 10.6%가 타깃에 대한 결합력을 나타낸 것으로 보고. 기존의 치료제로 사용 중인 항체보다 강력한 결합력을 보이는 새로운 항체 3종을 확인하였으며, 이들은 기존의 항체와 낮은 서열상동성을 보임
□ 국내 단백질 디자인 기반의 스타트업
○테라자인(Therazyne)
╶︎단백질 디자인 기술을 토대로 차세대 면역치료제용 단일클론항체 개발
╶︎창업자인 KAIST 오병하 교수는 워싱턴대학교 베이커 교수와 협업을 통해서 코로나 센서 단백질을 디자인하여 보고한 바 있음(21’ Nature)
○갤럭스(Galux)
╶︎물리화학과 인공지능을 접목한 신약 설계 소프트웨어 개발. 단백질 신약과 저분자 화합물 신약 발굴
╶︎서울대학교 석차옥 교수가 설립한 회사로, 기존 개발해 오던 갤럭시(Galuxy) 프로그램을 기반으로 함. 석차옥 교수는 구조예측대회인 CASP의 과학자문위원 중 한 명임
╶︎카카오브레인으로 50억 원 투자금 유치 및 공동연구 진행
○에이인비(AinB)
╶︎인공지능활용 항체 설계 스타트업
╶︎항체의약품 개발 전문가 박은영 대표(전 삼성종합기술원, ABL바이오)와 인공지능 전문가 서승우 CTO(전 삼성종합기술원, 스탠다임)가 설립하여, 실험적으로 검증된 인공지능 모델을 개발
╶︎유한양행과 메티톡스가 벤처투자에 참여하였고, 중소벤처기업부에서 총괄하는 2023년 민간투자 주도형 기술창업지원 프로그램(TIPS)에 선정됨
○아론티어(Arontier)
╶︎인공지능 기반의 신약 개발 스타트업
╶︎최근 항체 신약 스타트업 넥스아이와 업무 협약을 맺고, 인공지능 기반 면역원성 회피 전문가인 전남대 최윤주 교수를 자문위원으로 영입하는 등 인공지능 기반의 바이오의약품 개발에 투자
╶︎그래디언트(오가노이드), 아이진(mRNA), 퓨처메디신(뉴클레오사이드 신약) 등 다양한 분야 기업과 협약
□ 국내외 단백질 디자인 관련 학회
○로제타공동체(Rosettacommons, https://www.rosettacommons.org/)
╶︎오픈 소스 프로그램인 로제타의 개발자 커뮤니티로, 가입을 위해서는 허가가 필요. 2022년 12월 RosettaCommons Github 기준 300여 명의 개발자가 등록. 기본적인 소스코드를 공유하면서, 필요에 따라 새로운 기능을 개발하여 추가하는 시스템
○ML4proteinengineering(https://www.ml4proteinengineering.com/)
╶︎인공지능 기반의 단백질 설계 연구 결과를 공유하는 학생 중심 커뮤니티. 격주로 관련 연구 분야의 학계, 산업계의 연사가 온라인 세미나를 진행하고, 격월로 패널 토론은 진행함
○한국바이오디자인학회(KIDDS: Korean in silico bioDesign and Discovery Society, https://www.kidds.or.kr/)
╶︎계산생물학 관련 컴퓨터 프로그램 개발과 단백질 공학 및 디자인 연구자들의 학술 교류 모임으로 2022년 창립되어, 운영되고 있음(회장 : 서울대 석차옥 교수)
3. 결론 및 시사점
□ 사용자 친화적 발전과 높아진 디자인 성공률
○인공지능 기반 기술과 클라우드 기반 단백질 디자인 서버의 증가로 접근성이 매우 높아짐
╶︎인공지능 기반 단백질 디자인 기술 개발자들이 공개한 코드와 구글 Colab과 같은 클라우드 서비스를 이용하면 고성능 컴퓨터 없이도 단백질 디자인이 가능해짐
╶︎알파폴드, RFdiffusion, ProteinMPNN과 같은 경우는 서버를 통하면, 간단한 인터페이스 환경에서 클릭 몇 번으로 작동이 가능함
○큰 도약을 보인 디자인 성공률
╶︎기존의 단백질 디자인 기술은 성공률이 1% 미만이었기 때문에, 대용량 스크리닝 기술이 필수적이었음. 이 수치마저 단백질 디자인 전문가 기준의 성공률로 비전문가는 현실적으로 성공이 매우 어려웠음
╶︎RFdiffusion 기술로 간단한 결합 단백질의 경우 10% 이상의 성공률로 보고됨. 이 정도 성공률은 대용량 스크리닝 없이도 확인 가능한 수준
╶︎RFdiffusion All-atom을 이용한 저분자 결합 단백질 디자인에서도 표적 물질에 따라 성공률의 편차가 있었지만(22.6 ~ 0.067%), 최대 22.6%(38/168)의 성공률을 보임
○간편해진 인터페이스 뒤에 숨겨진 원리 이해 필요
╶︎다양한 단백질 디자인 기술들이 개발되어도 결국 적용해야 하는 것은 사용자임
╶︎사용자는 다양한 디자인 모델들의 원리와 한계점을 이해하고 있어야, 목적에 부합하는 모델을 효과적으로 활용할 수 있을 것
□ 지평을 넓혀가는 단백질 디자인
○단백질 디자인 분야는 사실상 바이오의 거의 모든 분야로 적용 가능함
╶︎생명체 내의 단백질은 가장 다양하고 많은 기능을 하는 물질로, 단백질 디자인을 통해서 개선하고, 설계할 수 있는 단백질의 범위가 넓어지면 결국은 단백질이 체내에서 관여하는 모든 분야에 활용될 수 있음
○이에 따라 백신, 세포치료제, 바이오센서, 나노구조체, 나노포어 등 지속적으로 적용 분야를 확장해 나가고 있음
○신규 단백질 설계 기술의 개발은 적용 범위를 급격하게 확장시키고 있음
╶︎2022년 말 공개된 RF디자인 기술을 기반으로 한 결과가 2023년 다수 발표
╶︎2024년에는 RFdiffusion 기술로 설계한 기술들이 발표될 것으로 기대
╶︎De novo 효소 디자인 기술의 공개는 새로운 단백질 디자인 기술들과 접목되어 신규 단백질 개발이 가속화될 것으로 기대
○RFdiffusion All-atom의 개발로 영역확장이 가속화될 것으로 기대
╶︎단백질 간의 상호작용만으로도 큰 파급효과를 불러일으킨 단백질 디자인 분야
╶︎단백질 구조 데이터베이스 상에 존재하는 다양한 생체분자를 고려할 수 있게 되면서, 단백질 디자인 분야에서 새로운 시대로 도입이 될 수 있음
╶︎유전자 편집, 후생유전학 등 핵산 결합 단백질과 효소 단백질 디자인 분야 발전이 전망됨
○2억 개 이상의 알파폴드 예측구조 데이터베이스 기반의 구조 기반 클러스터링으로 새로운 가능성 발견
╶︎기존의 단백질 클러스터링은 아미노산 서열 유사성을 기반으로 이루어짐
╶︎최근 서울대학교 Martin Steinegger 교수와 스위스 바젤대학교 Joana Pereira 교수는 각각 알파폴드의 거대한 데이터베이스를 활용하여 단백질 서열뿐 아니라 구조 정보를 반영하여 클러스터링한 결과를 Nature에 보고(23’ Nature, 23‘ Nature)
╶︎아미노산 서열상의 유사성은 낮지만 구조적 유사성은 높은 경우가 존재하며, 이들은 기능적으로 유사한 예를 확인함
╶︎이는 아미노산 서열 데이터베이스의 주석을 획기적으로 업그레이드할 수 있으며, 이는 다시 서열 정보와 주석을 기반으로 하는 언어 모델 기반의 단백질 디자인 기술 발전의 발판이 될 수 있을 것으로 기대됨
□ 현재 단백질 디자인이 넘어야 할 문제
○동역학적 문제에 대한 해결방법이 부재
╶︎특정 기능을 위한 구조를 설계하고 이제 적합한 서열을 생성하는 방법으로 도출된 단백질은 많은 경우 매우 안정적인 상태이나, 이는 다시 말하면 유연성이 없다는 것을 의미
╶︎효소, 단백질 스위치, 분자 기계(Molecular machine)와 같이 더 복잡한 기능의 단백질은 구조적 변화를 필요로 하는 경우가 많으나, 현재 단백질 디자인 기술로 구조적 변화는 시작 단계임
╶︎동역학적 문제를 반영한 인공지능 개발을 위해서는 현재보다 큰 규모의 정제된 데이터베이스가 필요함
○장벽을 허물기 위한 협력과 커뮤니티의 필요성
╶︎본 내용에서는 실험적 데이터가 포함된 내용을 중심으로 작성되었지만, 실제로는 대다수의 단백질 설계 관련 인공지능 기술 개발 논문에서 실험적 데이터가 부재함
╶︎실험적 검증이 어려운 환경에 있는 인공지능 개발 연구팀이 대다수이고, 일부의 실험적 검증과 디자인 기술 개발이 가능한 연구팀에서 대부분의 성과를 달성함
╶︎일반적으로 실험적 검증은 시간과 비용이 소모되므로, 디자인 기술 개발 데이터와 in silico 검증 단계에서 논문으로 출간. 사용자 입장에서는 in silico 데이터만 기준으로는 수 많은 디자인 기술 중 선택의 어려움이 있음
╶︎협력을 통하여 기술 개발과 실험적 검증의 긍정적 피드백이 절실하고, 협력과 교류를 위한 커뮤니티가 필요
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.


 2023년 WG 이슈페이퍼_9호_인공단백질디자인 WG.pdf
2023년 WG 이슈페이퍼_9호_인공단백질디자인 WG.pdf
 단백질 디자인과 단백질 구조 예측의 관계
단백질 디자인과 단백질 구조 예측의 관계
 주요 단백질 디자인 인공지능 모델
주요 단백질 디자인 인공지능 모델