BioINpro
(BioIN + Professional) : 전문가의 시각에서 집필한 보고서 제공[KRIBB 워킹그룹] 2023 식물분야 연구동향
- 등록일2023-12-15
- 조회수2262
- 분류생명 > 생명과학, 생명 > 생물공학, 플랫폼바이오 > 바이오융합기술
-
저자/소속
이효준 외 14명/한국생명공학연구원 식물시스템공학연구센터
-
발간일
2023-12-15
-
키워드
#RNA 바이러스#유전자교정#단일세포 단백체학#멀티오믹스 정보구축#작물 형질전환
- 첨부파일
-
차트+
?
차트+ 도움말
2023 식물분야 연구동향
2023년도 KRIBB 워킹그룹이슈페이퍼 제 12호
◈ 목차
1. 개요
2. 주요 동향 및 이슈
2.1. RNA 바이러스를 이용한 유전자교정
2.2. 해충의 부화 유도 물질 발견
2.3. Cut-dip-budding 작물 형질전환
2.4. 공간적 단일세포 단백체학
2.5. 담배 식물의 멀티오믹스 정보구축
3. 결론 및 시사점
◈본문
1. 개요
□ 2023년 발표된 논문 분석을 통한 식물분야 동향 파악
○식물분야는 식물을 대상으로 연구하는 모든 연구분야를 일컫는 것으로, 식물의 특성이나 종의 다양성 때문에 매우 다양한 세부 연구분야가 존재함
╶︎따라서 하나의 이슈페이퍼에서 2023년 연구 동향을 모두 파악하는 것은 불가능하며 효율적이지 않음
○식물은 자연에 존재하는 자체로도 활용할 수 있지만, 인류가 식량ㆍ바이오매스ㆍ바이오소재 등을 활용하기 위해서는 생명공학 등의 기술을 활용하여 식물을 개량하는 과정이 필요함
╶︎보통 자연계에 존재하는 야생 식물은 자신의 생존과 번식에만 집중하여 진화해 왔기 때문에 인류가 활용하기 위해서는 유용한 특성을 강화시키는 개량 과정이 필요함
○따라서, 본 이슈페이퍼에서는 식물 생명공학을 중심으로 하여 학술적인 가치 뿐 아니라 향후 활용가치나 실용화 가능성이 있는 창의적인 연구를 위주로 선정하여 소개하고자 함
○또한, 본 이슈페이퍼에서는 2023년에 발표된 논문으로만 한정하여 올해 새롭게 제시된 기술이나 학술적인 내용 등을 통해 가장 최신의 연구동향을 파악하고자 함
○마지막으로, 해당 분야 전문가들의 의견을 취합하여 소개된 연구의 향후 활용 방안 및 미래 가치 등을 추가하여 동향 파악에 그치지 않고 연구 방향을 제시하여 국내 연구자들의 연구 및 기술개발에 도움을 주고자 함
2. 주요 동향 및 이슈
2.1. RNA 바이러스를 이용한 유전자교정
□ 논문 개요
○본 논문(Liu et al., 2023)은 2023년 3월 Molecular Plant지에 발표되었음
╶︎총 5명의 저자로, 중국 Zhejiang 대학에서 발표
╶︎2023년 Molecular Plant지의 IF=27.5, JCR < 1%
□ 연구 배경
○식물세포의 유전자교정을 위해서는 유전자교정 단백질 및 RNA가 coding되어있는 DNA를 전달하거나 RNAㆍ단백질을 전달해야 함
○하지만 식물세포는 동물세포와는 다르게 세포벽이 존재하여 DNA, RNA, 단백질의 전달이 매우 어려워 아직까지 효율적인 전달방법이 존재하지 않음
○현재 사용하는 대표적인 방법은 Agrobacterium을 이용한 것이나 장ㆍ단점이 뚜렷하여 한계가 존재함
╶︎Agrobacterium은 DNA를 식물 genome에 삽입시키기 때문에 외래 DNA가 삽입된 식물체가 만들어져 GMO 규제를 받게 됨
╶︎Agrobacterium이 식물세포를 감염시켜야만 DNA 삽입이 이뤄지는데, 식물의 종에 따라 박테리아의 감염에 저항할 수 있어 적용에 한계가 있고 효율이 크게 차이가 남
○다른 방법으로 세포벽을 제거한 protoplast 형태로 만든 다음 동물세포와 마찬가지의 방법으로 DNA, RNA, 단백질을 전달할 수 있으나 protoplast로부터 온전한 식물체를 분화시키는 과정이 매우 어려움
╶︎Protoplast로부터 식물체로 분화되기 위해서는 세포벽이 우선 재생되어야 하고, 세포 분열을 통해 캘러스가 만들어져야 하며, 캘러스로부터 잎과 줄기가 분화되어야 하는 다양한 단계가 필요한데, 각 단계의 효율이 낮고 종에 따라 효율이 다르며 다루는 사람이나 실험 환경에 따라서도 효율이 달라져 표준화된 방법이 존재하지 않음
○한편 바이러스를 이용하여 식물체 내 RNA나 단백질을 발현시키는 방법도 존재하지만, 대부분의 식물 바이러스는 매우 작은 크기의 RNA나 단백질만을 만들기 때문에 크기가 큰 유전자교정 단백질인 Cas 단백질을 만들어내지 못함
╶︎또한, 바이러스 역시 식물 종에 따라 감염 효율이 매우 크게 달라져 다양한 식물 종에 적용시키기 어려운 한계 존재
□ 연구 내용
○본 연구에서는 Tomato Spotted Wilt Virus(TSWV; 토마토반점시듦바이러스)라는 RNA 바이러스를 활용하였음
╶︎TSWV는 1000개의 식물 종을 감염시킬 수 있어 매우 활용 범위가 넓음
╶︎또한 TSWV는 다른 식물 바이러스에 비해 상대적으로 큰 유전자를 가지고 있어, Cas와 같이 사이즈가 큰 유전자를 발현시킬 수 있는 잠재력이 있음
○TSWV 바이러스가 가진 유전자들 중 식물 감염에 필요한 유전자는 보존하고, 그 외 유전자들 중 제거 및 치환이 가능한 유전자를 선별
╶︎Glycoprotein precursor(GP) 유전자는 바이러스의 모양이나 곤충으로의 전이에 관여하며, 크기가 3.4 kb이기 때문에 약 3.9~4.3 kb 크기의 Cas 유전자로 치환이 가능할 것으로 판단(그림 1)
╶︎Nonstructural protein S(NSs)는 식물체 내 감염을 지속시키고 곤충으로의 전이를 유도하는 유전자로, 크기가 상대적으로 작아 유전자교정에 필요한 작은 크기의 gRNA를 coding하는 유전자로 치환이 가능할 것으로 판단
╶︎저자들은 NSs 위치에 gRNA와 함께 GFP 유전자를 추가하여 감염 위치를 파악하기 위한 지표로 사용함
|
출처 : Liu et al., 2023 |
[그림 1] TSWV 바이러스의 유전자 재조합 및 식물 감염성 확인 |
○재조합한 TSWV 바이러스를 통한 유전자교정 확인
╶︎재조합한 유전자를 벡터의 형태로 만든 뒤 Nicotiana benthamiana 식물에 주입하여 바이러스가 형성되도록 유도
╶︎이후 형성된 바이러스가 Cas 단백질 및 gRNA를 잘 발현시키는지 확인
╶︎바이러스에 의해 Cas 단백질이 잘 발현되며, 3개의 교정대상 유전자인 NbPDS, NbFucT, NbDCL2 유전자가 효율적으로 교정(30-70%)된다는 것을 확인함(그림 2)
|
출처 : Liu et al., 2023 |
[그림 2] 재조합 TSWV 바이러스를 통해 유전자교정이 효율적으로 일어남을 확인 |
○식물체 내 바이러스는 기존 바이러스에 비해 재조합한 바이러스의 형태가 다르며, 그에 따라 곤충으로의 전이가 일어나지 않음을 확인
○또한, Cas 단백질에 추가적인 단백질이 연결된 형태인 base editor의 경우에도 최대 약 5.2 kb의 크기이지만 TSWV 바이러스가 효과적으로 발현시키며, 그에 따라 base editing도 잘 일어남을 확인 (그림 3)
╶︎A to G, C to T로 바꾸는 base editor 두 종류를 대상으로 확인
╶︎특히 C to T 전환의 효율이 높았으며, 3개의 유전자를 동시에 교정할 수 있음을 확인
|
출처 : Liu et al., 2023 |
[그림 3] 재조합 TSWV를 통해 base editing이 잘 일어남을 확인 |
○담배 뿐 아니라 담배에서 형성된 바이러스를 다른 종의 식물에 접종하는 방식을 통해 토마토, 고추, 체리, 땅콩 등 7개 종에서 비슷한 감염율과 유전자교정이 일어남을 확인
╶︎고추와 땅콩을 대상으로는 다양한 품종을 활용하여 품종과도 관계없이 유전자교정이 잘 일어남을 확인
╶︎다만 특정 품종에서는 바이러스에 의한 감염증상이 나타남
○TSWV 바이러스를 통해 유전자교정된 잎을 조직배양하여 식물체로 재분화시킴으로써 유전자교정 식물체를 만들 수 있음을 확인
╶︎토마토를 대상으로 검증하였으며, 바이러스 감염된 잎을 식물 호르몬이 들어간 배지에서 배양하여 식물체 재분화를 시킴 (그림 4)
╶︎이후 재분화된 개체의 선별을 통해 유전자교정이 유지된 개체를 확인함
╶︎특히 바이러스를 제거하기 위해 다양한 항바이러스제를 사용했으며, 그 중 ribavirin을 사용할 경우 거의 100% 바이러스가 제거된다는 것을 확인함
|
출처 : Liu et al., 2023 |
[그림 4] 재조합 TSWV를 통해 유전자교정된 잎을 조직배양하여 |
2.2. 해충의 부화 유도 물질 발견
□ 논문 개요
○본 논문(Shimizu et al., 2023)은 2023년 3월 Science Advances지에 발표되었음
╶︎총 11명의 저자로, 일본 Kobe 대학에서 발표
╶︎2023년 Science Advances지의 IF=13.6, JCR < 10%
□ 연구 배경
○식물 해충 중 하나인 nematode는 가장 피해 규모가 큰 해충 중 하나로, 주로 뿌리에 기생함
○그 중 Potato Cyst Nematode(PCN; 감자낭종선충)은 가지과 식물인 토마토 및 감자에 발생하며, 대략 전세계 감자 생산량의 9% 감소를 일으킨다고 알려져 있음
╶︎PCN은 식물의 뿌리에 알을 놓는데, 알이 식물에서 분비되는 물질을 인식하여 부화한다고 알려져 있음
╶︎알의 부화를 유도한다고 하여 이를 부화 유도물질, 즉, hatching factor라고 부름
╶︎기존에 알려진 부화 유도물질이 몇 종류 있지만, 이것으로 PCN 알의 부화를 전부 설명하지 못해 추가 물질이 존재할 것으로 예상됨
○콩과작물에 기생하는 Soybean Cyst Nematode(콩씨스트선충)의 경우에는 매년 15억 달러의 손실을 일으킨다고 알려져 있음
□ 연구 내용
○본 연구에서는 감자의 수경재배를 통해 뿌리에서 분비되는 물질을 분석하였음
╶︎HPLC(High Performance Liquid Chromatography)를 통해 뿌리에서 분비되는 물질을 분리하였고, 각 fraction의 혼합물을 PCN 알에 처리하여 부화 정도를 확인함
╶︎그 결과, 특정 fraction(22-24, 28-30)의 혼합물에서 PCN 알의 부화가 크게 유도된다는 사실을 발견함 (그림 5)
╶︎이후 해당 fraction의 혼합물에 포함된 물질을 면밀히 분석하여 두 종류의 물질인 solanoeclepin A(SEA)와 solanoeclepin B(SEB)를 분리함
|
출처 : Shimizu et al., 2023 |
[그림 5] 감자의 뿌리 분비물로부터 SEA와 SEB를 분리 실험 |
○같은 가지과 식물인 토마토의 뿌리 분비물을 분석한 결과, 감자와 마찬가지로 SEB가 분비됨을 확인함
╶︎하지만, 감자와는 달리 토마토의 경우에는 SEA가 거의 분비되지 않아, SEB가 더 각 작물에서 공통적으로 분비되는 물질로 파악됨
○SEB의 생합성 원리를 파악하기 위해, SEB의 구조와 유사한 triterpenoid 계열 물질의 생합성과 관련된 CYP와 DOX 효소 관련 실험을 수행함
╶︎CYP와 DOX 효소의 기능을 억제하는 억제제를 처리한 결과, 두 종류의 억제제 모두 SEB 합성이 줄어드는 것을 확인하여 두 효소가 SEB합성에 중요하다는 것을 파악함
╶︎그 중 DOX 효소는 기존에 SGA라는 물질을 합성한다고 알려져 있어, 같은 계열이지만 아직 기능이 밝혀지지 않은 5개의 유전자를 대상으로 실험을 진행함
○각종 식물 호르몬 처리를 한 뒤 유전자 발현과 뿌리 분비물의 부화 유도효과를 비교한 결과, SOLA1, SOLA2, SOLA3가 가장 상관관계가 높은 것으로 분석됨 (그림 6)
○유전자교정 기술을 통해 SOLA1의 기능을 제거한 토마토의 경우 뿌리 분비물에서 SEB가 검출되지 않아, SOLA1이 SEB 합성에 중요한 기능을 하는 유전자임을 밝힘
|
출처 : Shimizu et al., 2023 |
[그림 6] SOLA1이 SEB를 합성하는 유전자임을 규명 |
○유전자교정 기술을 통해 SOLA2와 SOLA3의 기능을 각각 제거한 토마토를 제작하여 분석한 결과, SOLA1과 마찬가지로 SOLA2와 SOLA3도 기능이 제거되면 SEB를 합성하지 못함을 확인함
╶︎각 유전자교정 식물체의 분비물은 PCN 알 부화 유도효과가 거의 없는 것을 확인
╶︎한편 SOLA 유전자 기능을 제거해도 전체적인 식물 바이오매스에는 큰 변화가 없는 것을 확인
○비슷한 방법으로 CYP 유전자를 분석하여, 추가로 SEB 합성에 관여하는 유전자인 SOLA4와 SOLA5를 동정함
○뿌리로부터 분비된 SEB가 흙에서 SEA로 바뀐다는 사실을 추가로 발견
╶︎따라서 식물은 SEB를 주로 분비하지만, 흙에서 SEA의 형태로 바뀌어 PCN의 알 부화를 유도하는 것으로 추측됨
2.3. Cut-dip-budding 작물 형질전환
□ 논문 개요
○본 논문(Cao et al., 2023)은 2023년 1월 The innovation지에 발표되었음
╶︎총 13명의 저자로, 중국 Shandong Shunfeng Biotechnology 회사 연구진에서 발표
╶︎2022년 The innovation지의 IF=33.1
□ 연구 배경
○식물의 유전자교정기술은 식물 연구에 혁명을 일으켰으며 작물의 형질을 크게 개선할 수 있는 잠재력을 가지고 있음.
╶︎유전자 편집 도구는 종종 Agrobacterium tumefaciens 매개 형질전환 또는 입자 충격이라는 두 가지 방법 중 하나를 통해 식물 세포에 전달되고 있음. 그러나 상기의 유전자전달방법을 통한 유전자교정 작물 개발의 효율은 작물의 종류나 품종 등 유전자형에 크게 영향을 받으며, 필연적으로 식물조직배양 과정이 요구됨
╶︎현재 애기장대의 경우 조직배양 과정 없이 형질전환 식물체의 개발이 가능함
○전 세계적으로 370,000종 이상의 고등 식물이 존재하며, 유전자 전달 시스템의 한계로 인해 유전적으로 변형될 수 있는 식물은 0.1% 미만임
╶︎‘Root suckering’ 식물은 뿌리에 있는 새싹 원기로부터 새로운 새싹이 자라나서 번식이 이루어지는 영양번식 작물로써, 많은 식물 종은 이러한 클론 번식 방법을 통해 뿌리에서 완전한 식물을 재생할 수 있음
╶︎조직 배양 없이 식물에 유전자 편집 도구를 전달하기 위해 식물 바이러스 기반 시스템을 설계하려는 많은 시도가 이루어지고 있으나 유전자교정 개체를 선발하기 위해서는 여전히 조직배양 과정이 요구되고 있음
○본 연구에서는 식물 조직배양과정 없이 유전자교정 작물 개발 시스템을 개발하였음. 유전자 전달 체계는 cut–dip–budding(CDB) delivery system으로 여러 식물 분류군에서 6종의 식물 종을 성공적으로 유전자 변형시켰음
╶︎본 시스템을 이용하면 조직배양 없이 유전자교정 식물을 얻을 수 있어 기존에 형질전환이 불가능했거나 형질전환이 어려웠던 식물종의 안정적인 유전자 교정이 가능할 것임
|
출처 : Cao et al., 2023 |
[그림 7] cut–dip–budding (CDB) delivery system을 통한 유전자 형질전환 모식도 |
□ 연구 내용
○본 연구에서는 러시아 민들레를 이용하여 CBD를 통한 유전자 형질전환 가능성을 우선적으로 검증하였음(그림 8)
╶︎3~4주된 러시아 민들레 묘목을 절취한 다음 절단 부위에 Agrobacterium rhizogenes K599로 접종하고 인큐베이터에서 배양하였음
╶︎A. rhizogenes에는 뿌리 유도 플라스미드가 포함되어 있기 때문에 약 2주 후에 털이 많은 뿌리(모상근)가 형성됨
╶︎모상근 중 일부는 GFP 양성으로 나타났으며, GFP 양성 뿌리를 영양이 풍부한 토양으로 옮겨 증식한 다음 야생종과 마찬가지로 뿌리로부터 줄기 발생을 유도함.
╶︎GFP 양성 뿌리로부터 녹색 새싹이 형성되었으며 GFP의 PCR 분석과 전송 DNA 삽입 부위의 TAIL-PCR 검출을 통해 새싹이 형질전환되었음을 확인하였음
|
출처 : Cao et al., 2023 |
[그림 8] CDB 전달 시스템 및 러시아민들레 유전자 형질전환 |
○다음으로 CDB 전달 시스템을 사용하여 유전자편집 도구를 전달할 수 있는지를 조사하였음(그림 9)
╶︎Phytoene desaturase(PDS)는 카로틴 생성 경로의 핵심 효소이며, PDS의 파괴로 인해 알비노 식물이 발생되므로 유전자 교정 식물을 쉽게 식별하기 위한 표적으로 선택되었음
╶︎저자들은 러시아 민들레의 PDS 유전자를 교정시키기 위해 sgRNA와 Cas9뿐만 아니라 리포터 GFP를 식물 세포에 동시에 전달하기 위해 GFP 카세트 앞에 AtU6 구동 sgRNA 및 AtUBQ1 구동 Cas9 발현 카세트를 삽입하였음
╶︎러시아 민들레가 이배체(2n = 2X = 16)임에도 불구하고 본 연구에 사용된 품종에는 PDS 유전자의 복사본이 3개 존재하며 실험결과 PDS의 세 개 사본이 모두 편집됨
╶︎40개의 explant에서 총 34개의 양성 모상근을 확보하였으며, 이 중 16개 양성 식물체를 확보하고 총 13개의 유전자 편집 식물체를 확보하였음
|
출처 : Cao et al., 2023 |
[그림 9] CDB 전달 시스템을 사용하여 러시아 민들레의 유전자 편집 |
○CDB 전달 시스템이 민들레 이외에 다른 종에 적용될 수 있는지 확인하기 위해 고구마(Ipomoea batatas)에 적용하였음(그림 10)
╶︎고구마 줄기 선단에 Agrobacterium rhizogenes K599로 접종하고 인큐베이터에서 배양하였음
╶︎약 3주 후에 GFP 양성 형질전환 모상근이 형성되었으며 유도된 모상근을 토양으로 옮겨 증식시킨 후 비대해진 뿌리로부터 새싹을 유도함
╶︎GFP 양성 뿌리에 형성된 모든 새싹은 유전자가 도입된 양성 형질전환체 식물임
|
출처 : Cao et al., 2023 |
[그림 10] CDB 전달시스템 기반 고구마 형질전환체 개발 |
○CDB 전달 시스템이 목본 식물에 적용될 수 있는지 확인하기 위해 Ailanthus altissima (Mill) Swingle, Aralia elata (Miq.) Clerodendrum chinense 식물을 대상으로 조사한 결과 목본식물체 적용가능함을 확인함
○또한 콩과 목초식물인 Coronilla varia L.을 형질전환한 결과 CDB 전달 시스템이 잘 작동함을 확인함으로써 CDB 전달시스템의 광범위한 적용 가능성을 확인하였음
2.4. 공간적 단일세포 단백체학
□ 논문 개요
○본 논문(Zeng et al., 2023)은 2023년 6월 Science지에 발표되었음
╶︎총 13명의 저자로, 미국 MIT 연구소와 Harvard 대학에서 발표
╶︎2023년 Science지의 IF=56.9, JCR < 1%
□ 연구 배경
○유전자조절이 전사수준과 단백질번역수준 모두에 의해 영향을 받기 때문에 전사체와 단백체의 상이성이 존재하므로 유전자발현과 번역조절에 대한 포괄적 이해를 위해서는 단일세포 및 공간적 단백질 합성 프로파일링이 필요
○기존연구는 대량 및 단일세포 리보솜 프로파일링을 통해 전사체 규모에서 단백질 번역을 분석할 수는 있었으나 현재의 이미징 기반 방법으로는 공간적 정보는 얻을 수 없음
○이를 해결하기 위해 본 논문에서는 단일세포 또는 세포 소기관 수준에서 3차원 in situ 리보솜-결합 mRNA 맵핑(RIBOmap, 리보맵) 방법을 개발
□ 연구 내용
○리보맵 디자인과 가능성 확인
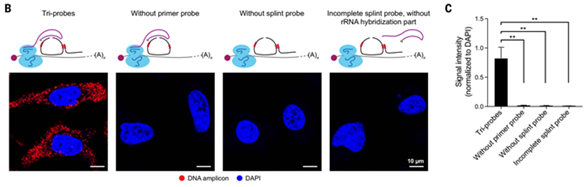
╶︎단백질이 번역 중인 mRNA를 선택적으로 감지하기 위한 전략을 위해 3개의 프로브를 사용함으로써 단일세포 수준에서 단백질 합성의 공간적 지도를 작성하도록 디자인 (그림 11A)
╶︎자물쇠(padlock) 프로브: 바코드를 포함하고 특정 mRNA를 결합
╶︎프라이머(primer) 프로브: 바코드를 포함하고 이를 통해 자물쇠 프로브와 결합하면 DNA 복제
╶︎부목(splint) 프로브: 리보솜 RNA와 결합하여 자물쇠 프로브를 원형화
╶︎결론적으로, HeLa cell에서 ACTB mRNA의 리보맵 형광신호를 탐지 (그림 11B, C)
|
출처 : Zeng et al., 2023 |
[그림 11] 리보맵을 통한 세포소기관 수준의 in situ mRNA 단백질 번역 |
○리보맵의 타깃 리보솜-결합 mRNA에 특이적 작동을 검증하기 위한 4가지 실험을 진행
╶︎ACTB mRNA에 대한 두 개의 네거티브 비암호화 RNAs(ncRNAs)를 사용한 실험결과 리보맵은 ACTB-특이적인 형광신호를 보임, 하지만 기존의 STAR맵은 네거티브에서도 형광신호를 보임(그림 12A-C)
╶︎두 번째는 시작 코돈에서 단백질 개시 리보솜의 트랩을 방해하는 화합물 Harringtonine 처리에 의한 리보맵 형광 시그널 감소 확인(115-405 nt downstream of the start codon) (그림 12D-G)
╶︎세 번째는 지질막 주입을 이용한 세포질에서의 단백질 번역 억제 시 리보맵 형광 시그널 감소 (그림 12H-J)
╶︎네 번째는 단백질 번역 중인 리보솜을 분리하는 RiboLace 결과와 리보맵의 분석을 비교한 결과 일치함을 확인
╶︎결론적으로, 리보맵 기술은 리보솜 결합 mRNA를 검출 특이성이 입증되었음
|
출처 : Zeng et al., 2023 |
[그림 12] 리보맵 특이성 검증 |
○세포배양을 통한 다중화 리보맵
╶︎리보맵의 세포성장 단계 및 주기별 차별화된 특정 번역 정보 제공 실험
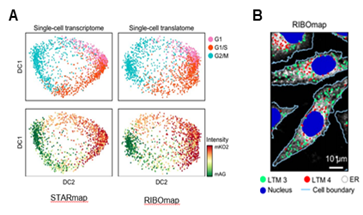
╶︎HeLa 세포를 이용한 세포소기관 981개 유전자의 단백질번역 리보맵 결과, 세포주기 의존적 패턴과 일치 (그림 13A)
╶︎세포 소기관 소포체-특이적 단백질 번역 리보맵 결과 (그림 13B)
|
출처 : Zeng et al., 2023 |
[그림 13] 리보맵을 통한 세포 주기 및 소기관-특이적 in situ mRNA 단백질 번역 리보맵 |
○마우스 뇌조직을 이용한 리보맵의 공간적 아틀라스
╶︎리보맵의 마우스 뇌조직의 세포 유형별, 조직영역별 번역 상태와 조절 상황을 분석 (그림 14A-C)
╶︎온전한 뇌조직을 이용하여 리보맵의 공간적 아틀라스를 증명
|
출처 : Zeng et al., 2023 |
[그림 14] 리보맵을 통한 세포 종류-특이적 조직영역-특이적 단백질번역 정보 |
2.5. 담배 식물의 멀티오믹스 정보구축
□ 논문 개요
○본 논문 (Ranawaka et al., 2023)은 2023년 10월 Nature Plants지에 발표되었음
╶︎총 26명의 저자로, 호주 Queensland 대학에서 발표
╶︎2023년 Nature Plants지의 IF=18.0, JCR < 2%
□ 연구 배경
○식물은 현재의 광범위한 분자농업 플랫폼으로 사용하기에 적합한 기본 재료로 여겨지며, 이를 활용하여 다양한 바이오의약품, 기능성식품, 바이오소재, 바이오연료 등의 생산이 가능함
○다양한 식물 중 형질전환이 수월하고 높은 대사 다양성과 고수율의 재배 능력을 가진 담배(Nicotiana benthamiana)는 현재의 분자농업 플랫폼으로 사용하기에 최적의 ‘Chassis’로 여겨짐
╶︎담배는 특히 바이오의약품 단백질 및 백신 생산을 위한 매우 중요한 식물 플랫폼이며, RNA 간섭(RNAi), 식물-병원체 상호작용, 대사경로 공학, 기능 유전체학, 합성생물학 및 유전자 편집의 근본적인 발견에 중요한 역할을 하였음
○바이오소재 생산 시스템으로 담배 식물이 갖는 장점에도 불구하고, 연구를 위해 필수적인 요소 중 하나인 기존 표준유전체의 완성도가 낮아 활용이 어려웠음
╶︎N. benthamiana는 ~3Gb의 유전체 사이즈를 가지며, 이질사배체(allotetraploid)의 유전체이기 때문에 정확한 유전체를 구축하는 데 어려움이 있음
□ 연구 내용
○N. benthamiana는 N. sylvestris와 N. glauca의 교잡으로 유도된 4배체 개체(ancestral Suaveolentes progenitor)가 배수체화 현상(polyploidization) 이후 일어나는 이배체화 현상(diploidization)을 통해 48개의 염색체가 38개로 줄어든 유전체 구조를 가지고 있음 (그림 15A)
○N. benthamiana는 다양한 환경에서 분리 동정된 다양한 accession이 존재하며, 본 논문에서는 LAB과 QLD accession을 유전체 분석에 활용하였음 (그림 15B)
╶︎LAB accession은 연구에 널리 사용되는 accession이며, QLD는 야생종에 가까운 accession임
|
출처 : Ranawaka et al., 2023 |
[그림 15] N. benthamiana의 표현형 및 다양성 |
○N. benthamiana의 유전체 재배열 분석을 위해서, N. sylvestris와 N. tomentosiformis에서 유도된 N. tabacum과 N. benthamiana간의 상동 유전자 블록(syntenic block)을 활용하여 유전체 구조 분석을 수행하였음 (그림 16)
╶︎N. tabacum의 경우, N. sylvestris와 N. tomentosiformis의 하위게놈(subgenome)으로 명확하게 분리되는 것을 확인할 수 있으며, subgenome간의 유사성도 확인할 수 있었음 (그림 16A)
╶︎N. benthamiana의 경우, 이배체화 현상에 의한 활발한 유전체 재배열(genome rearrangement) 현상으로 인해, N. tabaccum과 달리 하위게놈 간의 분리가 명확히 일어나지 않으며, 상동 유전자 블록도 상동 염색체가 아닌 다른 염색체에서 탐색되어 활발한 유전체 재배열 현상이 일어났음을 보여줌 (그림 16A와 16B)
╶︎일반적인 4배체 유전체는 2쌍의 상동 염색체가 존재하지만 N. benthamiana의 경우, 배수체화 현상 이후 일어난 이배체화 현상에 의한 염색체 손실로 인해 상동 염색체가 소실된 염색체 쌍을 확인할 수 있었음 (그림 16C)
╶︎LAB과 QLD에서의 상동 유전자 쌍(homeologous gene pair)의 분포를 비교한 결과, 50% 이상의 상동 유전자 쌍에서 대응되는 유전자의 소실이 확인되었으며, 이는 활발한 이배체화 현상에 따른 유전체 재배열 현상을 뒷받침해 줌 (그림 16D)
|
출처 : Ranawaka et al., 2023 |
[그림 16] N. benthamiana의 하위게놈 구조 |
○N. benthamina의 LAB 및 QLD 유전체와 같은 가지과 근연종과의 유전체 구조를 비교한 결과 가지과에서의 유전체 상동성을 확인할 수 있었음 (그림 17)
╶︎가지과 전반적인 유전체 구조 분석 결과, N. benthamiana 유전체 기준으로 1, 2, 3, 4 염색체에서는 높은 유전체 구조의 상동성을 확인할 수 있었음 (그림 17A)
╶︎이종상동성 유전자(orthologous gene)의 분포 탐색 결과에서도 1~4번 염색체에서 높은 비율의 이종상동성 유전자의 보존을 보이는 반면, 나머지 염색체에서는 이종상동성 유전자의 보존 비율이 낮아진 것을 확인할 수 있었음. 하위게놈 간의 유전자 비교에서도 A 유전체와 B 유전체에서의 상동성이 상실된 유전자의 비율이 비슷한 것을 확인할 수 있었음 (그림 17B와 17C)
╶︎N. benthamiana 유전체의 경우, 4번 염색체에서 활발한 유전체 재배열 현상으로 인한 빠른 유전적 상동성이 소실된 것을 확인할 수 있었음 (그림 17B와 17D)
╶︎토마토 유전체와의 비교에서도 1~4번 염색체에서 높은 보존율을 보여, 이와 같은 현상을 확인할 수 있었음 (그림 17E)
|
출처 : Ranawaka et al., 2023 |
[그림 17] N. benthamiana와 가지과 근연종의 유전체 구조 비교 |
○LAB과 QLD accession 간에 형질전환, 유전자 편집, RNA 간섭을 비교해 본 결과 대부분 유사한 효율을 보이나, 일시적 발현 시스템을 통한 단백질 발현에서는 LAB accession이 상대적으로 매우 높은 발현율을 보임 (그림 18)
○LAB과 QLD accession은 형태학적으로나 대사 다양성에 차이를 보이지만 특히 생장과 발달 속도에서 큰 차이를 보임
╶︎QLD accession은 LAB에 비교하여 종자 형성까지 2배 가까운 시간이 소요됨
○LAB accession은 QLD에 비교하여 빠른 생장과 고수율의 단백질 발현을 통해 현재 분자농업의 최적 플랫폼으로 선택되었음
|
출처 : Ranawaka et al., 2023 |
[그림 18] LAB과 QLD accession에서 일시적 단백질 발현 비교 결과 |
3. 결론 및 시사점
□ RNA 바이러스를 이용한 유전자교정
○TSWV 바이러스는 매우 다양한 식물 종을 감염시킬 수 있으므로 본 시스템을 이용하여 더 많은 식물 종에 적용시킨다면 외래 유전자가 남아있지 않은 유전자교정 식물체를 효율적으로 만들어 낼 가능성이 있음
○또한 TSWV 바이러스를 재조합하는 방식을 활용한다면 Cas나 gRNA 대신 다른 유용한 유전자를 식물 내 발현시킬 수 있고, 이는 유전자의 종류에 따라 다양한 시도가 가능함
╶︎예를 들어 식물체 재분화를 유도할 수 있는 유전자를 도입한다면, 유전자 삽입 없이 바이러스 감염된 식물 잎을 조직배양하여 재분화가 어려운 식물 종의 재분화를 유도할 수 있을 것으로 예상됨
○다만 TSWV 바이러스의 특허가 향후 기술개발에 문제가 될 수 있으므로, 국내 자생식물 조사를 통해 새로운 형태의 TSWV를 찾고 재조합하는 형식의 추가 연구가 필요해 보임
□ 해충의 부화 유도물질 발견
○PCN은 전세계적으로 가지과 작물의 생산량 및 수익에 큰 악영향을 미치는 해충으로, PCN을 방제하기 위해 PCN 알의 부화에 대한 연구가 필요함
○해충의 부화 유도물질을 발견함으로써, PCN이 식물이 분비하는 물질 중 SEB와 SEA를 인식하여 부화하여 식물의 존재를 파악한다는 것을 확인함
○또한 SEB를 합성하는 유전자를 동정하여, PCN에 대해 저항성이 생긴 작물을 제작할 수 있는 유전자를 제시하였음
○이를 통해 PCN의 피해가 심한 작물 종을 대상으로 저항성을 높인 작물을 개발할 수 있으며, 추가로 본 논문의 연구 방법을 도입하여 뿌리 분비물을 인식하여 부화하는 다양한 nematode에 대한 방제 기술을 개발할 수 있음
○이는 유전자교정을 통해 해충을 방제하는 것이기 때문에 농업에서 살충제의 양을 줄여 환경오염을 방지하는 효과도 함께 기대할 수 있음
□ Cut-dip-budding 작물 형질전환
○CDB를 통한 유전자 형질전환 및 유전자교정
╶︎고효율 유전자 전달방법의 개발은 육종에 있어 가장 큰 장애물임
╶︎현재 대부분 작물에서의 유전자 전달 방법은 지루하고 비용이 많이 드는 조직 배양 과정에 의존하고 있으며 매우 소수의 식물에만 적용될 수 있음
╶︎본 연구에서 확립된 CDB 전달 방법은 조직 배양이 필요하지 않으며 매우 간단하며 잠재적으로 많은 작물에 적용 가능함
╶︎식물체 재분화를 촉진할 수 있는 유전자를 도입하게 되면 재분화가 어려운 식물 종의 재분화를 유도할 수 있을 것으로 예상됨
○본 연구는 유전자교정작물 개발의 가장 큰 병목요인 중 하나인 조직배양 과정에서 분화조절을 극복할 수 있는 대안을 제시함
○CDB 전달 시스템을 사용하여 뿌리를 통한 영양번식이 가능한 모든 식물 종의 유전자 편집이 가능할 것으로 기대되며 특히 사과, 포도, 배, 복숭아 등 유실수와 딸기, 블루베리, 블랙베리, 레드 라즈베리 등 경제적으로 중요한 열매 식물의 유전적 개선을 촉진할 수 있을 것으로 예상됨
□ 공간적 단일세포 단백체학
○리보맵의 염기서열-특이성에 의한 한계점
╶︎알려지지 않은 mRNA에 대한 프로브 제작이 불가능하므로 현재까지 염기서열이 알려진 mRNA의 번역 상황을 분석한다는 한계점이 존재
╶︎하지만 복잡한 조직, 세포 수준에서의 mRNA를 공간적으로 프로파일링하게 되면 서열 특이적 RNA-단백질 상호작용과 번역에 대한 모니터링이 가능
╶︎RNA의 역동적 단계적 생애주기를 알 수 있고 외부 환경에 의한 변화를 모니터링할 수 있어 관련 기작 연구에 큰 도움이 될 것임
○리보맵을 통한 유전자발현 조절 이해 가속화
╶︎3가지 프로브를 이용한 리보맵은 단백질번역 뿐 아니라 mRNA 분해 속도를 연구할 수 있으며 RNA 생애 주기별 분석이 가능함
╶︎환경변화에 의한 RNA 변형 등으로 RNA 안정성과 단백질 번역으로의 영향 등은 게놈 수준의 공간적 아틀라스 형성에 중요한 메커니즘을 제시할 수 있음
╶︎손상되지 않은 intact 세포 및 조직에서 mRNA 조절과 단백질 합성 연구가 가능함
╶︎리보맵은 복잡한 폴리솜 분리 및 유전자조작을 우회하여 단일세포 수준에서 연구가 가능함
○리보맵의 활용 가능성
╶︎다른 이미징 기반 기술과 융합하여 후성유전체, 전사체, 단백제와의 공간적 다중 맵핑이 가능, 이를 통해 생물학적 시스템의 통합적 이해를 증진시킬 수 있음
╶︎RNA 번역을 후성학적 제어, 단백질 발현 및 대사기능과 연결할 수 있음
╶︎생명현상의 원리를 세포수준, 조직학적 수준에서 이해하는 데 기여할 수 있음
□ 담배 식물의 멀티 오믹스 정보 구축
○담배(Nicotiana benthamiana)는 높은 형질전환 효율, 높은 대사 다양성 및 고수율의 재배 능력을 가지며, 다양한 바이오의약품, 기능성식품, 바이오소재, 바이오연료 등의 생산을 위한 플랫폼으로 사용하기에 최적의 식물임
○상대적으로 큰 유전체 사이즈(~3Gb) 및 유전체의 복잡성(allotetraploid) 때문에 완성도가 낮아 활용이 어려웠던 기존의 표준유전체 정보를 향상시켜 다른 가지과 작물인 토마토, 고추, 가지, 감자 등과 유사한 품질의 완성도를 갖도록 하였음
○연구에 널리 사용되는 LAB accession은 야생종에 가까운 QLD에 비교하여 빠른 생장과 고수율의 단백질 발현을 통해 현재 분자농업의 최적 플랫폼으로 선택되었음을 확인함
○새로 구축된 담배(N. benthamiana)의 다양한 오믹스 정보는 향후 담배 식물을 이용한 대사, 발달 및 진화 연구를 크게 향상시키는 리소스를 제공할 것으로 기대됨
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
동향


 2023년 WG 이슈페이퍼_12호_식물 WG.pdf
2023년 WG 이슈페이퍼_12호_식물 WG.pdf
 TSWV 바이러스의 유전자 재조합 및 식물 감염성 확인
TSWV 바이러스의 유전자 재조합 및 식물 감염성 확인