제도동향
유산균종 확인 메타게놈 분석 프로그램 사용자 매뉴얼 마련 및 배포
- 등록일2019-08-22
- 조회수6098
- 분류제도동향 > 종합 > 종합
-
자료발간일
2019-08-13
-
출처
식품의약품안전처 식품의약품안전평가원
- 원문링크
-
키워드
#유산균종 확인#메타게놈#종 분석
- 첨부파일
유산균종 확인 메타게놈 분석 프로그램 사용자 매뉴얼 마련 및 배포
[목차]
1. 유산균종 확인 메타게놈 분석 프로그램 소개
2. 메타게놈 분석 프로그램 설치 및 실행
3. 유전체 데이터의 생산
4. 종 검출 및 상대 비율 분석
4.1 기본설정
4.2 분석 수행
5. 결과보고서
[내용]
1. 유산균종 확인 메타게놈 분석 프로그램 소개
유산균종 확인 메타게놈 분석 프로그램은 차세대 염기서열 분석(NGS) 장비를 통해 확보된 미생물 염기서열 정보와 식품에 존재하는 유산균을 비교 · 분석하여 정확한 유산균종을 판별하는 프로그램입니다. 이 프로그램은 각 유산균의 종별 대표 균종을 선정하여 구축한 데이터베이스에 대용량 유전체 염기서열 정보를 정렬하여 검출 종을 판별합니다. 또한 유산균종별 유전체 상도 (mapping depth)를 활용하여 제품 내 상대적인 비율을 제공합니다.
메타게놈 분석 프로그램의 구성은 아래 그림과 같습니다.
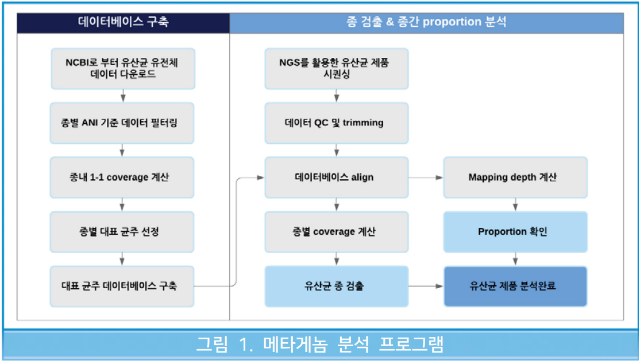
이러한 구성을 기반으로, 메타게놈 분석 프로그램은 사용자 편의성 향상을 위하여 대용량의 유전체 염기서열 정보 입력만으로 시각화된 분석결과가 나타날 수 있도록 고안되었습니다.
2. 메타게놈 분석 프로그램 설치 및 실행
메타게놈 분석 프로그램은 windows10 버전 1607 이상의 환경에서만 실행이 가능합니다. 분석에 있어서 python, R, TRIMMOMATIC, Bowtie2, Samtools 등 다양한 프로그램을 필요로 하며 자세한 설치 방법은 아래와 같습니다.
Step 1. Xming 설치
Program/Xming-6-9-0-31-setup.exe를 더블클릭하여 Xming을 설치합니다.
Step 2. 윈도우 for Ubuntu 설치
window 버전 1607 이상인지 확인합니다.
개발자 모드를 활성화 합니다.
설정 - 업데이트 및 복구 - 개발자용 (경우에 따라 재부팅을 요구합니다.)
Linux용 windows 하위 시스템을 설치합니다.
설정 - 검색 [windows 기능] - windows 기능 켜기 / 끄기 - 재부팅
cmd 창을 관리자 계정으로 열어서 lxrun / install을 입력합니다.
(삭제 시 : lxrun / uninstall / y)
Step 3. Ubuntu 내 패키지 설치
***아래 여섯 문장을 Ubuntu console 창에서 실행합니다.
sudo apt-get update
sudo apt-get upgrade
sudo apt-get install -y openjdk-8-jdk \
build-essential checkinstall \
libreadlinge-gplv2-dev libncursesw5-dew
libssl-dev libgtk-3-dev tk-dev libgdbm-dev libc6-dev libbz2-dev \
libgtk2.0-dev libgtk-3-dev libjpeg-dev libtiff-\
libsdl1.2-dev libgstreamer-plugins-base0.10-dev libnotify-dev freeglut3
freeglut3- dev libsm-dev \
libwebkitgtk-dev libwebkitgtk-3.0-dev \
python-minimal \
python-pip \
r-base-core
echo deb https://cloud.r-project.org/bin/linux/ubuntu xenial/ >>
/etc/apt/sources.list
sudo apt-get upgrade
sudo apt-get install r-base
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.


 유산균종+확인+메타게놈+분석+프로그램+사용자+매뉴얼.pdf
유산균종+확인+메타게놈+분석+프로그램+사용자+매뉴얼.pdf