기술동향
DNA 메틸화의 반전: 메커니즘, 유전체학, 생물학적 기능
- 등록일2015-04-28
- 조회수10241
- 분류기술동향
-
자료발간일
2015-04-23
-
출처
BRIC VIEW
- 원문링크
-
키워드
#DNA#메틸화#메커니즘#유전체학#생물학
- 첨부파일
출처 : BRIC VIEW
DNA 메틸화의 반전: 메커니즘, 유전체학, 생물학적 기능
저자 정현석 (The University of Tokyo)
본 자료는 Reversing DNA methylation: Mechanisms, genomics, and biological functions. Cell 2014; 156(1-2): 45-68 의 논문을 한글로 번역한 자료입니다.
목차
1. 서론
2. 포유류의 DNA 메틸화 및 탈메틸화 : 순환효소 연쇄반응
Box1) 5mC의 능동제거에 관한 알려지거나 추정되는 메커니즘
2.1 TET 단백질에 의한 5mC의 반복적인 산화
2.2복제의존적 희석에 의한 산화된 5mC의 C로 수동 복구
2.3 효소적 제거에 의한 산화된 5mC의C로 능동 복구
2.4 TET/TDG에 의한 복제 비의존성 DNA 메틸화의 중간 단계들
3. TET 의존성 DNA 탈메틸화 메커니즘
3.1. TET 효소 기질 특이성 및 접근성에 의한 조절
3.2. 대사물질과 보조인자에 의한 효소 활성 조절
3.3. TET 효소의 유전자 표적화에 의한 조절
3.4. TET 수준의 전사적 및 전사 후 조절
4. 산화된 메틸사이토신의 유전체 분포화 전사 조절에서의 가능한 역할
Box2) 5mC 산화물을 위한 유전자 전체 매핑 기술
4.1. 산화된 5mC 유전자 전체 매핑 기술
4.2. 유전자 프로모터에서의 산화된 메틸사이토신
4.3. 유전자체 내의 산화된 메틸사이토신
4.4. 말단 시스작용요소에서 산화된 메틸사이토신
4.5. 가능한 후생유전학적 마커로서의 산화된 메틸사이토신
5. 다양한 생물학적 과정에서 DNA 메틸화의 생물학적 기능
Box3) 전사 조절에서 TET 단백질의 효소 활성화 비의존성 역할
5.1. 착상 전에서 DNA 메틸화 동역학
5.2. 만능분화 줄기세포와 재프로그래밍의 DNA 메틸화
5.3. 생식세포 발달 과정에서 DNA 탈메틸화 동역학
5.4. 뉴런 발달 과정에서의 DNA 탈메틸화 역할
5.5 DNA 탈메틸화와 암
6. 결론 및 전망
1. 본문
유전체(genome)에서 유전 암호의 일부인 사이토신은 피리미딘 고리(pyrimidine ring)의 화학적인 변화를 통하여 후생유전적 정보를 전달하는 것으로 알려져 왔다. 사이토신과 연관된 이중 기능은 발달 단계 및 세포형(cell-type) 특이적인 후생유전학적 기억을 DNA 그 자체에 보존할 수 있는 수단을 제공한다. 사이토신의 5번째 위치 탄소의 메틸화(5-methylcytosine, 5mC)는 동식물, 균류에서 잘 보존된 후생유전학적 변화이며 유전체의 안정성, 유전자 발현 및 발생에 지대한 영향을 미친다[5, 6]. 포유류에서 새로운 DNA 메틸화 패턴은 새로이 합성된(de novo) DNA 메틸기전달효소 (methyltransferase)인 DNMT3A와 DNMT3B에 의해 조절된다 (그림 1 및 그림 2). DNMT3A 및 DNMT3B의 활성화는 촉매적으로 불활성화된 효소군인 DNMT3L에 의해 조절될 수 있다. 체세포에서 5mC는 역보상성
(palindrome) CpG 다이뉴클레오타이드(dinucleotides)로 제한되며, 다이뉴클레오타이드 (dinucleotides)는 대칭적인 방법으로 메틸화된다 (그림 4). CpG가 아닌 곳(non-CpG)에서의 메틸화는 식물에서 흔하지만 포유류 세포형(cell-type)에서는 희귀하다. 최근의 연구는 non-CpG는 난자(oocytes), 만능배아줄기세포(pluripotent embryonic stem cells, ESCs) 및 성숙된 신경 세포(neurons)에서 상대적으로 흔함을 시사하지만. 포유류 non-CpG 메틸화의 기능은 명확하지 않다. 인간 유전체는 약 2800만개의 CpG를 가지고 있는데 체세포에서는 60-80%의 CpG가 메틸화되어 있다. 감수분열(mitosis)에서는 전체의 CpG 패턴이 DNA 메틸기전달효소인 DNMT1와 절반이 메틸화된(hemimethylated) CpG를 인식하는 ubiquitin-like plant homeo-domain and RING finger domain 1 (UHRF1)에 의해 딸세포로 전달된다. 이와 같은 CpG의 유전은 유전자 발현의 침묵(silencing), 유전체 안정성 유지, 유전자 각인(genomic imprinting)과 같은 다양한 생물학적 과정에 요구되는 장기간의 후생유전학적 조절에 있어 5mC (5-methylcytosine)의 역할을 시사한다.
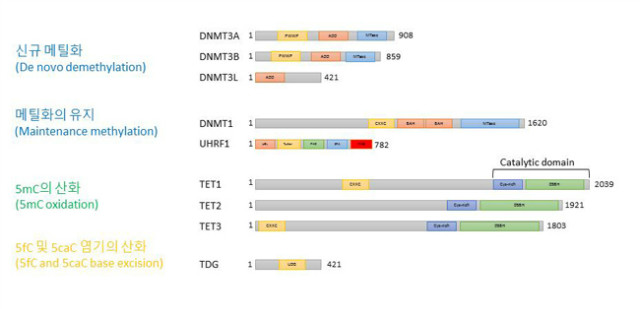
그림 1. 신규 메틸화, 메틸화 유지, 5mC 산화, 5fC/5caC 절단과 관련된 단백질의 기능 도메인
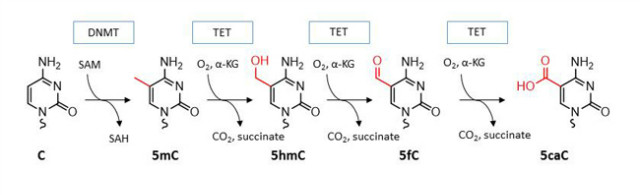
그림 2. 사이토신의 단계적인 변형 경로
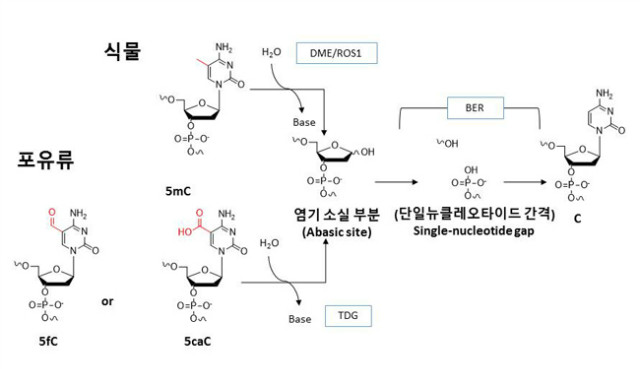
그림 3. DNA 탈메틸화를 완성하기 위한 5mC(식물) 또는 5fC/5caC(포유류)의 염기 절단 복구(base excision repair)
체세포에서의 DNA 메틸화 패턴은 안정적으로 유지되지만, 유전체 전체의 5mC의 손실 또는 DNA 탈메틸화는 착상전의 배아와
원시생식세포(primodial germ cell, PGC)와 같은 특별한 발생 단계에서 관찰된다. 전반적인 DNA 탈메틸화는 초기 배아의 만능
상태(pluripotent states) 를 설정하고 발달하는 PGC에서 부계 유래의 특이한 각인(imprint)를 제거하는데 중요하다. 이 두
가지 주요한 후생유전학적 재프로그래밍에서 신속한 5mC의 제거는 5mC의 복제의존성 수동 손실(replication-dependent
passive loss)에 의해 완전히 설명되지 않으며 이는 사이토신의 메틸기를 능동적으로 삭제하거나 변형하는 효소의 존재를 암시한다. 그러나
현재까지 포유류 세포에서 능동적인 DNA 탈메틸화 과정을 이해할 수 있는 통일된 메커니즘은 아직 나타나지 않았다. 여기서 토론하는 바와 같이,
5mC 산화효소로서 TET (ten-elven translocation)의 전환적인 발견은 능동 DNA 탈메틸화의 메커니즘에 대한 이해를 도와줄
것이다. 5mC의 산화적 변형에서 TET 효소에 대한 생화학적 이해는 최근에 많이 리뷰되었다. 이 리뷰에서는 포유류 DNA 탈메틸화 과정의
메커니즘, 유전체학, 생물학적 기능에 대한 포괄적인 이해에 초점을 맞출 것이다. 첫 번째로 현재의 수동 및 능동 DNA 탈메틸화 과정의 현재
메커니즘 이해를 요약할 것이다. 두 번째로 포유류 유전체에서 산화된 사이토신의 잠재적 조절 기능에 대한 이해뿐만 아니라 5mC 산화물을 위한
유전체 매핑 기술의 최근의 발전을 조사할 것이다. 마지막으로 DNA 탈메틸화 동역학(dynamics)의 별개의 방식들이 다양한 생물학적 경로의
필요성을 맞추기 위해 사이토신 변형을 촉매하는 경로의 조절을 통해 어떻게 달성될 수 있는지에 대해 토론할 것이다.
2. 포유류의 DNA 메틸화 및 탈메틸화: 순환 효소
연쇄반응
DNA 탈메틸화는 수동적(passively) 또는 능동적(actively)으로 일어날 수
있다. DNA 복제 과정에서 DNA 메틸화 유지 기구인 DNMT1/UHRF1은 초기 DNA 가닥에서 변형되지 않은 사이토신을 메틸화함으로써 대칭
CpG 메틸화 상태를 복구한다. 5mC의 수동적인 손실은 DNMT1/UHRF1이 존재하지 않을 때 연속적인 DNA 반복 복제에 의해 이루어질 수
있다 (그림 4의 passive dilution). 5mC의 복제 의존적 수동 손실에 대한 한가지 예는 마우스의 착상 전 발달 단계의 모계
유전체에서 5mC의 전반적인 손실이다.
주목할만한 증거는 식물 및 포유류에서 5mC가 DNA 복제과정과 무관하게 급속히 제거될 수
있음을 암시하고 있다. 이와 같은 변화는 능동 DNA 메틸화 과정을 포함하는데 5mC를 변형하거나 제거하는 하나 또는 그 이상의 효소 반응을
요구한다. 꽃을 피우는 식물에서는 DNA 글리코실화 효소(glycosylases) 군인 Demeter(DME)/repressor of
silencing 1 (ROS1) 및 염기 절단 복구(base excision repair, BER) 기구가 이를 수행한다 (그림 3). 그러나
포유류 세포에서는 상동성을 가지는 DME/ROS가 오랫동안 밝혀지지 않았다. 그리하여 포유류 세포는 다른 메커니즘으로 능동 DNA 메틸화를
수행한다. 초기의 노력은 DNA 복구 효소, 사이토신 탈아미노화 효소(deaminases), DNA 글리코실화 효소(glycosylates) 와
같은 알려진 다양한 DNA 변형 단백질을 시험하는데 초점이 맞추어졌다(Box 1). 그러나 이들 제안된 후보들은 아주 특별한 생물학적 환경으로
종종 제한되었고, 많은 경우에서 활성화는 후속 연구에 의해 입증되지 않았다. 그리하여 이들 연구는 포유류에서 능동 DNA 메틸화를 위한 메커니즘
체계를 밝히지 않았다.
그러나 2009년에 큰 발견이 있었는데, 인간 유전체에서 5mC의 산화 형태인 5hmC가 포유류 유전체에서
분명하게 설명되고 5mC를 5hmC로 변환하는 DNA 이산화효소(dioxygenases) TET군(family)가 알려졌다. 연속적인 연구는
TET 단백질이 5hmC를 5-formylcytosine (5-fC) 및 5-carboxycytosine (5-caC)으로 바꾼다는 사실을
밝혀내었다. 이와 같은 연속적인 발견은 능동 DNA 탈메틸화의 분자 기작을 이해하기 위한 생화학적인 기초를 세우게 하였다.
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
-
이전글
- BCL-2 family에 의한 세포사멸의 조절: 생리와 병리에 대한 영향
-
다음글
- Using Circulating Biomarkers for Cancer Diagnostics Development
지식
동향


 pdf_0002315.pdf
pdf_0002315.pdf