기술동향
시스템생물학의 합성생물학 적용 연구동향
- 등록일2015-09-15
- 조회수14286
- 분류기술동향
-
자료발간일
2015-09-10
-
출처
지능형 바이오시스템 설계및합성 연구단
- 원문링크
-
키워드
#시스템생물학#합성생물학#세포공장#유전체#바이오시스템
- 첨부파일
출처 : 지능형 바이오시스템 설계및합성 연구단
시스템생물학의 합성생물학 적용 연구동향
- 세포공장 유전체 재구성 연구를 중심으로 -

연세대학교 시스템생물학과 김지현 교수, 양희진 연구원
1. 개요
가. 시스템생물학 및 합성생물학의 정의
(1) 시스템생물학(systems biology)
시스템생물학은 유전체(genome), 전사체(tranome), 단백질체(proteome), 대사체(metabolome) 등의 오믹스 데이터를 기반으로 하여 세포 내 구성성분 간의 상호작용을 규명함으로써 생명현상을 개별 구성 성분이 아닌 전체 시스템 차원에서 통합적으로 이해하려는 학문을 의미한다(그림 1).
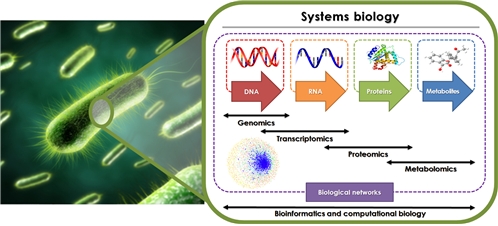
<그림 1> 시스템생물학의 개념
(2) 합성생물학(synthetic biology)
합성생물학은 DNA 염기서열 분석 및 합성, 시스템생물학, 유전공학 기술 등을 융합하여 생물체 본연의 특성으로부터 새로운 생물학적 부품(part), 장치(device), 회로(circuit) 또는 모듈(module)을 디자인하여 새로운 생물학적 기능을 부여하거나 유용 산물의 생산을 목적으로 기존에 존재하는 자연 상태의 생물학적 시스템을 개량 혹은 재설계(redesign)하여 인공적인 바이오시스템을 제작 및 합성하는 학문을 총칭한다(그림 2).
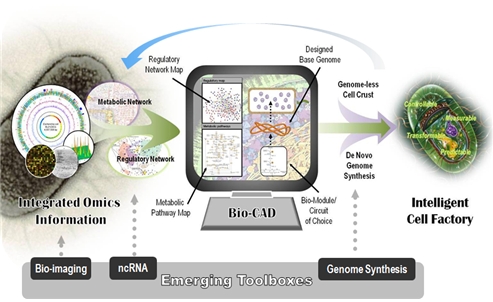
<그림 2> 합성생물학 및 응용 개념도
(3) 시스템생물학과 합성생물학의 융합
시스템생물학에서 밝혀진 생명체의 구성 원리와 일련의 유전자 조합을 포함하는 조절 네트워크는 합성생물학에서 인공 생물체 합성에 필요한 기본 생물학적 부품(bio-bricks) 혹은 유전자회로(circuit) 구성에 적용된다. 바이오 부품, 장치, 시스템을 설계 및 제작하기 위해서는 이들의 구현을 가능하게 하는 기반 기술인 유전체 분석 및 통합 오믹스 정보 등의 대용량 바이오정보 처리기술, 인실리코 시뮬레이션(in silico simulation) 및 Bio-CAD 기술 등의 합성생물학 파운드리(foundry) 기술의 도입이 요구된다[1]. 이와 같이 시스템 및 합성생물학의 융합을 통해 생체 시스템의 복잡성(complexity)을 수학적 모델링과 시뮬레이션으로 증명하고, 나아가 단일 생명체의 생체물질 및 대사 네트워크를 인위적으로 제어할 경우 나타날 표현형 결과를 예측 가능하게 한다(그림 3). 또한 재설계 된 바이오 시스템의 실험적 증명을 통해 인공 생명체의 구축 및 최적화의 가능성을 제시한다. 이는 자연과학적 생명현상의 이해와 공학적인 개념의 융합을 통해 생물체가 갖는 생물학적 한계를 극복하기 위한 실마리를 제공할 것이라 예상된다.
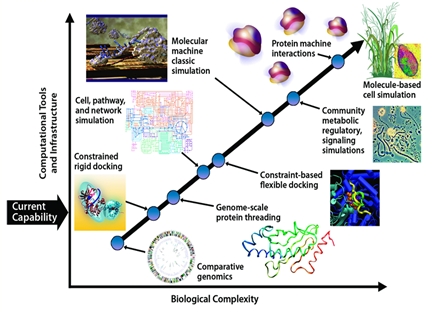
<그림 3> 바이오시스템의
복잡성과 합성생물학 파운드리 기술
(출처: 미국 에너지부, U.S. Department of Energy)
나. 시스템생물학 기반의 합성생물학 연구 활용 방안
시스템생물학 기반의 연구는 유전체 및 오믹스 데이터를 기반으로 한 통합된 시스템 구축을 목표로 한다. 특히 생장 및 유용물질 생산 등에 관련된 다양한 생체기능 유전자의 발굴과 이 유전자들의 인실리코 네트워크 분석은 생체 시스템의 재설계 및 신규 생체모듈의 도입 여부를 결정할 수 있으며, 고부가 가치 산물을 생산하는 세포공장(cell factory) 구축의 가능성을 제시한다(그림 4 및 표 1).
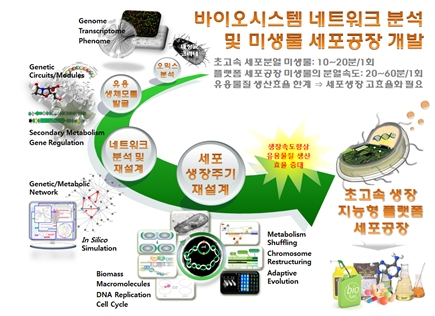
<그림 4> 시스템 및 합성생물학 기반의 미생물 세포공장 연구개발 사례
<표 1> 시스템생물학 기반의 미생물 세포공장 개량 연구개발 사례

이와 더불어 올리고 DNA 합성 기술과 차세대 시퀀싱(NGS, next-generation sequencing)기술의 융합을 통하여 초병렬적으로 유전자 및 유전체를 합성하고, 이 합성 산물을 유전체상에 도입하는 염색체공학(chromosome engineering) 기반의 합성생물학 연구는 미생물 세포공장(microbial cell factory)을 이용한 바이오화합물 생산 및 개발 연구에 직접 연계 될 수 있다(표 2).
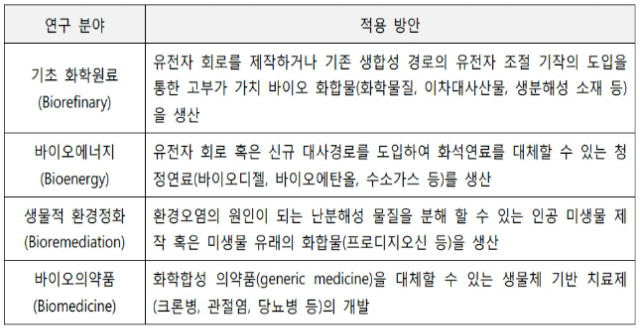
<표 2> 시스템생물학과 합성생물학의 적용 분야
2. 주요 연구동향
가. 미생물 유전체 재설계 및 인공 합성
박테리아의 염색체의 재설계 연구는 유전체 위치 특이적 재조합기술(site-specific recombination)을 기반으로 유전자의 제거 혹은 삽입에 따른 표현형 차이를 분석하는 연구들이 주로 수행되고 있다. 대표적인 예로는 염색체 내 일부분이 제거된 최소 유전체(minimal genome)를 갖는 맞춤형 대장균 균주가 개발된 연구 사례가 있다. 또한 염색체 구조 변경을 위한 연구로는 박테리오파지(bacteriophage N1)와 protelomerase를 이용하여 박테리아가 갖는 원형 염색체를 두 개의 선형 염색체로 분리하는 선행 연구가 있었고, 박테리아의 세포 주기 조절과 DNA복제개시부위(origin of replication)의 추가 삽입 혹은 위치 이동에 따른 세포의 DNA복제시스템 차이를 시간차 현미경(time-lapse micros)을 통해 이미징(imaging) 분석을 시도했던 연구도 수행된 바 있다[6]. 이외에도 대장균의 DNA복제기작에 관한 연구는 DNA복제개시부위와 DNA복제개시 단백질(DnaA)에 의존 및 비의존적인 복제개시기작이 존재함을 규명하였다. 대장균의 DNA복제개시부위와 복제개시단백질을 발현하는 유전자(dnaA)의 결실을 유도하였을 경우에는 세포의 생존력(cell viability)이 현저히 감소되는 경향을 보이는 반면, DNA복제종결자 단백질, RNA 중합효소, 전위효소를 발현하는 유전자들(tus, rpoB, recG)의 결실을 유도하였을 경우에는 DNA복제개시부위가 제거된 염색체에 안정DNA복제(stable DNA replication)이 유도되어 생존을 가능하게 한다는 연구결과가 보고되었다. 대장균의 복제기작에 대한 연구와 반대로 여러 개의 복제개시부위를 갖는 고세균(Haloferax volcanii)의 모든 복제개시부위를 제거하였을 경우에 생장속도가 7.5%가 향상되는 연구결과도 보고된 바가 있다.
유전체 인공합성 연구는 화학적으로 합성된 올리고 핵산을 생체 외(in vitro) 혹은 생체 내 재조합(in vivo recombination)을 통하여 인공적으로 합성하는 연구들이 진행되고 있다. 생체 외 재조합을 통하여 최초로 미생물 유전체(Mycoplasma genitalium, 583 kb)를 인공적으로 합성한 연구사례는 인공 생명체에 대한 관심을 불러 일으켰다[9]. 이 후 올리고 핵산 합성 기술의 진보와 함께 박테리아의 염색체를 효모(Saccharomyces cerevisiae)로부터 인공적으로 합성하여 약 1.1 Mbp 크기의 미생물(Mycoplasma mycoides) 유전체를 성공적으로 합성한 연구 사례가 보고되었다[10]. 이와 같이 미생물유전체 연구는 합성생물학 기반의 다양한 융합 기술의 도입과 생명현상 규명을 바탕으로 기초과학 분야의 발전에 기여하고 있다.
나. 오믹스 데이터 기반의 미생물 세포공장 개발
오믹스 데이터의 활용은 산업, 의약적 고부가 가치 산물에 관여하는 유전자 모듈 발굴 및 선별을 가능하게 한다. 이는 모델 플랫폼 세포(chassis) 또는 고효율 맞춤형 세포공장(smart designer cell factory)의 다양한 생물학적 기작들(유전자 발현, 단백질 형성, 대사 물질의 흐름 및 조절 기작 등)을 수식적으로 표현하고 모델화를 가능하게 한다. 실제 연구사례로는 유전체 정보와 유전체 발현정보를 기반으로 하여 대사 네트워크 모델에 조절 네트워크를 확률적으로 통합하는 모델을 개발함으로써, 대사흐름분석을 보다 정확히 할 수 있는 기술을 제시된 바 있다. 뿐만 아니라, 인위적으로 세포 내 대사회로를 수정하기 위해 특정 유전자를 제거하거나 새로운 유전자를 도입하였을 때의 생체 내 변화를 예측할 수 있고 최적화된 배양환경을 제시할 수 있는 장점을 가지고 있다. 이는 대사 네트워크의 화학양론(stoichiometry) 정보를 바탕으로 하여 그 내부에 존재하는 다양한 최적화된 루트들을 추적할 수 있는 방법을 개발한 연구사례에 부합한다.
유전자회로 및 대사회로개발은 인실리코 예측을 통하여 실험적 증명에 기여할 수 있다. 이는 수학 및 통계학적 모델을 근거로 예측된 조건들을 균주에 효율적으로 도입하여 유전체 리펙토링(genome refactoring)을 가능하게 한다. 해양미생물인 비브리오 균에서 한천 무수당을 분해할 수 있는 효소들을 발굴하고 대사 경로를 규명하여 대장균에서 바이오 에탄올의 생산이 가능한 새로운 대사기작을 확립한 연구사례와 대장균을 이용한 가솔린 생산을 목적으로 지방산의 길이를 줄여줄 수 있는 효소와 신규 대사경로를 도입하여 탄소수가 12개 이하인 짧은 사슬 알케인(short-chain alkane)을 생산한 연구 사례는 합성생물학을 기반으로 한 대사공학 연구동향을 말해준다. 이는 바이오에너지 생산의 경우에 효모 혹은 조류 대비 미생물이 갖는 한계인 낮은 생산효율을 증대 시킬 수 있는 가능성을 제시한 연구사례라 볼 수 있다. 따라서 현재까지 수행된 미생물 세포공장 개발 연구는 시스템생물학 및 합성생물학 기반으로 산업적 응용가능성을 확장시키기 위한 연구들이 진행되고 있음을 알 수 있다.
3. 전망 및
정책제언
시스템 및 합성생물학의 융합은 그동안 단편적으로 이해되어져 왔던 미생물의 생장 메커니즘에 대한 이해를 하나의 통합된 시스템의 차원으로 높일 수 있게 될 것으로 기대된다. 또한 복잡한 생명현상을 공학적 관점에서 재해석하여 생명공학의 활용성을 극대화하고자 하는 시도는 바이오시스템의 전반적인 이해 상승 및 유전체 재설계 기술을 발전을 이룩할 것으로 예측한다. 이는 유전체전체에 걸쳐 수백 ~ 수천 개의 목표 위치(target loci)를 동시에 엔지니어링하고 차세대 시퀀싱을 통하여 분석 할 수 있는 기술의 진보를 의미하며 향후 대사경로, 생합성 경로, 유전자 및 단백질 기능, 유전자 네트워크 등에 관한 연구에 유용하게 사용될 수 있으리라 예상한다. 경제적 측면에서 시스템 및 합성생물학 기반의 미생물 세포공장 개발 연구는 생물 산업 전반에 걸쳐 수율 및 생산성을 극대화할 수 있는 가능성을 열어주게 될 것이고, 특히 석유자원의 고갈을 대체할 바이오연료 산업에 활용 가능성이 높을 것으로 판단된다. 바이오연료의 생산에서 미생물이 갖는 한계점을 탈피하고, 조류 혹은 효모로부터 생산되는 바이오연료 효율 대비 높은 수율의 바이오연료 생산에 기여할 수 있을 것이다. 그러나 기술 집약적인 합성생물학의 발전과 인공 합성 생명체의 출현은 유전자변형생물체(LMO, living modified organism)의 잠재적인 위험성을 갖고 있다. 그러므로 생물안전성(biosafety), 생물안보(biosecurity) 그리고 생명윤리(bioethics)에 대한 올바른 법적 규제와 연구자의 인식이 필요할 것으로 사료된다.
< 참고문헌
>
1. U.S. DOE. Biological and
Environmental Research Information System, Oak Ridge National Laboratory. GTL
Roadmap. 2005. p. 89.
2. Datsenko KA, Wanner BL. One-step inactivation of
chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad
Sci USA. 2000. 97:6640-6645.
3. Yu BJ, Kang KH, Lee JH, Sung BH, Kim MS, Kim
SC. Rapid and efficient construction of markerless deletions in the Escherichia
coli genome. Nucleic Acids Res. 2008. 36:e84.
4. Yu BJ, Sung BH, Koob MD, Lee
CH, Lee JH, Lee WS, Kim MS, Kim SC. Minimization of the Escherichia coli genome
using a Tn5-targeted Cre/loxP excision system. Nat Biotechnol. 2002.
20:1018-1023.
5. Liang X, Baek CH, Katzen F. Escherichia coli with two linear
chromosomes. ACS Synth Biol. 2013. 2:734-740.
6. Wang X, Lesterlin C,
Reyes-Lamothe R, Ball G, Sherratt DJ. Replication and segregation of an
Escherichia coli chromosome with two replication origins. Proc Natl Acad Sci
USA. 2011. 26:243-250.
7. Rudolph CJ, Upton AL, Stockum A, Nieduszynski CA,
Lloyd RG. Avoiding chromosome pathology when replication forks collide. Nature.
2013. 500:608-611.
8. Hawkins M, Malla S, Blythe MJ, Nieduszynski CA, Allers
T. Accelerated growth in the absence of DNA replication origins. Nature. 2013
503:544-547.
9. Gibson DG, Benders GA, Andrews-Pfannkoch C, Denisova EA,
Baden-Tillson H, Zaveri J, Stockwell TB, Brownley A, Thomas DW, Algire MA,
Merryman C, Young L, Noskov VN, Glass JI, Venter JC, Hutchison III CA, Smith HO.
Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium
genome. Science. 2008. 319:1215-1220.
10. Benders GA, Noskov VN, Denisova
EA, Lartigue C, Gibson DG, Assad-Garcia N, Chuang RY, Carrera W, Moodie M,
Algire MA, Phan Q, Alperovich N, Vashee S, Merryman C, Venter JC, Smith HO,
Glass JI, Hutchison III CA. Cloning whole bacterial genomes in yeast. Nucleic
Acids Res. 2010. 38:2558-2569.
11. Chandrasekaran S, Price ND. Probabilistic
integrative modeling of genome-scale metabolic and regulatory networks in
Escherichia coli and Mycobacterium tuberculosis. Proc Natl Acad Sci USA. 2013.
107:17845-17850.
12. Pey J, Prada J, Beasley JE, Planes FJ. Path finding
methods accounting for stoichiometry in metabolic networks. Genome Biol. 2011.
12:R49.
13. Yun EJ, Lee S, Kim HT, Pelton JG, Kim S, Ko HJ, Choi IG, Kim KH.
The novel catabolic pathway of 3,6-anhydro-L-galactose, the main component of
red macroalgae, in a marine bacterium. Environ Microbiol. 2015.
17:1677-1688.
14. Choi YJ, Lee SY. Microbial production of short-chain
alkanes. Nature. 2013. 502:571?574.
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
지식
- BioINpro [국가 바이오 경쟁력] 국가 바이오 경쟁력 평가: 합성생물학(과학기술)과 바이오파운드리(인프라)를 중심으로 2025-04-14
- BioINpro [바이오 융합 소재·제조] 미생물-전극 간 전자 흐름의 재설계: 합성생물학과 바이오인터페이스 기술의 융합 2025-03-31
- BioINwatch 신기술(합성생물학, 유전자편집기술 등) 발전에 따른 유전자변형미생물(GMM)의 합리적 규제 방안 2025-03-27
- BioINregulation 바이오 분야 비용편익분석: '합성생물학 개발·실험 규제완화’를 중심으로 2024-10-07
- BioINwatch 합성생물학과 생체전자공학이 융합한 Living drug 2024-07-09
동향


 시스템생물학의 합성생물학 적용 연구동향_김지현 교수.pdf
시스템생물학의 합성생물학 적용 연구동향_김지현 교수.pdf