기술동향
Let-7 miRNA biogenesis
- 등록일2016-04-12
- 조회수6959
- 분류기술동향
-
자료발간일
2016-03-31
-
출처
지능형 바이오시스템 설계 및 합성연구단
- 원문링크
-
키워드
#유전자 발현 조절#유전자 발현#줄기세포 조절#miRNA#Let-7
- 첨부파일
출처 : 지능형 바이오시스템 설계 및 합성연구단
Let-7 miRNA biogenesis
저자 : 한국과학기술원 이대엽 교수
1. 개요
MicroRNA (이하 miRNA)는 약 22 nucleotide 길이의 noncoding RNA로서 다양한 진핵 생물에서 다양한 유전자의 발현을 조절하는 것으로 알려져 있다. MiRNA는 mRNA의 3’UTR에 있는 상보적인 서열과의 결합을 통해 표적 mRNA의 전사 방해, 단백질로의 번역 억제, mRNA의 분해 등의 여러 가지 방법으로 표적 유전자의 발현을 억제한다. 대표적인 miRNA인 let-7 miRNA는 C. elegance에서 처음으로 발견된 miRNA 중 하나로서 C. elegans (예쁜꼬마선충)의 발달 시점을 조절하는 역할을 하며 C. elegans 뿐만 아니라 초파리, 포유류에도 존재하고 그 역할이 진화적으로 잘 보존되어 있는 것으로 알려져 있다. Let-7 의 발현은 포유류의 발달 과정에서 증가하는 것으로 알려져 있으며 다른 여러 생물학적 과정에 관여한다는 사실도 알려져 있다. 또한 정상적인 발달과정뿐만 아니라 특정 암 세포에서도 let-7 의 발현이 증가한다는 사실로 미루어보아 let-7의 발현 조절이 매우 중요하다고 유추할 수 있으며 이 조절과정에 LIN28이 매우 중요한 역할을 하는 것으로 알려져 있다.
유도만능줄기세포의 주요 인자 중 하나인 LIN28은 인간, 쥐와 같은 포유류에서 LIN28A와 LIN28B의 두 가지 형태가 존재하며 줄기세포의 자기재생과 전분화능 그리고 분화와 발달과정을 조절하는 중요하며, 세포성장 조절에 결정적인 c-Myc 유전자 발현을 조절하여 줄기세포뿐만 아니라 암세포의 조절에도 관여한다. Let-7 은 여러 단계의 가공 과정을 통해 성숙 let-7 miRNA를 생성하는데, LIN28은 여러 단계의 let-7 성숙과정을 조절함으로써 세포성장을 조절하는 것으로 알려져 있다. 이런 사실을 통해 LIN28에 의한 let-7의 성숙 저해 조절기전에 대한 연구는 배아줄기세포의 전분화능을 유지뿐만 아니라 항암치료 등의 임상 단계까지 폭 넓게 응용할 수 있을 것으로 기대된다.
2. Let-7 miRNA의 성숙과정
Let-7 miRNA는 일반적인 miRNA와 유사하게 두 종류의 가공효소인 Drosha와 Dicer에 의한 성숙 과정을 거친다. Let-7 은 mRNA와 같이 핵 내에서 RNA 중합효소 II에 의해 전사되어 5’ capping과 3’ poly(A) tail을 가지며 (Bracht et al., 2004), 전사체 내부의 상보적 결합을 통해 hairpin stem-loop의 구조적 특징을 가진 primary let-7 (이하 pri-let-7)을 형성한다. 이 pri-let-7 은 Drosha와 DGCR8으로 이루어진 RNA분해효소 복합체에 의해 약 60~70 nucleotide 크기의 precursor let-7 (이하 pre-let-7)을 형성하는데 pri-let-7 과 마찬가지로 stem-loop이라는 구조적 특징을 가지고 있으며 3’ 말단 1 혹은 2 nucleotide 길이의 overhang을 지닌다 (Lee et al., 2003; Denli et al., 2004; Gregory et al., 2004; Landthaler et al., 2004).
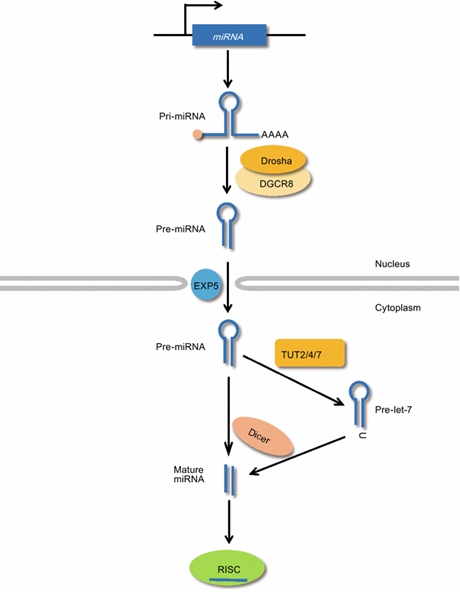
Figure 1. Let-7 miRNA 성숙과정 (Lee et al., 2015)
Drosha에 의해 가공된 pre-let-7은 Ran-GTP 의존적 수송체인 exportin 5에 의해 핵에서 세포질로 이동되며 이 곳에서 두 번째 가공효소인 Dicer에 의해 추가적인 성숙 과정을 거치면서 약 22 nucleotide 길이의 성숙 let-7 을 형성한다 (Bernstein et al., 2001; Grishok et al., 2001; Hutvagner et al., 2001; Ketting et al., 2001; Knight and Bass, 2001). Dicer는 3’ 말단에 2 nucleotide overhang을 가진 precursor를 선호하기 때문에 1 nucleotide overhang을 가진 일부 pre-let-7의 경우 추가적인 과정이 더 필요하다. 이 경우 3’ 말단이 텃효소 (TUTase, Terminal Uridylyl Transferase)에 의해 모노유리딜화 됨으로써 Dicer가 선호하는 2 nucleotide길이의 3’ 말단 overhang을 만들어 Dicer에 의한 성숙과정을 돕는다 (Heo et al., 2012). 이후 생성된 성숙 let-7 은 Argonaute와 함께 표적 mRNA과의 상보적 결합함으로써 표적 mRNA의 발현을 저해하는 역할을 수행한다.
3. LIN28에 의한 let-7 miRNA 성숙의 조절
지금까지 연구 결과에 따르면 let-7 miRNA의 성숙과정의 조절은 크게 두 종류로 나눌 수 있는데 이 두 과정 모두 LIN28에 의해 이루어지는 것으로 알려져 있다. 먼저 LIN28A를 HEK293T 세포주에서 과발현 할 경우 pre-let-7 의 3’ 말단이 텃효소에 의해 올리고유리딜화 된다는 사실이 알려졌으며 (Heo et al., 2008; Hagan et al., 2009; Heo et al., 2009), 올리고유리딜화 된 pre-let-7 은 Dicer에 의한 성숙이 저해되는 대신에 오히려 DIS3L2에 의해 분해됨으로써 성숙 let-7 이 생성되지 못하도록 한다 (Chang et al., 2013; Malecki et al., 2013; Ustianenko et al., 2013). 반면 LIN28A가 없는 상황에서는 pre-let-7 이 모노유리딜화 되면서 Dicer에 의해 성숙 let-7 을 만들어낸다. 이런 현상은 HEK293T 세포주뿐만 아니라 배아줄기세포와 특정 암세포에서도 일어난다는 사실이 알려졌다.
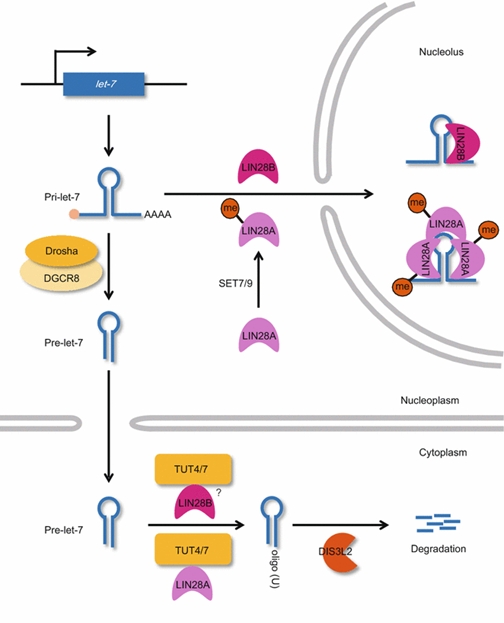
Figure 2. Let-7 miRNA 성숙과정의 조절 (Lee et al., 2015)
LIN28B는 LIN28A와 다르게 텃효소-비의존적으로 let-7의 성숙을 저해한다 (Piskounova et al., 2011). 핵에 주로 존재하는 LIN28B는 LIN28A와 같이 RNA와 직접 결합할 수 있는 도메인을 가지고 있지만, LIN28A와 달리 핵 내 세포 소기관인 인으로 이동할 수 있는 NoLS (Nucleolar-Localization Signal)을 가지고 있다. 이런 LIN28B의 특징으로 인해, 핵 내에서 pri-let-7 과 직접 결합한 LIN28B는 Dicer가 없는 인으로 pri-let-7 과 함께 이동함으로써 Dicer에 의한 추가적인 let-7의 성숙과정이 일어날 수 없게 함으로써 텃효소-의존적으로 let-7의 성숙을 막는 LIN28A과는 다른 방법으로 성숙 let-7 생성을 억제한다.
최근 연구에 의하면 LIN28A도 LIN28B와 유사하게 텃효소-비의존적으로 성숙 let-7의 생성을 저해할 수 있다는 사실이 밝혀졌다 (Kim et al., 2014). LIN28A는 주로 세포질에 존재하지만 일부 핵 내에도 존재하는데, 핵 내에서 히스톤 모노메틸화 효소인 SET7/9과의 결합을 통해 135번째 라이신에 특이적으로 모노메틸화 된다. 이 모노메틸화된 핵 내 LIN28A는 에서 pri-let-7 과 다중 복합체 형성을 촉진하고 LIN28B와 유사하게 인으로 이동함으로써 pri-let-7 이 Dicer에 의해 성숙되는 것을 저해한다. 또한 LIN28B가 발현되는 특정 암세포의 경우 LIN28B 역시 LIN28A와 유사하게 DIS3L2와 상호작용하는 사실이 추가적으로 밝혀졌다 (Suzuki et al., 2015). 이런 최근의 연구결과로 미루어보아 LIN28A와 LIN28B의 let-7 성숙 저해 과정이 어느 정도 공통적인 기전을 통해서 이루어질 것이라 예상되며 이에 대한 후속연구가 필요하다.
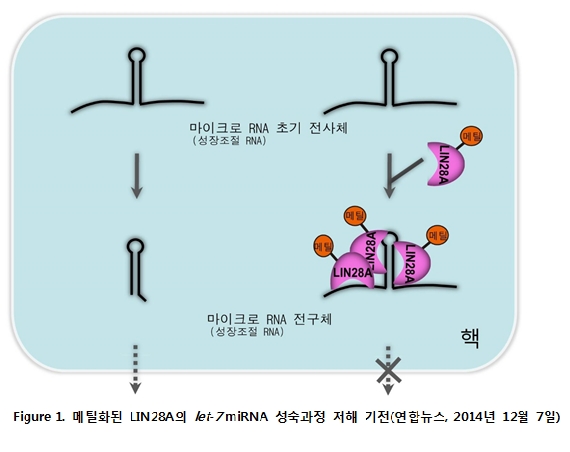
4. 고찰
Let-7 miRNA는 줄기세포의 분화를 촉진하며 세포성장을 조절하는 역할을 한다. 따라서 let-7 miRNA의 조절과정에 대한 연구는 배아줄기세포의 전분화능의 조절을 이해하는 데 매우 중요하다. 뿐만 아니라 LIN28A이 유도만능줄기세포 제작에 필요한 주요 요소임을 감안할 때, let-7 miRNA의 성숙과정에 대한 연구를 통해 현재 매우 낮은 유도만능줄기세포 제작의 효율을 증대시킬 수 있는 새로운 방법론을 제시할 수 있으리라 예상된다. 또한 몇몇 암세포에서 let-7 의 발현이 정상세포에 비해 증가한다는 사실과 LIN28 역시 암세포와 밀접한 관련이 있다는 사실을 종합해보면 let-7 과 LIN28에 대한 연구는 항암물질의 새로운 표적의 발굴이나 항암물질의 스크리닝 등 임상분야에도 다양하게 응용될 수 있으리라 기대된다.
5. 참고문헌
1. Bracht J, Hunter S, Eachus R, Weeks P, Pasquinelli AE (2004) Trans-splicing and polyadenylation of let-7 microRNA primary trans. RNA 10:1586?1594
2. Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Radmark O, Kim S et al (2003) The nuclear RNase III Drosha initiates microRNA processing. Nature 425:415?419
3. Denli AM, Tops BB, Plasterk RH, Ketting RF, Hannon GJ (2004) Processing of primary microRNAs by the microprocessor complex. Nature 432:231?235
4. Gregory RI, Yan KP, Amuthan G, Chendrimada T, Doratotaj B, Cooch N, Shiekhattar R (2004) The microprocessor complex mediates the genesis of microRNAs. Nature 432:235?240
5. Landthaler M, Yalcin A, Tuschl T (2004) The human DiGeorge syndrome critical region gene 8 and Its D. melanogaster homolog are required for miRNA biogenesis. Curr Biol 14:2162?2167
6. Bernstein E, Caudy AA, Hammond SM, Hannon GJ (2001) Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature 409:363?366
7. Grishok A, Pasquinelli AE, Conte D, Li N, Parrish S, Ha I, Baillie DL, Fire A, Ruvkun G, Mello CC (2001) Genes and mechanisms related to RNA interference regulate expression of the small temporal RNAs that control C. elegans developmental timing. Cell 106:23?34
8. Hutvagner G, McLachlan J, Pasquinelli AE, Balint E, Tuschl T, Zamore PD (2001) A cellular function for the RNA-interference enzyme Dicer in the maturation of the let-7 small temporal RNA. Science 293:834?838
9. Ketting RF, Fischer SE, Bernstein E, Sijen T, Hannon GJ, Plasterk RH (2001) Dicer functions in RNA interference and in synthesis of small RNA involved in developmental timing in C. elegans. Genes Dev 15:2654?2659
10. Knight SW, Bass BL (2001) A role for the RNase III enzyme DCR-1 in RNA interference and germ line development in Caenorhabditis elegans. Science 293:2269?2271
11. Heo I, Ha M, Lim J, Yoon MJ, Park JE, Kwon SC, Chang H, Kim VN (2012) Mono-uridylation of pre-microRNA as a key step in the biogenesis of group II let-7 microRNAs. Cell 151:521?532
12. Lee H, Han S, Kwon CS, Lee D (2015) Biogenesis and regulation of the let-7 miRNAs and their functional implications. Protein Cell
13. Heo I, Joo C, Cho J, Ha M, Han J, Kim VN (2008) Lin28 mediates the terminal uridylation of let-7 precursor MicroRNA. Mol Cell 32:276?284
14. Hagan JP, Piskounova E, Gregory RI (2009) Lin28 recruits the TUTase Zcchc11 to inhibit let-7 maturation in mouse embryonic stem cells. Nat Struct Mol Biol 16:1021?1025
15. Heo I, Joo C, Kim YK, Ha M, Yoon MJ, Cho J, Yeom KH, Han J, Kim VN (2009) TUT4 in concert with Lin28 suppresses microRNA biogenesis through pre-microRNA uridylation. Cell 138:696?708
16. Chang HM, Triboulet R, Thornton JE, Gregory RI (2013) A role for the Perlman syndrome exonuclease Dis3l2 in the Lin28-let-7 pathway. Nature 497:244?248
17. Malecki M, Viegas SC, Carneiro T, Golik P, Dressaire C, Ferreira MG, Arraiano CM (2013) The exoribonuclease Dis3L2 defines a novel eukaryotic RNA degradation pathway. EMBO J 32:1842?1854
18. Ustianenko D, Hrossova D, Potesil D, Chalupnikova K, Hrazdilova K, Pachernik J, Cetkovska K, Uldrijan S, Zdrahal Z, Vanacova S (2013) Mammalian DIS3L2 exoribonuclease targets the uridylated precursors of let-7 miRNAs. RNA 19:1632?1638
19. Piskounova E, Polytarchou C, Thornton JE, LaPierre RJ, Pothoulakis C, Hagan JP, Iliopoulos D, Gregory RI (2011) Lin28A and Lin28B inhibit let-7 microRNA biogenesis by distinct mechanisms. Cell 147:1066?1079
20. Kim SK, Lee H, Han K, Kim SC, Choi Y, Park SW, Bak G, Lee Y, Choi JK, Kim TK et al (2014) SET7/9 methylation of the pluripotency factor LIN28A is a nucleolar localization mechanism that blocks let-7 biogenesis in human ESCs. Cell Stem Cell 15:735?749
21. Suzuki HI, Katsura A, Miyazono K (2015) A role of uridylation pathway for blockade of let-7 microRNA biogenesis by Lin28B. Cancer Sci. doi:10.1111/cas.12721
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.


 Let-7 miRNA biogenesis_이대엽 교수.pdf
Let-7 miRNA biogenesis_이대엽 교수.pdf