기술동향
차세대 씨퀀싱 기술의 10년 [1]
- 등록일2017-07-20
- 조회수7944
- 분류기술동향
-
자료발간일
2017-07-11
-
출처
생물학연구정보센터(BRIC)
- 원문링크
-
키워드
#genome#genomics#DNA sequencing#Next Generation Sequencing
- 첨부파일
출처 : 생물학연구정보센터(BRIC)
차세대 씨퀀싱 기술의 10년 [1]
Laura HA / Pennsylvania State University
[목 차]
1. 서론
2. 짧은 판독 길이(short-read)를 가진 차세대시퀀싱 기술들
2.1 결찰을 통한 시퀀싱(Sequencing by Ligation, SBL)
2.2 결합을 통한 시퀀싱(Sequencing by Synthesis, SBS)
2.3 짧은 판독 길이(short-read length)를 가진 차세대 시퀀싱 기술들의 비교
3. 긴 판독 길이(long-read length)를 가진 차세대시퀀싱 기술들
3.1. 단일분자 실시간 시퀀싱(Single molecule real-time sequencing, SMART)
3.2. 짧은 판독 길이 시퀀싱을 응용한 합성 고판독시퀀싱(Synthetic long reads)
4. 결론
[요약문]
2003년 휴먼 게놈 프로젝트가 완성된 이래 다양한 씨퀀싱 기술과 가격의 비약적인 발전이 이루어져 왔다. 이렇게 생산된 방대한 양의 데이터는 유전자의 엄청난 복잡성을 이해하는데 크게 기어했을 뿐만 아니라, 상대적으로 길이가 긴 DNA 가닥을 시퀀싱 하는데 가능해짐에 따라, 깊이 있는 유전자 연구가 가능하게 되었다. 하지만, 지난 10년간 이루어진 비약적인 발전에도 불구하고, 높은 착오율과 낮은 판독 길이(read length)는 아직까지도 문제점으로 지적되고 있다. 이 논문에서는 차세대 시퀀싱 기술이 사용하고 있는 다양한 접근 방법과 차세대 시퀀싱 기술 분야의 비약적인 발전이 어떻게 유전학 연구에 응용되고 있는지 알아보고자 한다.
1. 서론
휴먼 게놈프로젝트 이후에, 복잡한 생물학적 궁금증을 해소할 수 있는 개선된 시퀀싱 기술의 필요성이 대두되어 왔으나 시퀀싱의 비싼 가격과 처리 양의 한계는 기술의 실용화에 크나큰 장벽으로 평가되었다. 지난 10년간 이루어진 비약적인 차세대 시퀀싱 기술의 발전은 단일 샘플에서 읽을 수 있는 DNA의 기본 쌍의 수(read length)의 증가뿐 아니라 시퀀싱에 들어가는 비용 또한 크게 낮추어 임상실험 도구로서의 가능성을 시사하였다. 하지만, 이런 비약적인 발전에도 불구하고 전통적인 생거 시퀀싱(Sanger sequencing)에 비하여 현재의 차세대 시퀀싱 기술은 착오율이 0.1-15%로 높을 뿐 아니라 판독 길이(read length)는 35-700 bp로 더 낮아 아직까지도 성공적 실용화 단계에 어려움을 가지고 있는 실정이다. 또한, 차세대 시퀀싱 기술 중 비교적 긴 판독 길이(read length)를 가진 방법들조차, 비싼 가격과 낮은 처리 양으로 효율성 면에서 많은 인기를 누리지 못하고 있는 현실이다.
이 리뷰에서는 차세대 시퀀싱 기술이 사용하고 있는 다양한 접근 방법과 차세대 시퀀싱 기술 분야의 비약적인 발전이 어떻게 유전학 연구에 응용되고 있는지 알아보려고 한다.
2. 짧은 판독 길이(short read length)를 가진 차세대 시퀀싱 기술들
시퀀싱을 위한 준비단계는 크게 DNA를 작은 조각으로 자르는 파쇄와 템플릿의 양을 증가시키는 복제/증폭(clonal amplification)의 두 단계로 나뉜다.
시퀀싱 준비의 첫 단계인, 샘플 DNA의 파쇄의 경우 DNA가 적절한 방법으로 작은 조각들로 나누어진다. 이렇게 만들어진 DNA 조각들은 복제와 증폭(clonal amplification) 단계를 위한 어댑터와 결합된다. DNA에 연결된 어댑터는 보통 고체 표면(solid surface)에 고정된 올리고 핵산염과 상보적 관계에 있기 때문에, DNA가 연결된 어댑터와 올리고 핵산염에 결합이 고체 표면(solid surface)에서 일어나게 된다. 고체 표면(solid surface)에서의 성공적 결합 후, emPCR을 통하여 DNA 템플릿의 복제가 이루어지고 최대 100만개의 복제된 DNA 조각이 고체 표면(solid surface)에 만들어진다. DNA의 복제와 증폭을 위한 다양한 고체 표면(solid surface) 기술이 개발되어 왔는데, 구슬 기반(bead based), 고체 상태(solid state), DNA 나노볼 생성(DNA nanoball generation)이 그 대표적인 예이다.
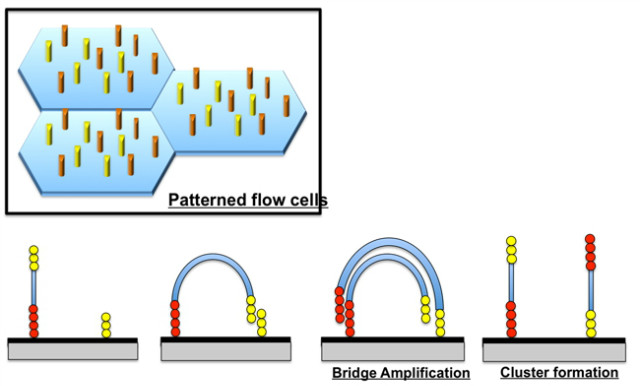
그림 1. 고체 표면(Solid surface) 브릿지 DNA 증폭 방법의 예시
고체 상태 증폭기술(solid state amplification)의 경우는, 정방향 및 역방향 프라이머(forward and reverse prime)가 고체 표면(solid surface)에 공유결합으로 연결되어 있고 이 primer는 외가닥 DNA가 결합할 수 있는 상보적 말단(complementary ends)을 제공한다. 이 방법의 경우, 성공적인 DNA 증폭을 위해서 템플릿의 밀도를 적적히 컨트롤하는 것이 중요한데, 최근의 차세대 시퀀싱 플랫폼은 패터닝이 된 플로우 셀(flow cell)를 이용함으로써 더 많은 DNA 템플릿이 국소적으로 생성되고 좋은 시퀀싱 결과를 내는 게 효율적이다.
현재까지 템플릿 증폭을 액체 안에서 할 수 있는 기술은 매우 제한적으로 발전되어 왔다. 그 중 가장 잘 알려진 기술이 Beijing Genomic Institute (BGI)의 기술인 DNA 나노볼(DNA nanoball)이다. DNA가 반복적인 결합과 원형화(circularization), 그리고 분리를 통하여 4개의 각기 다른 어댑터 층을 가진 템플릿을 만들어 낸다. 이러한 회전 원형 증폭(rolling circle amplification, RCA) 과정을 통하여 2천만 개의 DNA 나노볼(DNA nanoball)이 만들어지며 DNA 증폭이 이루어진다.
이렇게 고체 표면(solid surface)에서 복제되고 증폭된 DNA는 준비과정을 마치고, 시퀀싱 단계에 들어가게 된다. 이 장에서 소개할 짧은 판독 길이(read length)를 가진 시퀀싱의 방법은 크게 결찰을 통한 시퀀싱(sequencing by ligation)과 결합을 통한 시퀀싱(sequence by synthesis)으로 나뉜다. 두 가지 방법을 사용하는 차세대 시퀀싱 기술들에 대해 조금 더 자세히 알아보도록 하자.
2.1 결찰을 통한 시퀀싱(Sequencing by ligation, SBL)
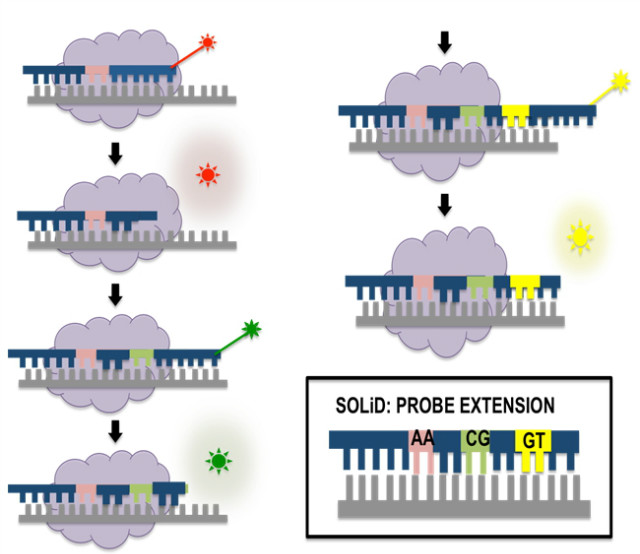
그림 2. 결찰을 통한시퀀싱 기술 예시(SOLiD)
SBL을 사용하는 차세대 시퀀싱 기술들은 형광단에 연결되어 있는 프로브가 먼저 타깃 DNA와 결합한 후 2~10의 뉴클레오티드로 이루어진 중합체인 올리고 핵산염이 결합한다. 이러한 결합은 결찰(ligation)의 시작점이 되어 형광단으로 하여금 특정 스펙트럼의 빛을 투사하도록 한다. SBL에서는 이러한 형광단의 발광을 이미징함으로써 염기의 정체를 식별해 낸다.
SBL 방법을 사용하는 대표적 플랫폼 중 하나는 SOLiD이다. SOLiD 플랫폼은 두 개의 뉴클레오티드 염기를 가진 형광단/프로브를 이용하는데, 각각의 형광 시그널은 다이뉴클레오티드 (dinucleotide)를 나타낸다. 따라서 총 16개의 다이뉴클레오티드(dinucleotide)의 조합이 가능하고 4종류의 형광색으로는 표현이 불가능함으로 각각의 형광 시그널은 여러 가지의 다이뉴클레오티드 (dinucleotide) 조합을 나타내게 된다. 따라서 이미징에서 만들어지는 날것 그대로의 데이터는 추가적인 데이터 처리 과정을 필요로 한다. SOLiD의 경우에 프로브의 결합, 결찰, 이미징, 분열의 반복으로 상보적인 DNA 사슬(complementary strand)의 연장이 일어나는데 여러 번의 사이클을 거치면서 템플릿 내의 모든 뉴클레오티드 염기가 안전하게 시퀀싱될 수 있도록 씽글뉴클레오티드 (single nucleotide)의 오프셋(offset)이 도입된다.
2.2 결합을 통한 시퀀싱(Sequencing by Synthesis, SBS)
SBS의 가장 큰 특징은 SBL과는 달리 폴리메라아제(polymerase)가 이용된다는 점이다. 형광단 시그널은 신장 가닥(elongation strand)의 뉴클레오티드의 결합을 통하여 표출되고 이런 형광의 발광을 모니터 함으로써 시퀀싱이 이루어진다. SBS은 결합 방법에 따라 싸이클릭 가역적 종결(cyclic reversible termination, CRT)과 싱글 뉴크리오티드 첨가(Single nucleotide addition, SNA)로 나뉜다.

그림 3. 결합을 통한 시퀀싱 기술(Illumina)
싸이클릭 가역적 종결(CRT)를 사용하는 가장 대표적 시스템은 Illumina이다. 싸이클릭 가역적 종결(CRT) 방법은 생거 시퀀싱(Sanger sequencing)과 비슷하게 라이보스(ribose)의 3’에 있는 히드록실기(hydroxyl group)가 차단되어 있는 종단 분자(terminator molecules)를 이용한다. 시퀀싱의 시작은 DNA 템플릿이 어댑터와 상보적인 시퀀스를 가진 프라이머와 합체되고, 이 이중 가닥(double strand) DNA에 폴리메라아제가 결합한다. 매 싸이클마다 4개의 형광단으로 표식화된 dNTP 혼합물이 추가되고 각각의 dNTP는 신장 가닥(elongating complementary strand)과 결합하며 형광을 발산한다. dNTP 식별은 두 개 혹은 4개의 레이저 채널을 가진 total internal reflection fluorescence micros를 통하여 달성된다.
싸이클릭 가역적 종결(CRT)를 사용하는 또 다른 시스템은 Qiagen의 Intelligent BioSystems CRT 플랫폼이다. 이 시스템의 특징은 Illumina와 비슷한 접근 방식을 가지나, 샘플 준비부터 분석까지 한 번에 처리되는 올인원 차세대 시퀀싱(all-in-one Next Generation Sequencing) 플랫폼이라는 것이다.
싱글 뉴크리오티드 첨가(SNA)는 싸이클릭 가역적 종결(CRT)와 다르게 4종류의 형광단이 연결된 뉴클레오티드가 한꺼번에 넣어지는 것이 아니라, 순차적으로 추가된다. 처음으로 만들어진 차세대 시퀀싱 기기는싱글 뉴크리오티드 첨가(SNA)의 일종인 454 Pyrosequencing이다. 이 시스템은 템플릿이 연결된 구슬(bead)과 효소 혼합체/칵테일을 PicoTiterPlate에 분배한다. 플레이트 안에서 dNTP와 DNA 가닥의 결합으로 생체발광(bioluminescence)이 일어나고 이 빛을 Charge coupled device (CCD) 카메라를 이용하여 감지해 시퀀싱이 이루어진다.
2.3 짧은 판독 길이(short read length)를 가진 차세대시퀀싱 기술들의 비교
각각의 짧은 판독 길이를 가진 차세대 시퀀싱 플랫폼들은 처리량, 가격, 오류 프로파일, 판독 구조(read structure)에서 차이를 보인다. SOLiD와 Complete Genomics 시스템은 99.99%라는 높은 정확성을 가지고 있으나 민감도(sensitivity)와 특이성(specificity)는 부족함을 가진다. 또한 SOLiD는 최대 판독 길이가 75 bp로 매우 낮기 때문에 genomic assembly와 structural variant detection 응용에 어려움이 있다. Illumina가 상대적으로 짧은 판독 길이를 가진 차세대 시퀀싱 시장을 장악할 수 있었던 이유는 높은 레벨의 호환성(cross platform compatibility)과 플랫폼의 다양성 때문이다. 하지만 Illumina는 CRT 시스템인 만큼 하나의 뉴클레오티드 염기가 반복되는 시퀀스 부분(homopolymer)의 오류에 취약하고 AT와 GC가 밀집된 부분에서 과소 표시(under representation)가 일어나거나 대치 오류(substitution )가 일어나는 문제점을 가지고 있다.
표 1. 짧은 판독 길이를 가진 차세대 시퀀싱 기술들의 비교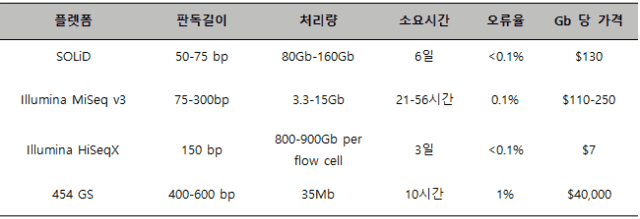
3. 긴 판독 길이(long read length)를 가진 차세대 시퀀싱 기술들
게놈은 길고 반복적인 요소를 가질뿐 아니라 구조적 다양성을 가진 매우 복잡한 구조체이다. 이렇게 복잡하고 긴 구조체는 짧은 판독 길이를 가진 차세대 시퀀싱 테크놀로지로는 풀어내는 것에는 한계가 있다. 따라서 긴 판독 길이를 기반으로 한 시퀀싱 기술은 이런 거대한 구조적 특성을 효과적으로 파헤침으로써 게놈의 성분적 모호함을 해결하는데 도움을 준다. 긴 판독 길이를 사진 차세대 시퀀싱 기술의 경우 특히 트랜스크립톰(Tranomics)의 분석에 매우 유용한 정보를 제공한다. 뿐만 아니라 연구자가 엑손(exon)의 결합이나 유전자 동형 단백질(gene isoform)을 정밀하게 구분할 수 있도록 도움을 준다.
현재까지 두 종류의 긴 판독 길이를 가진 시퀀싱 기술이 존재하는데 단일분자 실시간 시퀀싱(single molecule real-time sequencing)과 짧은 판독 길이 시퀀싱 테크놀로지를 기반으로 합성하여 긴 길이의 DNA를 시퀀싱 해내는 Synthetic long reads 시퀀싱 기술이 그것이다. 두 기술 중 단일분자 실시간 시퀀싱(single molecule real-time sequencing)은 증폭된 DNA 조각의 복제품을 기반으로 시그널을 감지하지 않는다는 점에서 그리고 dNTP 추가에 화학적 사이클링을 사용하지 않는다는 점에서 짧은 판독 길이 시퀀싱 방법과 다르다.
3.1 단일분자 실시간 시퀀싱(Single molecule real-time sequencing, SMART)
현재 가장 널리 이용되는 긴 판독 길이 시퀀싱 플랫폼은 Pacific Bioscience의 단일분자 실시간 시퀀싱(SMART)이다. 이 장비는 수천 개의 피코리터 웰(picoliter well)을 가진 특별한 플로우 셀(flow cell)을 이용하는데 웰의 바닥에 고정된 폴리메라아제가 DNA 가닥을 읽으면서 시퀀싱을 진척시킨다. dNTP가 각각의 단일분자 염기와 결합하게 되면 바닥에 고정된 폴리메라아제가 dNTP에 연결된 형광단을 분리시킨다. 이렇게 분리된 형광단이 내뿜는 형광은 레이저와 카메라 시스템으로 지속적으로 시각화되고 녹음되어 시퀀스가 판독된다. 단일분자에서 나오는 형광의 강도가 매우 약하기 때문에 피코리터 웰에서 고도의 집중된 레이저를 응집하여 형광을 측정하는 Zero Mode Waveguide라는 기술이 이미징에 사용된다.
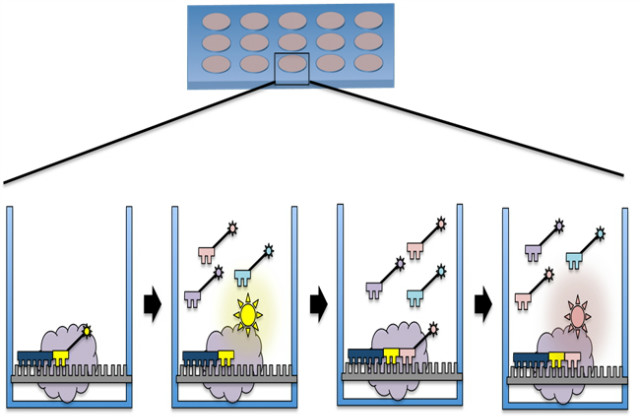
그림 4. 긴 판독 길이를 가진 차세대시퀀싱 기술(SMART)
SMART의 경우 비록 DNA 템플릿이 3 kb보다 길 경우 여러 번 시퀀스 되는데 어려움이 있지만 짧은 DNA 템플렛의 경우 여러 번 시퀀스 되는데 문제가 없고 이런 복수 판독은 공통 배열(consensus sequence)를 만드는데 사용된다.
2014년에 Oxford Nanopore Technology에서 개발된 MinION은 DNA가 단백질 구멍을 통과하게 함으로써 시퀀싱을 시작하는데 단백질에서 일어나는 DNA 이동은 전압의 끊어짐을 일으키고 이것은 단백질 구멍 내의 전류의 변화를 일으킨다. 여기서 일어나는 전극의 변화를 시간에 따라 추적함으로써 시퀀스가 판독된다.
3.2 짧은 판독 길이 시퀀싱을 응용한 합성 고판독 시퀀싱(Synthetic long read sequencing)
합성 고판독(Synthetic long reads) 시퀀싱 기술의 특징은 기존의 짧은 판독 길이 시퀀서에 의해 시퀀스된 DNA 조각(fragment)에 바코딩을 더하는 것이다. 현재 이 기술을 이용하는 기술 중 하나인 Illumina synthetic long read sequencing 플랫폼의 경우, DNA 템플릿이 8-10 kb 정도 길이로 나누어지고, 이렇게 조각난 DNA를 극미량웰(microtitre well)에 1 웰당 3,000개 정도의 템플릿을 가지도록 분배한다. 이 웰 안에서 DNA는 350 bp 정도의 작은 조각으로 다시 조각내어지고 바코딩 된다. 이렇게 만들어진 같은 웰 안의 DNA 조각들은 같은 바코드를 가지게 되고, 풀링 “Pooling”이라는 작업을 통하여 모든 웰 안에 있던 DNA가 모아진 후에 짧은 판독 길이 시퀀싱의 방법으로 시퀀싱 된다.
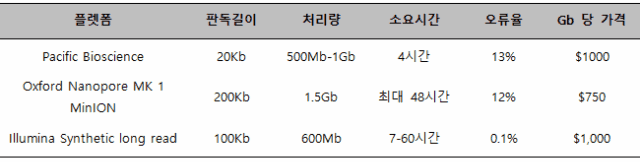
아래의 표는 다양한 긴 판독 길이를 가진 차세대 시퀀싱 플랫폼들은 처리량, 가격, 오류 프로파일, 판독 구조(read structure)에서 차이를 비교한 표이다.
표 2. 긴 판독 길이를 가진 차세대 시퀀싱 기술들의 비교
4. 결론
지난 10년간의 차세대 시퀀싱 기술의 발전은 총 유전체 시퀀싱(whole Genome Sequencing) 분야의 흥행을 이루었을뿐 아니라, 전사체학 연구(Tranomic research) 분야나, 유전체의 규제 메커니즘에 관한 엑솜 시퀀싱과 표적 시퀀싱 분야에서도 눈부신 발전을 이루어냈다. 2012년 Ellis 연구팀에 의하여 연구된 유방암 환자의 방향화효소(aromatase inhibitor) 테라피와 유전자의 관련성에서 보여지듯이, 차세대 시퀀싱 기술의 성장은 많은 연구자들에게 유전체의 심도 있는 연구와 각각의 유전자가 내포하고 있는 생물학적 의미를 이해할 수 있는 기회를 제공해 주었다.
최근의 차세대 시퀀싱 패러다임의 변환은 긴 DNA를 시퀀싱 해내는 능력의 진화이다. 복잡하고 반복되는 부분이 많은 DNA 경우, 그 동안 짧은 판독 길이 시퀀싱 기술로는 시퀀스를 풀어내는데 어려움이 있어왔다. 하지만 최근 연구에서 Chaisson 그룹이 발명한 긴 판독 길이 시퀀싱 기술을 용하여 GrCH37의 매우 복잡한 표준 유전체의 신원을 성공적으로 확인해 냈다.
이번 리뷰에서 우리는 차세대 시퀀싱 기술의 현주소를 살펴보았다. 비록 아직까지는 많은 숙제들이 남아있으나, 방대한 DNA 양의 염기서열 정보를 병렬적으로 처리할 수 있는 차세대 시퀀싱 기술의 능력은 새로운 생명체의 유전정보를 확인하거나 암이나 기타 질병 진단을 위한 용도로써의 그 활용도가 기대된다.
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
지식
동향
- 기술동향 분류학적 난제 해결을 위한 계통유전체학(Phylogenomics) 연구동향 2023-06-13
- 기술동향 PROTACs: Reinvention of an old workhorse-the small molecule drug 2019-12-11
- 기술동향 개인유전체(personal genome)를 활용한 질병 위험 예측의 허와 실 2019-07-03
- 기술동향 Predictive genomics를 통한 종양 임상 표현형을 예측하는 방법론 new 2018-09-18
- 기술동향 전장 유전체(whole-genome sequencing)을 이용한 질병 유전학 연구 동향 2018-05-17


 pdf_0002770.pdf
pdf_0002770.pdf