인터페론 감마에 의한 대식세포의 후성유전체적 조절
강규호 (충북대학교 생명과학부)
서론
세포에서 다양한 외부 자극들에 대한 반응이 일어나는 것은 생명체에서 일어나는 가장 중요한 일이면서 가장 복잡한 과정이기도 하다. 면역 반응에 있어서 면역 세포들이 인식하는 외부 자극
중에서도 중추적인 역할을 하는 cytokine 단백질은 최근까지도 새로운 종류가 발견되고 있을 정도로 굉장히 그 종류가 다양하고 그만큼 면역 반응에 있어 다양한 역할을 수행한다. 인터페론
감마(IFN-γ)는 가장 잘 알려진 cytokine 중에 하나로 NK 세포, Th1 세포, cytotoxic T세포 등에 의해 분비되고 이에 대한 수용체를 갖는 면역 세포에서
JAK(Janus kinase)-STAT1 pathway에 의해 ISGs(Interferon-stimulated genes)라 불리는 유전자들을 활성화시키면서 기능을 수행하게 된다.
인터페론 감마의 가장 중요한 target이 되는 면역 세포는 대식세포(macrophage)로 인터페론 감마가 초기에 'macrophage-activating factor'로 불려진 것처럼
대식세포의 활성화에 있어서 핵심적인 역할을 한다(1).
대부분의 경우 세포에 대한 특정 자극이 어떠한 영향을 미치는지를 연구할 때 그 시작은 먼저 특정 자극에 의해 활성화되는 signaling pathway를 찾고 그 downstream에
해당하는 전사 인자(transcription factors)가 특정 유전자의 조절 부위(promoter 또는 enhancer)에 결합하여 발현을 증가시키는 유전자들을 찾는 것이다. 인터페론
감마 역시 Jak-STAT1-ISGs의 핵심 부분에 대한 연구는 상당히 오랜 기간 동안 수많은 연구들이 진행되어왔고 많은 조절 기전에 대해 잘 알려져 있다(2). 생리학에서 중요하게
생각하는 항상성(homeostasis)의 개념에서처럼 생명체에서는 균형이 중요하고 이를 음양(yin and yang)의 원리로 생각하면 세포에서 일어나는 특정 자극에 대한 반응을 볼 때
대부분 한 부분에만 치우쳐 있다는 것을 알 수 있다. 실제로 특정한 자극에 대한 세포의 반응은 많은 유전자들의 발현을 증가시키는 것과 동시에 다른 그룹의 많은 유전자들의 발현은 반대로
감소시킨다. 하지만 앞서 언급했듯이 대부분의 경우 먼저 자극에 의해 증가하는 유전자들을 RT-qPCR과 같은 방법을 통해 찾고, promoter 부분에 결합하는 전사 인자를 ChIP을 통해
확인하게 된다. 이러한 경우 한쪽 방향에만 치우치기 쉬워서 자극에 대한 전체적인 변화를 하나의 코끼리로 볼 때 '장님 코끼리 만지기'의 함정에 빠질 수 있다. 최근 10년동안 차세대
염기서열 분석 기술의 발달로 전사체(transcriptome)와 후성유전체(epigenome)를 분석하는게 가능해짐에 따라 세포의 자극에 대한 반응을 총체적으로 알 수 있게 되었다.
이번 논단에서는 전체적인 전사체와 후성유전체 수준에서 특정한 자극(인터페론 감마)에 의해 반응하는 대식세포의 조절 기전을 기존과 다른 시작에서 접근하여 그동안 잘 밝혀지지 않았던 인터페론
감마의 면역 조절 반응에서의 역할과 이에 중요한 후성유전학적 기전 그리고 이러한 결과가 만성 염증 및 인간 질병에 대한 이해에 어떠한 중요한 부분을 포함하는지에 대해 이야기 하려고 한다.
본론
인터페론 감마에 의한 히스톤 메틸레이션의 증가
특정한 자극에 의해 발현이 감소하는 유전자들에 대한 기전을 설명하기 위해서는 신호 전달 과정의 변화로만 이해하기 어려운 부분들이 많다. 실제로 인터페론 감마에 의한 반응에서도 앞에서
언급되었던 발현이 증가하는 ISGs가 아닌 반대로 발현이 억제되는 많은 유전자들의 경우 Jak-STAT1과 같은 신호 전달 과정의 변화만으로는 많은 경우 설명이 되지 않는다. 그렇다면
어떠한 반대 방향의 조절 기전이 인터페론 감마에 의해 유전자들의 발현을 억제시킬 것인가? 유전자 발현 억제와 관련되어 발생 과정에서 많이 알려진 후성유전학(epigenetics)인 변화가
중요한 역할을 한다(3). 이중에서도 히스톤 메틸레이션(histone methylation)의 증가는 gene silencing과 관련하여 중요한 역할을 하며 주로 H3K27me3와
H3K9me3의 변화가 일어날 때 유전자 발현의 감소가 나타난다(4). 이러한 히스톤 마커(histone marker)들을 전체 염색체 수준에서 알아보기 위해 이 마커들에 결합하는 항체들을
이용한 ChIP-seq 방법을 사용할 수 있다. 이 방법을 통해 인터페론 감마가 대식세포를 자극하였을 때 어떠한 지역에서 H3K27me3과 같은 negative 히스톤 마커가 증가하였는지를
찾아낼 수 있다. 분석 결과 특정 유전자들의 promoter에서 H3K27me3 마커가 인터페론 감마에 의해 증가하는 것이 확인되었고 이러한 유전자들은 인터페론 감마를 처리하기 전에는 높게
발현하고 있다가 자극에 의해서 현저하게 억제된다는 것을 확인했다(5). H3K27me3의 히스톤 메틸화를 유도하는 histone methyltransferase인 EZH2에 결합이 이
부분에 있었고 EZH2에 대한 inhibitor를 처리하였을 때 인터페론 감마에 의한 유전자들의 발현 억제 효과가 감소하는 것을 확인하였다. 인터페론 감마에 의해 억제되는 유전자들 중에
사실 위에서 언급된 히스톤 메틸화와 관련된 유전자들은 전체의 10%도 되지 않는다. 그렇다면 이러한 유전자들에 대한 조절 기전은 어떤 중요한 의미를 갖는가? 또한 나머지의 대다수의
유전자들은 어떠한 다른 기전에 의해 조절되는가 하는 의문을 갖게 된다.
히스톤 메틸화에 의해 억제되는 소수의 유전자들 중에서는 MERTK, PPARG, RANK와 같은 M2-like 기능에 중요한 유전자들이 조절되기 때문에 대식세포의 기능과 관련하여 큰 의미를
갖는다. 인터페론 감마는 서론에서 언급되었듯이 대식세포를 염증 반응을 증가시키는 형태(M1-like)로 활성화 시키는데 기여한다. 그리고 이와 동시에 정반대되는 특성인 M2-like
대식세포가 되지 못하도록 차단하게 된다 (그림 1). 이뿐만 아니라 이러한 negative histone marker와 관련된 억제는 또다른
중요한 대식세포의 기능과 연관되어 있다. 최근
연구결과들에 따르면 대식세포는 특정한 자극에 일정 시간 이상 노출되었을 때 그 자극에 대한 반응성을 일정 기간이상 유지하려고 하는 특성을 갖는데 이를 'innate immune
memory' 또는 'trained immunity'라 부른다(6, 7). 이러한 특성은 만성 염증과 같은 환경에서 염증이 지속적으로 유지되고 악순환을 반복하게 되는 이유와
연관되어있다(8). 실제로 만성 염증과 관련된 많은 질병에서 이러한 현상이 관찰된다. 인터페론 감마에 의해 H3K27me3가 promoter에 증가된 유전자들은 인터페론 감마가 완벽하게
제거된 상황에서도 수일 동안 억제된 상태를 유지하게 된다. 이러한 억제된 상태는 H3K27me3 마커가 promoter에 계속적으로 남아있는 것과 일치한다. 또한 M2-like 기능과
관련된 유전자들의 발현을 증가시키는 Dexamethasone이나 IL-4와 같은 자극을 인터페론 감마가 제거된 이후에 처리하여도 일정기간 동안 발현이 증가하지 않는다. 이러한 인터페론
감마와 반대되는 다른 자극에 반응성을 억제하는 것은 만성 염증 질환의 치료를 위한 약물의 효과가 효과적으로 나타나지 못하는 것과 관련이 되어있다. 이처럼 면역 세포에 인터페론 감마라는
하나의 자극이 나타내는 결과는 단순히 이에 반응하는 유전자들의 발현을 증가시키는 것보다 훨씬 더 복잡한 조절 기전을 갖고 있으며 생리학적으로도 염증 반응, 질병과 연관되어 중요한 의미를
갖는다. 다음의 질문으로 히스톤 메틸레이션으로 설명되지 않는 많은 수의 유전자들은 어떠한 다른 조절을 기전을 갖는지에 대해 알아보기 위해 유전자 조절 부위 중 enhancer에 대한 접근을
해보고자 한다 (그림 2).
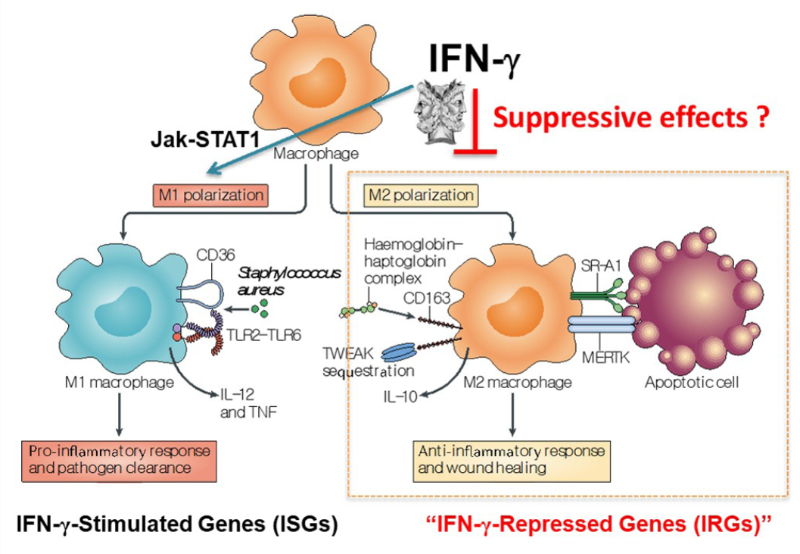
그림 1. 인터페론 감마에 의한 대식세포의 양방향의 조절
그림 2. 인터페론 감마 자극에 의한 대식세포의 후성유전체적 변화
대식세포의 특징을 결정하는 인핸서들과 인터페론 감마에 의한 조절 기전
발생과정에서 세포의 특성을 결정하는 중요한 전사 인자들은 세포 타입마다 특이적인 인핸서(enhancer)들을 결정하는데 중요한 역할을 한다(9). 세포마다 하나의 유전자는 같은 위치에
하나의 프로모터(promoter)만을 갖고 있지만 각각의 세포마다 enhancer들은 특이적으로 다른 부분들이 존재하게 된다. 이러한 cell type-specific enhancer들은
세포의 특성을 결정하는데 중요한 역할을 한다. 보통 이러한 enhancer들은 세포가 분화하는 과정에 미리 결정되어 특정한 세포로 분화가 끝난(terminally
differentiated) 이후에는 특정 자극에 의해 enhancer들의 위치가 변화하지 않는다는 것이 기존의 가설이었다. 하지만 2013년 Natoli 그룹에서 Cell에 발표한
latent enhancer에 대한 논문에 의해 그렇지 않은 enhancer들도 존재한다는 것이 밝혀졌다(10). Natoli 그룹은 마우스의 대식세포를 이용하여 LPS에 대한 자극을
주었을 때 특이적으로 소수의 발생과정에서 enhancer가 아니었던 부분이 새롭게 enhancer가 되는 것을 확인하였다. 보통 enhancer를 결정짓는 특징인 open
chromatin과 lineage determining transcription factors(LDTFs)의 결합은 어떠한 자극에 의해서 크게 변화하지 않는다. 하지만 이 연구에서 찾은
latent enhancer는 LPS나 인터페론 감마와 같은 자극에 의해서 새롭게 closed chromatin이 열리게 되고 LDTFs의 결합이 증가하는 것을 확인하였다. 이러한
latent enhancer는 앞에서 언급되었던 대식세포의 중요한 특징인 innate immune memory와 관련하여 특정 유전자들의 발현이 자극이 사라진 이후에도 유지되는데 중요한
역할을 한다(11). 이러한 관점에서 인터페론 감마에 의해 발현이 증가되는 유전자들 중에 일부가 latent enhancer에 의해 조절된다고 할 때, 이와 정반대가 되는 현상이 혹시 있지
않을까 하는 의문을 갖게 된다. 이에 대한 해답을 얻기 위해 다음과 같은 후성유전체적 접근 방식을 사용하여 enhancer들을 분석해볼 수 있다.
Enhancer repertoires(또는 landscapes)를 분석하기 위해선 open chromatin을 찾아내기 위해 사용되는 ATAC-seq과 PU.1과 같은 LDTFs에 대한
ChIP-seq 그리고 active enhancer 마커인 H3K27ac에 대한 ChIP-seq 결과가 종합적으로 사용되어진다(12). 이러한 접근 방식은 특정 조직에 일부 존재하여 조직의
특성을 조절하는 tissue-resident macrophages에서도 활용되어 lung, liver, kidney등에서 존재하는 대식세포들이 각각 다른 enhancer
repertoires를 갖고 있다는 것이 밝혀졌다(13-15). 인터페론 감마를 자극한 대식세포와 그렇지 않은 대식세포에 대하여 위와 같은 active enhancer landscape을
분석해보면 실제로 RNA-seq을 통해 분석한 유전자들의 변화와 상당히 유사한 패턴으로 enhancer들의 active status (H3K27ac)가 변화하는 것을 확인할 수 있다.
이중에서도 일부의 enhancer들은 위에서 언급된 latent enhancer와는 정반대로 원래는 open chromatin과 LDTFs의 결합을 갖고 있다가 인터페론 감마에 의해서
LDTFs의 결합이 현저히 감소하고 closed chromatin으로 변하는 것을 확인하였다. 이러한 부분에 대한 enhancer peaks을 이용하여 motif 분석을 수행한 결과 MAF
라고 하는 전사 인자가 이 enhancer들에 중요하다는 것을 알게 되었고 인터페론 감마에 의한 MAF의 감소가 latent enhancer와 정반대의 특성을 보이는
'disassembled enhancer'에 관련이 되어있다는 것을 밝히게 되었다(16). 이러한 전사 인자 MAF의 감소와 disassembled enhancer의 변화와 관련된
유전자들의 발현 감소는 류마티스 관절염 환자에서 분리된 synovial macrophages에서도 확인되었다. 이를 통해 인터페론 감마는 promoter 부위에서의 히스톤 메틸레이션과
관련되지 않는 많은 유전자들도 enhancer 부위에서의 후성유전체적 변화를 통해서 억제할 수 있다는 것을 알게 되었다. 앞에서 언급된 음양(yin and yang)의 변화가 유전자 수준과
후성유전체 수준에서 평행하게 진행된다는 것을 대식세포를 통해 확인한 것이다.
대식세포의 2차 자극에 대한 반응성을 변화시키는 인터페론 감마
특정한 자극에 의한 대식세포의 후성유전체적 변화는 이후에 오는 2차 자극에 대한 반응성을 변화시킨다. 예를 들어 인터페론 감마로 일정 시간이상 자극된(priming 이라고 함) 대식세포의
경우 또다시 인터페론 감마로 자극했을 때 처음보다 훨씬 더 높은 수준의 ISGs 발현을 오랜 시간 동안 유도하게 된다(17). 이러한 현상은 다른 2차 자극인 TLR4를 통해 신호를
전달하는 LPS로 자극했을 때도 나타난다(18). 보통 TNF나 IL6와 같은 염증성 cytokine 유전자는 인터페론 감마에 의해서는 발현이 유도되지 않고 LPS에 의해서 유도된다.
LPS에 의한 TNF, IL6 유전자의 발현 증가는 transient하게 일어나서 일정시간이 지나면 negative feedback에 의해 발현이 억제된다. 하지만 인터페론 감마의 자극에
먼저 노출된(IFN-γ primed) 대식세포의 경우, 그 이후에 LPS를 자극을 받게 되면 원래의 TNF, IL6 유전자 발현보다 훨씬 더 높은 수준의 발현을 유도하게 되며 오랜 시간
동안 발현을 유지한다(19). 인터페론 감마의 자극이 있을 때 TNF, IL6 유전자의 발현을 직접적으로 유도하지는 않지만 이때 유전자 조절 부위(promoter, enhancer)의
chromatin 상의 변화를 일으켜 LPS 자극이 왔을 때 보다 빨리 transcription을 할 수 있고 오랫동안 유지시켜주게 한다. 이러한 역할은 인터페론 감마에 의해 유도되는
STAT1과 IRF1의 결합에 의해서 이루어진다. 자가면역질환 환자에게서 흔히 발견되는 Interferon signature는 인터페론에 반응하는 많은 유전자들과 연관되어있다. 인터페론
감마는 단순히 Jak-STAT1 신호전달에 직접적으로 반응하는 ISGs의 발현을 유도하는 것뿐만 아니라 후성유전체적 변화를 일으켜 LPS와 같은 2차적인 자극에 대한 반응성까지 변화시키면서
만성 염증 반응에 크게 기여하게 된다(20).
인터페론 감마에 의한 후성유전체적 변화는 2차 자극에 대한 반응성에 있어서도 한쪽 방향이 아닌 양쪽 방향에 동시에 영향을 준다. 위에서 언급했듯이 인터페론 감마는 LPS에 의해 유도되는
TNF와 IL6와 같은 유전자들의 발현을 더욱 증가시키고, 이와 동시에 IL10과 같은 유전자의 발현은 반대로 감소시킨다. LPS에 의한 반응에 있어서 중요한 역할은 하는 IL-10은
anti-inflammatory cytokine으로 염증성 cytokine 유전자들에 비해 더 늦은 시간에 발현되어 negative feedback을 통해 염증 반응을 억제한다(21).
인터페론 감마에 의해 유도되는 핵심 전사 인자가 STAT1이라 하면 IL-10은 STAT3 전사 인자를 유도하는데 이 두 전사 인자는 같은 DNA 결합 부위를 두고 서로 경쟁을 하기 때문에
이 두 전사 인자의 균형이 면역 반응에 있어서 중요한 역할을 하게 된다. 일반적인 LPS 반응에 있어서 STAT3의 결합을 ChIP-seq을 통해 확인해보면 IL-10과 같은 유전자들의
조절 부위(특히 enhancer)에 집중되어있다(22). 이러한 STAT3의 결합은 active enhancer 마커인 H3K27ac와 연결되어있고 또한 promoter-enhancer
looping 관련된 Mediator, Cohesin의 결합과도 관련되어있다. 이러한 STAT3의 결합은 인터페론 감마가 priming된 상황에서 STAT1이 dominant한 상황이 되면
같은 DNA 결합 부위를 갖고 있는 STAT3의 경우 경쟁적으로 그 결합이 억제된다. 이러한 경쟁관계에 있어서 또 하나 특이한 점은 STAT1과 STAT3 둘다 동일한 DNA 결합 부위를
인식하지만 이 결합 부위 주변에 어떤 다른 전사 인자의 결합 부위가 있는지를 분석해보면 서로 상반된 결과를 보인다는 점이다. STAT1의 경우 STAT motif와 IRF motif를 갖고
있는 부위에 결합하기를 선호하고 그렇기 때문에 IRF1과 같은 전사 인자가 co-binding을 하는 경우가 많다. 반대로 STAT3의 경우엔 STAT motif와 AP-1 motif가
같이 있는 경우에 결합하기를 선호하는 경향이 있다. 그러므로 실제 TNF, IL6와 같은 유전자 조절 부위에서 찾아지는 STAT 결합 부위는 STAT-IRF motif를 보이고, IL10과
같은 유전자 조절 부위에서는 STAT-AP1 motif를 주로 보인다. 이러한 STAT1/STAT3의 균형 조절은 유전자에 따라 DNA 상의 결합 부위에서 intrinsic하게 암호화
되어있다는 것을 알 수 있고 이에 따라 인터페론 감마와 같은 자극이 왔을 때 유전자 조절 부위의 후성유전체적 변화를 일으키는데 있어 핵심적인 역할을 한다고 할 수 있다. 실제로 STAT3가
결합되어있는 enhancer 부위에서 발현하는 eRNA(enhancer RNA)를 억제하였을 때 인터페론 감마와 같은 방식으로 IL-10 유전자를 억제하는 것을 확인할 수 있다(23).
이는 인터페론 감마에 의한 STAT1/STAT3의 균형의 변화가 초래하는 후성유전체적 변화가 enhancer 부위에서 주로 일어나고 이를 통해서 LPS 반응에 있어서 서로 다른 그룹의
유전자들을 상반된 방향으로 조절할 수 있다는 것을 나타낸다. 이러한 인터페론 감마에 의한 양방향의 조절은 대식세포를 염증성 M1-like 형태로 만들어주는데 있어 큰 역할을 하고 이는 만성
염증을 동반하는 다양한 인간 질병에 있어서 중요한 역할을 하게 된다(24).
인터페론 감마와 선천성 면역 기억
면역학에 있어서 면역학적 기억(immunological memory)은 후천성 면역(adaptive immunity)에 핵심이며 주로 T세포와 B세포에 의해서 이루어진다. 그러므로 선천성
면역 세포인 대식세포의 경우 면역 기억과는 전혀 관련이 없다는 것이 과거에 일반적인 생각이었다. 하지만 이번 논단에서 주로 다룬 인터페론 감마의 경우에서 볼 수 있듯이 대식세포에도 어느
정도 일정기간 그 자극에 대한 효과가 유지되는 '기억'과도 같은 현상이 존재함을 알 수 있다(25). 면역학적 기억과는 다른 개념이지만 기억이라는 말을 사용하는 것에 대한 거부감을 갖는
사람들이 역시 존재하여 다른 말로 훈련된 면역(trained immunity)이라고도 많이 쓰이고 있다. 이러한 반응은 BCG 백신과 같은 경우에도 실제로 대식세포를 통해 후성유전체적
변화가 일어나는 것이 확인되었고 이는 백신에 의한 효과에도 관련이 있음을 알게 되었다. 반응이 증가하는 관점에서 trained immunity를 이야기 한다면 다른 관점에서 이와는 반대로
억제되는 유전자들의 경우는 Sepsis 환자들에서 나타나는 endotoxin tolerance와 같은 현상에서 찾아볼 수 있다(26). 이러한 경우엔 특정 유전자들이 이전에 경험한 자극에
의해서 반응성이 감소하는 것을 보이기 때문에 tolerance라고 부를 수 있다. 이러한 두가지의 경우 모두 우리가 보고자 하는 유전자들(TNF, IL-6와 같은 염증성 cytokine)을
기준으로 이야기 하는 것이기 때문에 실제 전사체, 후성유전체 수준에서의 변화는 인터페론 감마에서 볼 수 있듯이 항상 양쪽 방향의 변화가 동시에 일어난다. 인터페론 감마의 경우 보통
박테리아에 대한 감염에 의한 반응으로 주로 일어나게 되는데 이와 반대되는 기생충에 대한 감염은 IL-4라는 cytokine에 의해서 일어난다. 실제로 박테리아에 대한 감염의 경험은 기생충에
대한 면역 반응을 감소시키고 반대로 기생충에 대한 감염은 박테리아에 대한 면역 반응을 감소시킨다. 이러한 변화는 인터페론 감마와 IL-4에 의해 유도되는 STAT1과 STAT6 전사 인자에
의해 후성유전체적 수준에서 대식세포가 변화하기 때문에 나타나는 현상이다(27). 이처럼 인터페론 감마는 IL-4 또는 IL-10과 같은 서로 상반된 cytokine들과 대식세포에 서로
경쟁하며 반응에 있어서 균형을 이루고 이러한 균형이 무너졌을 때 일어나는 후성유전체적인 변화는 실제 질병과도 밀접한 관련이 있음을 알 수 있다.
결론
차세대 염기서열 분석 방법을 이용한 연구의 가장 큰 이점은 unbiased approach를 통해 기존에 간과했던 부분에 대한 연구를 가능하게 했다는 것이다. 세포에 특정한 자극을 주었을
때 나타나는 반응을 전사체와 후성유전체 수준에서 보게 되면 증가하는 유전자들과 감소하는 유전자들이 모두 존재하고 유전자 조절 부위에서의 변화 역시 이와 마찬가지로 일어나게 된다. 인터페론
감마에 대한 연구가 과거에 주로 증가하는 유전자들에 초점이 맞춰져 있었고 이와 관련된 신호전달에 대한 이해를 제공했다고 하면 최근 10년간의 연구 이와는 다른 방향으로 진행되어왔다.
인터페론 감마에 의해 오히려 발현이 감소하는 유전자들과 또한 2차 자극에 의한 반응 역시 감소하는 유전자들을 통해 새로운 인터페론 감마의 기능들을 알게 하였다. 이를 통해서 만성 염증
반응의 악순환과 관련된 중요한 단서들을 찾는데 큰 역할을 하게 되었다(28).
대식세포를 포함한 선천성 면역세포에서 후성유전체적 변화를 통한 innate immune memory에 대한 연구는 최근 5년간 여러 질병들과 연관하여 아주 활발하게 진행되어 많은 논문들이
발표되고 있다. 인터페론 감마를 포함한 세포 주변 환경의 자극들 사이에서 일어나는 서로간의 상호 조절과 이에 핵심이 되는 전사 인자에 대한 연구, 후성유전체적 변화와 더불어 일어나는
세포에서의 metabolic change에 대한 연구(29), scRNA-seq, scATAC-seq 등의 기술을 통한 인간 질병 조직에서의 주변 환경에 대한 면역 세포 연구 등은 앞으로도
많은 새로운 질문을 가지고 이에 대한 답변을 얻기 위해 연구들이 진행될 것으로 예상한다(30). 이러한 연구들은 궁극적으로 만성 염증과 관련된 인간 질병의 복잡한 기전을 이해하는데 큰
기여를 하게 될 것이다.
저자약력
2000-2004 서강대학교 생명과학과, 학사
2004-2006 광주과학기술원 생명과학과, 석사
2010-2016 Cornell University 면역학, Ph.D.
2016-2017 Hospital for Special Surgery, Postdoctoral fellow
2017-2018 한국과학기술연구원, 선임연구원
2018-현재 충북대학교 생명과학부, 조교수
참고문헌
1. Ivashkiv LB. 2018. IFNγ: signalling, epigenetics and roles in immunity, metabolism, disease and cancer immunotherapy. Nature Reviews Immunology
2.
Ehrt S, Schnappinger D, Bekiranov S, Drenkow J, Shi S, Gingeras TR, Gaasterland T, Schoolnik G, Nathan C. 2001. Reprogramming of the Macrophage Transcriptome in Response to Interferon-γ and Mycobacterium tuberculosis: Signaling Roles of Nitric Oxide Synthase-2 and Phagocyte Oxidase. The Journal of Experimental Medicine 194: 1123-403.
Smale ST, Tarakhovsky A, Natoli G. 2014. Chromatin Contributions to the Regulation of Innate Immunity. Annual Review of Immunology 32: 489-5114.
Fang TC, Schaefer U, Mecklenbrauker I, Stienen A, Dewell S, Chen MS, Rioja I, Parravicini V, Prinjha RK, Chandwani R, MacDonald MR, Lee K, Rice CM, Tarakhovsky A. 2012. Histone H3 lysine 9 di-methylation as an epigenetic signature of the interferon response. The Journal of Experimental Medicine 209: 661-95.
Qiao Y, Kang K, Giannopoulou E, Fang C, Ivashkiv Lionel B. 2016. IFN-γ Induces Histone 3 Lysine 27 Trimethylation in a Small Subset of Promoters to Stably Silence Gene Expression in Human Macrophages. Cell Reports 16: 3121-96.
Netea MG, Latz E, Mills KHG, O'Neill LAJ. 2015. Innate immune memory: a paradigm shift in understanding host defense. Nat Immunol 16: 675-97.
Netea MG, Joosten LAB, Latz E, Mills KHG, Natoli G, Stunnenberg HG, O’Neill LAJ, Xavier RJ. 2016. Trained immunity: A program of innate immune memory in health and disease. Science 3528.
Langlais D, Barreiro LB, Gros P. 2016. The macrophage IRF8/IRF1 regulome is required for protection against infections and is associated with chronic inflammation. J. Exp. Med. 2139.
Heinz S, Romanoski CE, Benner C, Glass CK. 2015. The selection and function of cell type-specific enhancers. Nat Rev Mol Cell Biol 16: 144-5410.
Ostuni R, Piccolo V, Barozzi I, Polletti S, Termanini A, Bonifacio S, Curina A, Prosperini E, Ghisletti S, Natoli G. 2013. Latent Enhancers Activated by Stimulation in Differentiated Cells. Cell 152: 157-7111.
Kaikkonen Minna U, Spann Nathanael J, Heinz S, Romanoski Casey E, Allison Karmel A, Stender Joshua D, Chun Hyun B, Tough David F, Prinjha Rab K, Benner C, Glass Christopher K. 2013. Remodeling of the Enhancer Landscape during Macrophage Activation Is Coupled to Enhancer Transcription. Molecular Cell 51: 310-2512.
Gosselin D, Glass CK. 2014. Epigenomics of macrophages. Immunological Reviews 262: 96-11213.
Glass CK. 2015. Genetic and genomic approaches to understanding macrophage identity and function. Arterioscler. Thromb. Vasc. Biol. 35: 755-6214.
Lavin Y, Winter D, Blecher-Gonen R, David E, Keren-Shaul H, Merad M, Jung S, Amit I. 2014. Tissue-Resident Macrophage Enhancer Landscapes Are Shaped by the Local Microenvironment. Cell 159: 1312-2615.
Saeed S, Quintin J, Kerstens HHD, Rao NA, Aghajanirefah A, Matarese F, Cheng S-C, Ratter J, Berentsen K, van der Ent MA, Sharifi N, Janssen-Megens EM, Ter Huurne M, Mandoli A, van Schaik T, Ng A, Burden F, Downes K, Frontini M, Kumar V, Giamarellos-Bourboulis EJ, Ouwehand WH, van der Meer JWM, Joosten LAB, Wijmenga C, Martens JHA, Xavier RJ, Logie C, Netea MG, Stunnenberg HG. 2014. Epigenetic programming of monocyte-to-macrophage differentiation and trained innate immunity. Science 34516.
Kang K, Park SH, Chen J, Qiao Y, Giannopoulou E, Berg K, Hanidu A, Li J, Nabozny G, Kang K, Park-Min K-H, Ivashkiv LB. 2017. Interferon-γ Represses M2 Gene Expression in Human Macrophages by Disassembling Enhancers Bound by the Transcription Factor MAF. Immunity 47: 235-50.e417.
Glass CK, Natoli G. 2016. Molecular control of activation and priming in macrophages. Nat. Immunol. 1718.
Chen J, Ivashkiv LB. 2010. IFN-γ abrogates endotoxin tolerance by facilitating Toll-like receptor-induced chromatin remodeling. Proceedings of the National Academy of Sciences 107: 19438-4319.
Qiao Y, Giannopoulou Eugenia G, Chan Chun H, Park S-h, Gong S, Chen J, Hu X, Elemento O, Ivashkiv Lionel B. 2013. Synergistic Activation of Inflammatory Cytokine Genes by Interferon-γ-Induced Chromatin Remodeling and Toll-like Receptor Signaling. Immunity 39: 454-6920.
Ivashkiv LB. 2013. Epigenetic regulation of macrophage polarization and function. Trends in Immunology 34: 216-2321.
Hu X, Ivashkiv LB. 2009. Cross-regulation of Signaling Pathways by Interferon-γ: Implications for Immune Responses and Autoimmune Diseases. Immunity 31: 539-5022.
Kang K, Bachu M, Park SH, Kang K, Bae S, Park-Min K-H, Ivashkiv LB. 2019. IFN-γ selectively suppresses a subset of TLR4-activated genes and enhancers to potentiate macrophage activation. Nature Communications 10: 332023.
Lam MT, Li W, Rosenfeld MG, Glass CK. 2014. Enhancer RNAs and regulated transcriptional programs. Trends Biochem. Sci. 39: 170-8224.
Begitt A. 2014. STAT1-cooperative DNA binding distinguishes type 1 from type 2 interferon signaling. Nat. Immunol. 1525.
Monticelli S, Natoli G. 2013. Short-term memory of danger signals and environmental stimuli in immune cells. Nat Immunol 14: 777-8426.
Novakovic B. 2016. β-Glucan reverses the epigenetic state of LPS-induced immunological tolerance. Cell 16727.
Piccolo V, Curina A, Genua M, Ghisletti S, Simonatto M, Sabo A, Amati B, Ostuni R, Natoli G. 2017. Opposing macrophage polarization programs show extensive epigenomic and transcriptional cross-talk. Nat Immunol 18: 530-4028.
Schultze JL, Rosenstiel P. 2018. Systems Medicine in Chronic Inflammatory Diseases. Immunity 48: 608-1329.
Phan AT, Goldrath AW, Glass CK. 2017. Metabolic and Epigenetic Coordination of T Cell and Macrophage Immunity. Immunity 46: 714-2930.
Giladi A, Amit I. 2018. Single-Cell Genomics: A Stepping Stone for Future Immunology Discoveries. Cell 172: 14-21
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.



