기술동향
단분자 서열 분석기를 이용한 RNA 수식 연구
- 등록일2020-09-29
- 조회수5058
- 분류기술동향
-
자료발간일
2020-09-15
-
출처
한국분자ㆍ세포생물학회
- 원문링크
-
키워드
#단분자#서열#RNA
단분자 서열 분석기를 이용한 RNA 수식 연구
장혜식(서울대학교 생명과학부)
RNA 수식의 기능과 종류
잘 알려진 사람 단백질의 0.8%는 RNA를 수식(modify)할 수 있다 [1]. DNA나 단백질과 마찬가지로 RNA도 기본 화학 구조에서 추가적인 변화를 겪으면 안정성과 구조, 단백질 결합 등에서 차이가 생겨난다 [2, 3]. 아데노신의 N6에 메틸기가 붙는 6-methyladenosine (m6A), 유리딘의 N1 대신 C5에 당이 결합하는 pseudouridine (ψ), 백본 2′ 자리의 수산화기에 메틸이 추가되는 2′-O-methylation (Nm)이 대표적이다. 수식된 염기가 가장 흔한 곳은 tRNA다. 세포 안의 tRNA에서는 20-30%의 염기가 ψ와 dihydrouridine을 포함한 다양한 형태로 변한다. rRNA도 또한 7-methylguanosine (m7G), 1-methyladenosine (m1A) 등을 포함한다. mRNA도 낮은 빈도이지만 수식이 존재한다. m6A가 가장 잘 알려져 있는데, mRNA의 안정성과 단백질 생산 효율에 영향을 미친다 [3]. 포유동물의 mRNA에서 ψ, m7G, m1A이 얼마나 기능이 있을지는 아직 좀 더 연구가 필요하다 [4].
잘 알려진 사람 단백질의 0.8%는 RNA를 수식(modify)할 수 있다 [1]. DNA나 단백질과 마찬가지로 RNA도 기본 화학 구조에서 추가적인 변화를 겪으면 안정성과 구조, 단백질 결합 등에서 차이가 생겨난다 [2, 3]. 아데노신의 N6에 메틸기가 붙는 6-methyladenosine (m6A), 유리딘의 N1 대신 C5에 당이 결합하는 pseudouridine (ψ), 백본 2′ 자리의 수산화기에 메틸이 추가되는 2′-O-methylation (Nm)이 대표적이다. 수식된 염기가 가장 흔한 곳은 tRNA다. 세포 안의 tRNA에서는 20-30%의 염기가 ψ와 dihydrouridine을 포함한 다양한 형태로 변한다. rRNA도 또한 7-methylguanosine (m7G), 1-methyladenosine (m1A) 등을 포함한다. mRNA도 낮은 빈도이지만 수식이 존재한다. m6A가 가장 잘 알려져 있는데, mRNA의 안정성과 단백질 생산 효율에 영향을 미친다 [3]. 포유동물의 mRNA에서 ψ, m7G, m1A이 얼마나 기능이 있을지는 아직 좀 더 연구가 필요하다 [4].
전사체 수준의 기존 RNA 탐색 방법
전사체 수준에서 수식을 탐색하려면, 생화학적 전처리 후 RNA-Seq으로 변화를 찾는다. 세부적으로는 전처리 방법에 따라 다양한 접근법이 있다. MeRIP-seq에서는 RNA를 약하게 파편화한 뒤에, 수식 염기에 대한 항체로 RNA를 정제해서 분석한다. 화학반응을 이용해서 표지하거나 정제하기도 하는데, ψ에 대해서는 N-cyclohexyl-N′-(2-morpholinoethyl)carbodiimide (CMC)를 이용해서 선택적으로 표지해서 정제할 수 있다 [5, 6]. 항체나 단백질을 공유결합으로 붙인 뒤 남은 부분을 이용해서 검출하거나 [7, 8], 수식된 염기를 자르지 못하는 선택적 RNA분해효소를 처리해서 서열분석 결과에서 생성되는 패턴을 비교하여 수식된 염기의 위치를 확인한다 [9].
2세대 서열분석기를 활용하는 방법들은 여러가지 다른 방법으로 만든 정보를 대량으로 읽는데 유용하게 사용되고 있다. 하지만, 2세대 서열분석기는 cDNA로 반드시 변환해야 하기 때문에 역전사 과정에서 정보가 손실되며, 짧은 리드 길이로 인하여 스플라이싱, 폴리A 꼬리 같은 RNA 분자 상태가 소실된다. 유전자 수준에서 수식 빈도 분석이나 샘플 간 비교에서 빈도 변화를 분석할 수는 있지만, 유전자 또는 isoform 내에서 분자별로 발생하는 차이를 관찰하는 데는 활용하기 어렵다. 세포 내에서 RNA의 생애 주기 중 다양한 단계를 거치고 여러가지 다른 단백질과 상호작용하고 있는 분자들이 모두 혼합된 통계만 볼 수 있을 뿐이다.
단분자 서열분석기의 활용
대안으로 긴 리드를 제공하면서 증폭하지 않고 세포 유래 분자를 그대로 분석할 수 있는 단분자 서열분석기가 활용되기 시작했다. PacBio의 Single Molecule, Real-Time (SMRT) 서열분석기와 Oxford Nanopore Technologies (ONT)의 나노포어 서열분석기가 대표적이다 [10]. PacBio는 긴 리드를 활용하여 분자 단위 비교가 가능하지만 RNA는 cDNA로 전환이 거의 필수적이므로 RNA 수식을 개별적으로 분석하기는 어렵다. ONT의 나노포어 서열분석기는 리드 단위의 서열 정확도가 93–95% 정도로 상용화된 서열분석기 중 가장 낮다. 하지만, 100 kbp가 넘는 더욱 긴 리드를 제공하며, DNA중합효소를 사용하지 않아 왓슨-크릭 염기쌍으로 검출할 수 없는 핵산 염기들의 화학적 조성을 읽어낼 수 있다. 나노포어 서열분석기에서는 전기가 통하지 않는 막 사이에 1 nm 내외의 구멍을 내고 그 사이로 핵산을 통과시킨다 (그림 1). 핵산이 이동하면서 염기의 크기와 전자 밀도에 따라 구멍을 통해 통과할 수 있는 이온의 양이 시시각각 바뀌게 되고, 그 전류를 측정하여 서열을 분석할 수 있다 (그림 2).
대안으로 긴 리드를 제공하면서 증폭하지 않고 세포 유래 분자를 그대로 분석할 수 있는 단분자 서열분석기가 활용되기 시작했다. PacBio의 Single Molecule, Real-Time (SMRT) 서열분석기와 Oxford Nanopore Technologies (ONT)의 나노포어 서열분석기가 대표적이다 [10]. PacBio는 긴 리드를 활용하여 분자 단위 비교가 가능하지만 RNA는 cDNA로 전환이 거의 필수적이므로 RNA 수식을 개별적으로 분석하기는 어렵다. ONT의 나노포어 서열분석기는 리드 단위의 서열 정확도가 93–95% 정도로 상용화된 서열분석기 중 가장 낮다. 하지만, 100 kbp가 넘는 더욱 긴 리드를 제공하며, DNA중합효소를 사용하지 않아 왓슨-크릭 염기쌍으로 검출할 수 없는 핵산 염기들의 화학적 조성을 읽어낼 수 있다. 나노포어 서열분석기에서는 전기가 통하지 않는 막 사이에 1 nm 내외의 구멍을 내고 그 사이로 핵산을 통과시킨다 (그림 1). 핵산이 이동하면서 염기의 크기와 전자 밀도에 따라 구멍을 통해 통과할 수 있는 이온의 양이 시시각각 바뀌게 되고, 그 전류를 측정하여 서열을 분석할 수 있다 (그림 2).
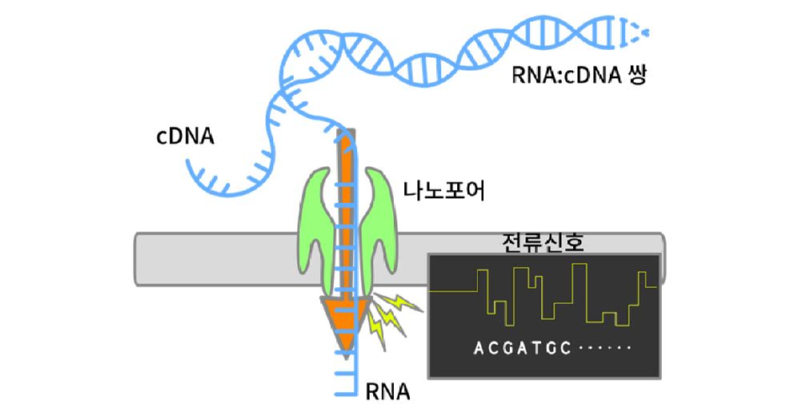
그림 1. 나노포어 RNA 시퀀싱 모식도.직경 1 nm 정도의 구멍이 있는 나노포어가 막에 삽입되어 있다. 위 아래로 -180 mV 정도의 전압이 걸려서 RNA가 +극인 아랫쪽으로 이동한다. 이동하는 RNA에서 통과하고 있는 염기의 종류에 따라 전류가 바뀌고, 그 전류를 측정해서 서열을 확인한다. (이 이미지는 DBCLS에서 제작한 TogoTV에서 재사용했음. Creative Commons 4.0-BY)
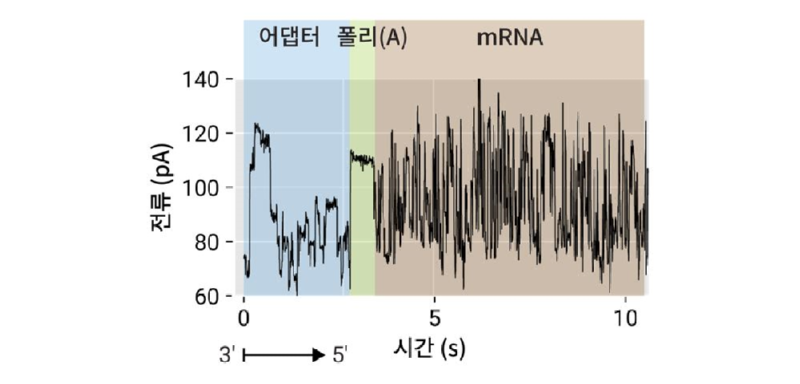
그림 2. 나노포어 RNA 시퀀싱 신호. 나노포어 RNA 시퀀싱은 3′에서 5′로 진행한다. DNA로 구성된 어댑터가 가장 먼저 등장한다. 120 pA에서 머무르는 부분은 RNA 이동 속도를 조절하는 모터 단백질이 붙는 부분이다. RNA의 3′ 끝에 있는 폴리(A)가 먼저 등장하고, mRNA가 그 다음에 따라온다.
단분자 서열분석기를 이용한 수식 검출
처음으로 염기 수식 검출의 가능성이 보인 것은 메틸화된 CpG DNA였다. 메틸화된 사이티딘(5mC)의 경우에는 C와는 꽤 다른 신호를 만들어냈기 때문에, 오히려 DNA서열분석에서는 정확도 향상에 방해가 됐다. 곧 5mC와 5-hydroxymethyl-2′-deoxycytidine (5hmC)에 대한 모델이 정립되면서 유전체 DNA의 메틸화 지도를 반수체별로 분리하여 작성하는 것이 전보다 훨씬 용이해졌다 [11, 12]. 그 외에도 6-methyl-2′-deoxyadenosine (6mA)에 대한 모델도 발표되고 [13, 14], 5-bromo-2′-deoxyuridine (BrdU), 5-ethynyluridine (EU) 등 대사표지를 이용해서 DNA 합성이 일어나는 부분을 높은 효율로 정확하게 조사하는 방법이 등장했다 [15, 16].
처음으로 염기 수식 검출의 가능성이 보인 것은 메틸화된 CpG DNA였다. 메틸화된 사이티딘(5mC)의 경우에는 C와는 꽤 다른 신호를 만들어냈기 때문에, 오히려 DNA서열분석에서는 정확도 향상에 방해가 됐다. 곧 5mC와 5-hydroxymethyl-2′-deoxycytidine (5hmC)에 대한 모델이 정립되면서 유전체 DNA의 메틸화 지도를 반수체별로 분리하여 작성하는 것이 전보다 훨씬 용이해졌다 [11, 12]. 그 외에도 6-methyl-2′-deoxyadenosine (6mA)에 대한 모델도 발표되고 [13, 14], 5-bromo-2′-deoxyuridine (BrdU), 5-ethynyluridine (EU) 등 대사표지를 이용해서 DNA 합성이 일어나는 부분을 높은 효율로 정확하게 조사하는 방법이 등장했다 [15, 16].
RNA 수식 검출은 초기부터 많은 기대를 모으며 제품 개발 단계에서부터 가장 중요한 관심사였다. ONT에서는 m6A와 m5C가 각각 A와 C와는 다른 신호를 발생해서 베이스콜 오류를 발생시키는 사례를 보고했다 [17]. UCSC에서는 세균의 16S rRNA에서 ψ와 m7G 위치를 마찬가지로 베이스콜 오류로 찾아서 다른 신호가 발생하는 것을 발견했다 [18]. 그리고, 2019년에는 나노포어 서열분석기로 RNA를 연구하는 학계 연구실들의 컨소시엄에서 인간 lymphoblastoid cell line인 GM12878에서 전사체 수준에서 mRNA에서 폴리(A), 스플라이싱 등과 함께 m6A를 분석했다 [19].
나노포어 서열분석기로 RNA 수식 위치를 확인하는 소프트웨어들은 미세한 차이도 민감하게 검출할 수 있도록 전류 신호 단계에서 주로 분석한다. Nanopolish와 Tombo는 검출된 신호를 참조 서열에서 예상되는 신호에 정렬한 뒤에, 참조 서열의 각 염기 위치에서 통계수치를 계산하는 방법으로 수식 위치를 찾는다 (표 1). 신호를 대조하는 방법들은 RNA의 다양한 부분과 연계 분석하는 새로운 기회를 제공했지만 여전히 활용이 쉽지는 않다. 한 염기가 아니라 주변의 5–9개 정도의 염기가 한꺼번에 특정 시점의 전류에 영향을 주며, RNA가 움직이는 속도가 크게 바뀌어서, 기술의 민감도와 활용도를 크게 제한한다 [10]. 분자 단위에서 통계적 불확실성이 높아 수식 빈도가 높은 곳이 아니면 위양성률이 높다. 정확도를 높이자면 반드시 양성 대조군이 될 표준 참조 모델이나 음성 대조군이 될 수식이 없는 샘플 등, 비교할 짝이 반드시 필요하다. 신호 수준의 검출법은 앞으로도 많은 개선이 필요할 전망이다.
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
관련정보
지식
동향

