기술동향
미생물의 염기 편집 기술(Base editing):효과적인 미생물 게놈 편집
- 등록일2020-11-18
- 조회수5464
- 분류기술동향
-
자료발간일
2020-11-12
-
출처
생물학연구정보센터(BRIC)
- 원문링크
-
키워드
#미생물#염기#편집#게놈#CRISPR
- 첨부파일
[BRIC View 리뷰논문요약]
미생물의 염기 편집 기술(Base editing):효과적인 미생물 게놈 편집
노명현 (포항공과대학교 화학공학과)
요약문
게놈 편집 및 개량 기술은 기초적이나 폭넓은 활용 분야를 갖는다. 이에 관해 CRISPR/Cas시스템을 활용한 상동 재조합(Homologous recombination, HR) 기술이 다양하게 개발되었으며, 간단하고 효율적이며 폭넓게 적용될 수 있다는 장점을 가진다. 최근에는 CRISPR/Cas 시스템과 핵산 탈아미노효소(Nucleobase deaminase)를 결합한, 염기 편집(Base editing) 기술이개발되었고 게놈 편집 기술의 새로운 장을 열고 있다. 특히, 해당 기술은 기존 CRISPR/Cas기반 HR 기술의 세포에 치명적인 DNA의 이중 가닥 절단, 외부 DNA의 도입과 같은 과정없이도 핵산 변이를 진행할 수 있다는 점에 장점을 갖는다. 본 논문에서는 최근 이러한 염기 편집 기술의 개발과 미생물 내의 다양한 활용에 대해 다루고자 한다. 더불어, 해당 기술과 관련된 gRNA 디자인 및 데이터 분석을 용이하게 해 줄 생물정보학적 도구에 대해서도요약해보고자 한다.
Key Words: 염기 편집 기술(Base editing), 게놈 편집, CRISPR 시스템
※ 본 자료는 Microbial Base Editing: A Powerful EmergingTechnology for Microbial Genome Engineering. Trends Biotechnol.(2020).의 논문을 한글로 번역, 요약한 자료입니다.
[목 차]
1. 염기 편집 기술: CRISPR/Cas 시스템의 활용을 통한 신규 게놈 편집 기술
2. 원핵 생물 내 염기 편집 기술
2.1. 대장균(Escherichia coli)
2.2. 코리네박테리움 글루타미쿰(Corynebacterium glutamicum)
2.3. 스트렙토마이세스 종(Streptomyces Species)
2.4. 클로스트리디움 베이예린키이(Clostridium beijerinckii)
2.5. 슈도모나스 종(Pseudomonas Species)
2.6. 로도박터 스페로이데스(Rhodobacter sphaeroides)
2.7. 슈와넬라 오나이덴시스(Shewanella oneidensis)
2.8. 병원성 박테리아(Pathogenic Bacteria)
3. 진핵 생물 내 염기 편집 기술
3.1. 사카로미세스 세레비시아(Saccharomyces cerevisiae)
3.2. 야로위아 리포리티카(Yarrowia lipolytica)
3.3. 아스페르질루스 니게르(Aspergillus niger)
4. 염기 편집 기술의 활용을 위한 생물정보학적 보조 도구
4.1. gRNA 디자인 도구
4.2. 시퀀싱(Sequencing) 데이터 분석 도구
5. 결론 및 향후 기술 전망
1. 염기 편집 기술: CRISPR/Cas 시스템의 활용을 통한 신규 게놈 편집 기술
CRISPR/Cas 시스템은 간편성과 효율성 덕에 유전자 편집 기술 분야에 폭넓게 활용되고있으며, 해당 분야의 획기적인 발전을 이루고 있다. CRISPR/Cas 시스템을 통해 이중 가닥절단(dsDNA breaks, DSBs)이 생기면 원하는 형태로 도입된 DNA 절편의 상동성 재조합(Homologous recombination, HR)이 일어나는 원리로 유전자 편집이 진행될 수 있다. 진핵 생물 내에서는 DNA 절편의 도입 없이도 non-homologous end joining (NHEJ)를 통해 편집이 진행될 수 있으며, 이경우에는 자체적인 효율 특성으로 삽입-결실(Indel) 돌연변이가 도입될 수 있다. 하지만, 원핵 생물의경우에는 NHEJ의 효율이 낮기 때문에 필연적으로 DNA 절편의 도입에 의존한 HR에 의존해왔으며,이 경우 동시 유전자 편집 시 낮은 세포 생존율이나 특정 미생물에서는 활용이 불가능하다는 점등의 한계가 존재해왔다.
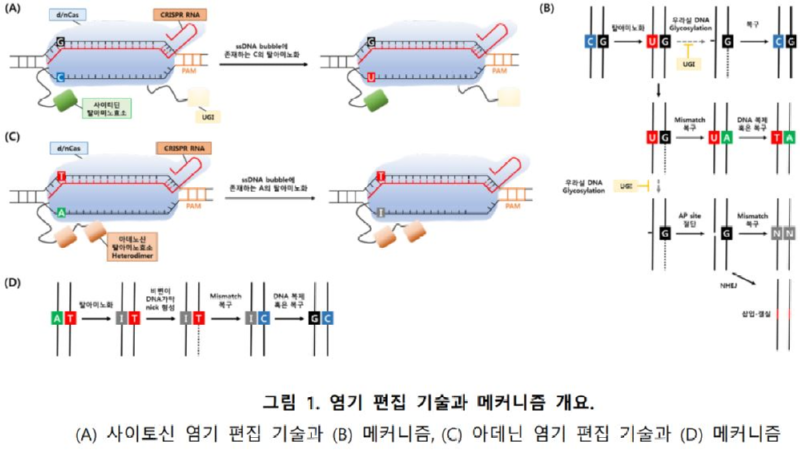
염기 편집(Base editing, BE) 기술은 위에서 서술한 문제를 해결 가능하다는 점에서 개발이되었고 다양하게 활용되어 왔다. 해당 기술은 CRISPR/Cas 시스템의 목적 특이성(Targeted specificity)과 핵산 탈아미노효소(Nucleobase deaminase)의 특성을 활용한 기술로 DSBs나 DNA절편의 도입 없이도 원하는 서열의 돌연변이를 일으킬 수 있다 (그림 1). 크게 두 가지 타입의 DNA 염기 편집 기술이 지금까지 개발되어 왔는데, 첫 번째는 사이토신 염기 편집 기술(Cytosine baseeditors, CBEs)로 C:G 염기 조합을 T:A로 변이할 수 있다 (그림 1A & 1B). 또 다른 한 가지는 아데닌염기 편집 기술(Adenine base editors, ABEs)로 A:T 염기 조합을 G:C로 변이시킨다 (그림 1C & 1D).해당 기술은 적용이 간단하다는 점, 높은 효율성, 동시 편집이 가능하다는 장점으로 유전자비활성화, 효소 진화(Protein evolution), 조합적 돌연변이 도입(Combinatorial mutation) 등 다양한분야에 활용될 수 있었다 (표 1). 본 리뷰에서는 최근 염기 편집 기술의 동향과 산업, 의학 분야와밀접한 연관을 갖는 미생물 내의 활용에 대해 주로 다루고자 한다.
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
지식
- BioINpro [바이오 융합 소재·제조] 미생물-전극 간 전자 흐름의 재설계: 합성생물학과 바이오인터페이스 기술의 융합 2025-03-31
- BioINwatch 신기술(합성생물학, 유전자편집기술 등) 발전에 따른 유전자변형미생물(GMM)의 합리적 규제 방안 2025-03-27
- BioINwatch 피부 유익균을 활용한 바르는 미생물 백신 2025-02-04
- BioINpro [2024 바이오 미래유망기술(하)] 바코드 미생물 2024-06-26
- BioINwatch 장내 미생물은 보호하면서도 병원성 세균만 선택적으로 공격하는 항생물질 발굴 2024-06-13
동향
발간물
- 이슈 보고서 2019 바이오미래유망기술의 이야기 - 제9화 “플라스틱 분해 인공미생물” 편 2019-12-06
- 이슈 보고서 2019 바이오미래유망기술의 이야기 - 제7화 “미토콘드리아 유전체편집을 통한 대사조절기술” 편 2019-09-05
- 이슈 보고서 2017년 BioINdustry 산업동향 보고서 2018-01-22
- 연구 보고서 바이오그린21사업 핵심기술별 특허분석-전반부(작물, 동물, 곤충, 농용미생물, 조류(algae)를 중심으로) 2007-04-01
- 연구 보고서 Protein factory program-미생물(박테리아, 효모, 곰팡이) 발현 시스템 및 동물세포 발현시스템 2005-04-30


 pdf_0003634.pdf
pdf_0003634.pdf