기술동향
전장유전체 염기서열분석 유전형검사법(wgSNP)을 통한 결핵 집단발생의 감염경로 분석
- 등록일2021-02-02
- 조회수6087
- 분류기술동향
-
자료발간일
2021-01-07
-
출처
질병관리청
- 원문링크
-
키워드
#결핵#유전형 분석#전장유전체염기서열분석
- 첨부파일
전장유전체 염기서열분석 유전형검사법(wgSNP)을 통한 결핵 집단발생의 감염경로 분석
초록
결핵의 집단발생 시 감염원 추적과 감염경로를 확인하기 위해 결핵균 유전형검사가 이용되고 있다. 기존 결핵균 유전형검사에는 Spoligotyping법과 24 유전자좌 MIRU-VNTR법을 사용하였으나, 변별력이 다소 떨어져 감염경로 추적에 어려움이 있었다. 질병관리청에서는 보다 변별력이 향상된 분석법으로 알려진 전장유전체 염기서열분석 유전형검사법(wgSNP)으로 대체하기 위해 2019년부터 시범 사업을 진행하고 있다.
본 글에서는 감염경로 확인이 불가능했던 집단발생 사례에 wgSNP법을 적용하여 감염경로를 재분석한 결과를 보고하고자 한다. 2015년부터 2018년 동안 국내 한 지역 내 4개 대학교에서 시행한 집단시설 역학조사를 통해 결핵균 11주를 수집하였다. 기존 유전형검사법으로 분석한 결과, 모든 균주의 유전형이 TBG0014로 동일한 것을 확인하였고, 역학적 연관관계 분석으로 집단감염으로 확인되었으나, 대학 간의 연관성은 확인되지 않았었다.
대학 간 연관성 분석을 위해 wgSNP법을 적용하여 분석한 결과, 각 대학교 내 결핵균주 간 유전자변이(SNP)는 0~1개로 확인되어 동일 감염원으로 발생되었음을 재확인하였으나, 대학 집단 간 유전자변이(SNP)는 196~264개로 3개 대학교에서 발생한 집단발생 사례는 서로 연관관계가 없는 것으로 확인하였다.
이 결과를 토대로, 집단시설 결핵역학조사의 분자역학검사법으로 wgSNP법을 적용하고자 하며 분석된 감염원 추적 등의 정보는 결핵 집단발생의 관리를 위해 제공하고자 한다.
들어가는 말
결핵은 Mycobacterium tuberculosis complex(MTBC)에 감염되어 유발되는 감염병으로, 전 세계적으로 연간 약 천만명의 신환자가 발생하는 것으로 보고되고 있다. 국내의 경우 해마다 20,000여명 이상의 신환자가 보고되고 있어 결핵관리를 위한 적극적인 대응이 요구되고 있다.
결핵은 매개체 없이 공기감염(비말핵)을 통해 전파되며, 감염인의 약 5~10%가 활동성 결핵으로 이환된다. 활동성 결핵환자 중 약 50%는 감염 2년 내에 발병하며, 나머지 50%는 평생에 걸쳐 발병하기 때문에 통상적인 역학조사만으로는 감염원 추적과 감염경로 확인에 한계가 있다[1].
이러한 문제점을 극복하기 위해 미국, 영국 등 세계 여러 나라에서는 감염된 결핵균 간의 연관관계 확인을 위해 결핵균
유전형검사(TB molecular typing)를 활용하고 있다. 이 결과를 이용하여 결핵환자 간 감염경로 뿐 아니라 특정지역 또는 인구집단 내 유행주 확인, 동일 전파경로에 포함된 추가 환자 또는 역학적 연관성을 확인하지 못한 환자군에서 발생한 집단발생의 조기 인지, 결핵실험실 내 오염, 재발 및 재감염 환자 감별 등으로 확대 적용하고 있다.
우리나라에서는 2015년부터 결핵균 유전형검사를 적용하여 집단시설 결핵역학조사용 결핵균에 대한 연관성 분석을 위한 ‘결핵균 감염경로 추적감시망’을 운영하고 있다.
결핵균 유전형검사법은 1990년대 중반부터 IS6110-RFLP가 사용되다가 변별력, 재현성, 검사자의 생물안전, 분석기술의 발전 등으로 2000년대 중반부터 12, 15, 24 유전자좌 MIRUVNTR(Mycobacterial Interspersed Repetitive Unit-Variable NumberTandem Repeat)법과 Spoligotyping법이 활용되고 있다.
최근에는 전장유전체염기서열(whole genome sequencing, WGS) 분석법이 대중화됨에 따라 결핵균 유전형분석법에도 WGS를 활용하는 사례가 증가하고 있다[2].
WGS법을 활용한 유전형검사법으로는 전장유전체염기서열 내 단일염기다형성 분석법(Whole Genome Single Nucleotide Polymorhism analysis, wgSNP)이 있으며, 이 방법은 결핵균 전체 유전자(약 4.4Mb)의 90% 이상을 분석하기 때문에 기존 검사법(전체 유전자의 약 1%를 대상으로 분석)에 비해 변별력이 월등히 높은 장점을 가지고 있다[3].
본 글에서는 기존의 Spoligotyping과 24 유전자좌 MIRUVNTR 검사법으로는 감염경로 확인이 불가능한 사례를 대상으로wgSNP법을 적용하여 감염경로를 재분석한 결과를 소개하고자 한다.
몸말
결핵균 감염경로 추적감시망을 통해 2015년부터 2018년까지 3,167개 집단시설(학교, 직장, 의료기관 등) 로부터 수집한 결핵균
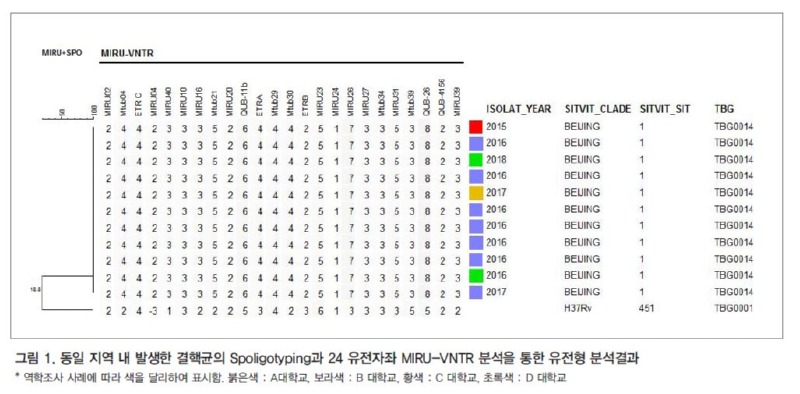
3,461주를 대상으로 Spoligotyping법과 24 유전자좌 MIRUVNTR법으로 분석한 결과, 일개 지역 내 4개 대학교에서 선별된 11주의 결핵균의 유전형이 TBG0014(Spoligotyping: Beijing clade, 24 유전자좌 MIRU-VNTR: 244233352644425173353823)로 동일한 것을 확인하였다(그림 1).
A 대학교에서는 지표환자(2015년)로부터 TBG0014형 1주가 수집되었고 추가환자는 없었다. B 대학교에서는 2016~2017년 사이에 7명의 환자가 발생하였고 동일 유전형임을 확인하였다. C 대학교에서 2017년 동일 유전형 1주가 분리되었고 역학조사 결과 B 대학교의 집단발생과 밀접접촉자로 확인되었다. D 대학교에서 2016년과 2018년에 동일 유전형을 가진 결핵균이 각각 1주씩 분리되었고, 역학조사 결과 학내 전파로 확인되었다. B 대학교와 C 대학교 간의 역학적 연관관계는 확인되었으나, 다른 학교 간의 연관관계는 확인되지 않은 상태였다.
wgSNP 결과, 11개의 TBG0014는 A 대학교, B와 C 대학교, D 대학교의 3개 집단으로 구분되었다. 각 집단 내 결핵균 간 유전자
변이 개수(SNP)는 0~1개로 확인되었고, A 대학교와 B 대학교 간 SNP는 264개였으며, A 대학교와 D 대학교 간의 SNP는 196개였다.
...................(계속)
☞ 자세한 내용은 내용바로가기 또는 첨부파일을 이용하시기 바랍니다.
관련정보
지식


 전장유전체+염기서열분석+유전형검사법(wgSNP)을+통한+결핵+집단...
전장유전체+염기서열분석+유전형검사법(wgSNP)을+통한+결핵+집단...