BioINpro
(BioIN + Professional) : 전문가의 시각에서 집필한 보고서 제공단일세포 유전체 연구분야의 성장동력
- 등록일2019-10-08
- 조회수8486
- 분류플랫폼바이오 > 바이오기반기술
-
저자/소속
이혜옥 수석연구원/삼성서울병원 유전체연구소
-
발간일
2019-09-30
-
키워드
#단일세포유전체#오믹스#세포생물학#안건세포지도 프로젝트
- 첨부파일
-
차트+
?
차트+ 도움말

삼성서울병원 유전체연구소
이혜옥 수석연구원
1. 개요
단일세포 유전체분석은 오믹스 형태로 소개된 이후 [1], 지속적으로 플랫폼과 실험 및 분석법이 발전하여, 이제는 세포생물학 전 분야에 적용될 수 있을 정도의 매우 보편적인 기술로 성장하였다. 이러한 발전의 열매인 동시에 앞으로의 성장 동력이기도 한 인간세포지도 (Human Cell Atlas) 프로젝트를 중심으로 단일세포유전체 분석 기술의 발전 과정과 앞으로의 전망에 대해 간단히 정리해 보고자 한다.
2. 본론
가. Human Cell Atlas
2016년 10월 13, 14일 양일에 걸쳐 영국 런던에서 정상과 질병 상태의 Human Cell Atlas (이하 HCA)를 제작하기 위한 모임이 열렸다. HCA 콘소시움은 미국 Broad Institute의 Aviv Regev와 영국 Wellcome Trust Sanger Institute의 Sarah Teichmann이 공동 의장을 맡고 있으며 10개국 27명의 연구자가 의사 결정을 하는 구조이다. 세포는 생명체를 구성하는 기본 단위로 개별 세포 및 그들이 조직과 기관, 나아가 생명체를 구성하기 위해 연결된 방식을 이해하는 것은 생명과학 분야의 궁극적인 목표이다. 세포 타입에 대한 정의는 형태학적 (morphological) 분류로부터 소수의 단백질 마커 발현에 기반한 표현형 (phenotypical)을 사용하여 이루어져 왔으며 인간에게 대략 200 여종이 있다고 알려져 있다. 이러한 분류법을 유전체데이터로 보완 혹은 대체하면 더욱 다양한 세포 타입을 정확히 분류해낼 수 있을 것으로 판단한 것이다. 2017년 10월에 발간된 HCA white paper ([q-bio.TO]
arXiv:1810.05192)에는 HCA 프로젝트를 수행하기 위한 로드맵이 제시되어 있으며 “massively parallel single cell RNA sequencing” (대용량 단일세포 전사체 분석)이 그 중심에 있음을 확인할 수 있다 (그림 1).
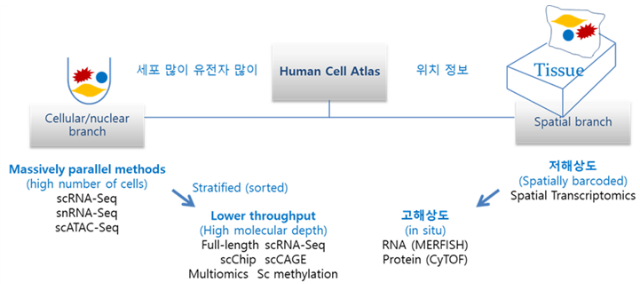
그림 1. Human Cell Atlas의 전략: Massively parallel vs. Spatial branch (HCA white paper 참조)
HCA는 open 콘소시움 형태로 참여자에 대한 제한이나 특별한 자격요건이 없어 홈페이지를 통해 (humancellatlas.org) 간단히 가입할 수 있다. 1년에 2회 general 미팅을 개최하며 등록 및 직접 참석은 국가별로 정해진 대표 (delegate)를 통해 신청이 가능하다. 2019년 현재 삼성유전체연구소의 박웅양박사와 한국생명공학연구원의 김선영박사가 한국대표로 활동하고 있다. 등록비는 무료이나 많은 인원을 수용할 수 없는 관계로 HCA 미팅은 스트리밍 서비스를 통해 생중계되어 누구나 on-line을 통한 미팅 참여가 가능하다. Scientific 결과보다는 주로 working group의 활동을 보고하거나 breakout 세션으로 나누어 현안에 대한 그룹 논의를 이어가는데, 2019년도 상반기 7차 미팅의 경우 1) Working group 활동 보고: Standards and Technology, Common coordinate framework, Data coordination platform, Ethics 2) Cross collaboration: HCA in Asia, China, Australia, Swedish development consortium, French developmental consortium, Wellcome trust consortium. MRC consortium, NIH HubMAP consortium 3) Tissue 혹은 Organ별 Atlas: Brain, Gut, Immune cell, Liver, Tumor, Lung 4) Round table discussion: Ethics, Funding, Cross-collaboration 5) Breakout 세션: Experimental design and method, Data standardization, normalization, and integration, Common coordinate framework로 진행되었고 토론 내용 정리로 마무리되었다. 2019년 하반기 미팅이 10월 11-12일 스페인 바르셀로나에서 개최되니 on-line 참여를 통해 HCA에 대해 알아보기 바란다. 아울러 HCA asia 미팅도 한국, 싱가폴, 일본을 중심으로 개최되고 있으니 관련 내용을 찾아보면 HCA에 대한 정보를 얻을 수 있을 것이다. 2019년도 HCA asia 미팅은 싱가폴에서 개최 예정이다. (https://www.gisevents.org/hcam2019/)
나. Massively parallel single cell RNA sequencing
대용량 단일세포 전사체 분석 플랫폼의 선두 주자는 단연코 10X Genomics사의 Chromium 플랫폼으로, 미세오일방울 안에 한 개의 세포와 바코드시퀀스가 붙은 gel bead를 가두어 세포바코드와 UMI (unique molecular identifier)를 mRNA로부터 역전사되는 cDNA에 부착하게 된다. 바코드가 태깅된 cDNA를 증폭하여 시퀀싱 라이브러리를 제작, transcriptome과 세포바코드 정보를 함께 얻는 과정을 거치며 transcriptome의 정량화는 UMI count를 통해 얻는다. UMI는 100,000개 정도의 고유한 시퀀스를 사용함으로써 증폭 바이어스를 최소화하게 된다 (그림 2).
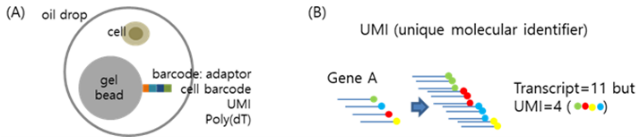
그림 2. Massively parallel RNA sequencing의 주로 사용되는 droplet 방식의
single cell capture (A)와 UMI(B)에 대한 간단한 모식도
Massively parallel single cell RNA sequencing의 기본은 3’sequencing으로 polyA tail에 근접한 100bp정도만의 정보를 사용하여 유전자의 정체를 파악한다. 따라서 3’RNA sequencing data를 사용하면 3’을 벗어나는 시퀀스를 활용하는 여러 정보들, 예를 들면 isoform, T cell receptor (TCR), B cell receptor (BCR), SNP, SNV등은 얻을 수 없거나 혹은 매우 제한적으로 얻게 된다. 이를 극복하기 위해 최근 5’RNA sequencing 을 사용하는 경우가 늘고 있다. 이 경우, polyA tail에 붙는 oligo-dT primer를 soluble form으로 사용하고, gel bead에는 대신 5’switching oligo를 부착하여 세포바코드와 UMI를 5’reads에 붙여주게 된다 (그림 3).
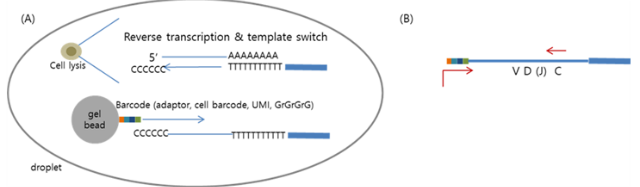
그림 3 (A) 5’ single cell RNA-Seq (B) 5’-tagged cDNA를 template로 사용한 TCR혹은 BCR target enrichment
5’RNA sequencing의 중요한 장점 중 하나는 TCR이나 BCR을 증폭시켜 sequence 정보를 얻을 수 있는 것이고 최근 adaptive immune system연구가 활발해지며 3’RNA sequencing을 대체하고 있다. 물론 full-length transcriptome을 활용하면 여러가지 transcript sequence 에 대한 정보를 최대로 얻을 수 있으니 (데이터 용량과 시퀀싱 코스트 상승) 목적에 따라 적절하게 선택하면 되겠다. Full-length RNA sequencing을 얻기 위해서는 대부분 plate 기반 기술과 SMART-seq 시약을 사용한다.
다. 세포의 공간적 정보를 보존하는 시퀀싱 방법들
HCA white paper의 로드맵을 보면 (그림 1), HCA 프로젝트의 1차 목표 데이터는 많은 숫자의 세포 혹은 핵을 분리하여 생산하는 massively parallel RNA sequencing [2]이었다. 이는 기술의 접근성과 완성도에 기인한 것으로 보이며, 사실 정확한 세포지도 완성을 위해서는 조직내 위치 정보가 반드시 필요하다. 즉, 조직의 이미지 정보와 세포의 전사체 정보를 통합할 수 있는 spatial branch 플랫폼 개발이 필요한데 대략 2가지 접근법이 있다. 첫번째 바코딩 방법 [3]은 위치바코드가 심어진 슬라이드에 조직을 붙이고 이미지 촬영 후, in situ cDNA 합성을 함으로써 cDNA가 생산된 위치를 표지하는 것으로 최근 10X Genomics사에서 VISIUM이라는 제품으로 출시를 예고하고 있다. 장점은 고가의 장비가 필요하지 않다는 점이고 단점은 위치바코드가 심겨진 간격이 단일세포 해상도가 아니라는 점이다. 두번째 방법은, in situ transcriptome data를 in situ sequencing [4]이나 in situ probe hybridization (예: MERFISH) [5] 을 통해 얻는 것이다. In situ sequencing은 고가의 현미경과 시퀀서를 결합하여야 하므로 개별 실험실 구축이 매우 어렵다. MERFISH는 FISH 프로브를 비교적 짧은 oligo에 부착하여 binary code로 transcript를 동정하는 방법으로 in situ sequencing 보다는 상대적으로 접근성이 높다. 또한 단일세포 혹은 세포 내 위치 정보까지 확인이 가능한 고해상도 기술이므로 첫번째 바코드 기술을 보완할 수 있을 것이다.
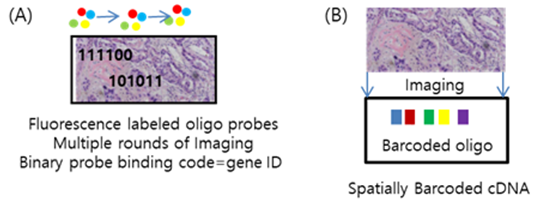
그림 4. Spatial branch 데이터 생산 플랫폼 (A) in situ (MERFISH) (B) barcoded method
올 하반기부터 향후 2~3년 간은 Spatial data와 관련된 실험 및 분석 기술의 발전이 단일세포 유전체 분석 분야의 최대 화두가 될 것으로 예측된다. VISIUM과 같은 위치바코드 플랫폼의 비약적 도약이 기대되며 MERFISH가 머지 않은 미래에 접근이 용이한 상용플랫폼으로 출시되길 기대해 본다.
지식
동향
- 기술동향 BRAIN Initiative Cell Census Network (BICCN) 및 BRAIN Initiative Cell Atlas Network (BICAN) 최신 연구동향 2024-12-20
- 기술동향 다중오믹스와 머신러닝 기반의 종양항원예측 알고리즘과 암치료 2021-08-11
- 기술동향 오믹스를 이용한 통합 분석의 임상적 활용 2019-12-24
- 기술동향 질병 연구를 위한 오믹스 통합 분석 2019-07-24
- 기술동향 Predictive genomics를 통한 종양 임상 표현형을 예측하는 방법론 new 2018-09-18


 BioINPro_2019.09_67호_Human Cell Atlas_이혜옥.pdf
BioINPro_2019.09_67호_Human Cell Atlas_이혜옥.pdf
 Human Cell Atlas의 전략 Massively parallel vs. Spatial branch
Human Cell Atlas의 전략 Massively parallel vs. Spatial branch