BioINpro
(BioIN + Professional) : 전문가의 시각에서 집필한 보고서 제공단일 뉴런 분석기술 (Single neuron analysis)
- 등록일2018-04-26
- 조회수21090
- 분류플랫폼바이오 > 바이오기반기술
-
저자/소속
구자욱 책임연구원 /한국뇌연구원
-
발간일
2018-04-26
-
키워드
#단일 뉴런 분석기술 #미래유망기술
- 첨부파일
-
차트+
?
차트+ 도움말

1. 기술의 정의
단일뉴런 분석기술(Single neuron analysis)은 “1개”의 신경세포를 분리하여, 이들의 유전자 발현 및 단백체 결합, 이들 개별 뉴런간의 연결성, 그리고 이들의 신경활성 변화를 종합적으로 분석하여 각 개별 신경세포간의 기능적 네트워킹을 파악하는 기술이다.
종래의 연구에서는 뇌에서의 기능적 네트워킹을 파악하기 위해, ‘뇌영역’간의 연결성을 확인하고, 특정 뇌영역에서의 유전체나 단백체적 특성을 분석하며, 관심 뇌영역의 신경활성 변화를 측정하였다. 하나의 뇌영역안에서 신경세포의 ‘유형’에 따라 유전체나 단백체적 특성을 분석하고 이와 연관지어 그 신경활성의 변화를 세포유형별로 종합적으로 분석한 것 또한 오래전 일이 아니다. 대부분의 뇌영역에서 다수를 차지하는 글루탐산성 뉴런이나 가바성뉴런들 사이에 극히 적은 비율 (1~5%)로 존재하지만 중요한 스위치 역할을 하는 특정 중간뉴런(interneurons)의 특성을 세포군 수준에서 분석해내는 연구방법은 최근에도 주요 저널에 등장하고 있다.
하지만 이렇게 특정 ‘뇌영역’안에 특정 ‘유형’의 뉴런을 분리-분석할 수 있는 첨단기술을 이용한다 하여도, 이질적인 연결성 및 내적 특성을 갖는 세포의 집단을 통째로 분석하면, 개별 뉴런의 특징은 사라지고 역시 평균값만을 얻게 된다.
우리의 뇌는 외부 환경 자극에 민감하게 그리고 적절히 반응하기 위해 진화되어, 신체의 어느 기관보다도 큰 가소성을 갖는다. 따라서, 특정 뇌영역의 같은 종류의 세포군이라고 하더라도, 심지어는 비슷한 모양과 유사한 유전적 구성을 갖더라도, 다른 세포 및 다른 뇌영역과의 연결 특성이나 뉴런 활성패턴에 따라 각 개별 뉴런의 특성 및 기능은 시시때때로 크게 달라질 수 있다. 결국 단일 뉴런 하나하나의 연결 패턴을 분석하고 각 개별세포의 유전체 및 단백체적 특성, 그리고 그 신경활성을 종합하여 분석해야 할 필요성이 대두된다.
▶ 단일뉴런 분석기술의 요소기술
단일뉴런 분석기술은 크게 오믹스 프로파일링 기술, 단일뉴런 표지 및 활성 측정기술, 단일뉴런 신경연결망 분석기술, 그리고 단일뉴런 정보 통합 네트워크 분석기술로 나누어 생각할 수 있다.
(1) 단일뉴런 오믹스 프로파일링 기술
단일뉴런 오믹스(Omics) 프로파일링 기술은 크게 유전체(Genome), 전사체 (Tranome), 후성유전체(Epigenome), 그리고 단백체 (Proteome) 프로파일링 기술로 나눌 수 있다.
정상적인 발달단계를 거친 뇌의 경우 유전체내에 유전적 돌연변이를 찾아내기는 쉽지는 않지만, 체세포 변이(somatic variation) 등의 유전적 이질성을 확인하거나 뇌질환 병리에서의 변이 역할을 이해하는데 단일뉴런 유전체 분석이 충분히 유효하다 (1,2). 실제로 몇몇 뇌질환은 유전체 및 염색체 불안정성 (예를 들면, number variations 등)과 깊이 관련되어 있다. 각 뉴런으로부터 제작되는 라이브러리에 고유 서열을 갖는 바코드를 붙여 수십-수만 개의 샘플을 함께 시퀀싱하여, 시퀀싱 후 각 세포별로 데이터를 분석하는 것이다.
유전체(genome)에 비해 전사체는 훨씬 역동적인 변화 양상을 보인다. 외형상 동질적으로 보이는 하나의 신경세포군에서조차도 그 유전자 발현은 세포마다 다르다고 익히 잘 알려져 있다. 또한, 뉴런의 전사체는 신호전달 반응과 같은 내적요소 뿐만 아니라, 스트레스 반응과 같은 외적요소를 반영하여 역동적으로 변하며, 세포의 기능 및 유형을 반영하며, 세포의 발달단계를 반영하기도 한다(그림 1).
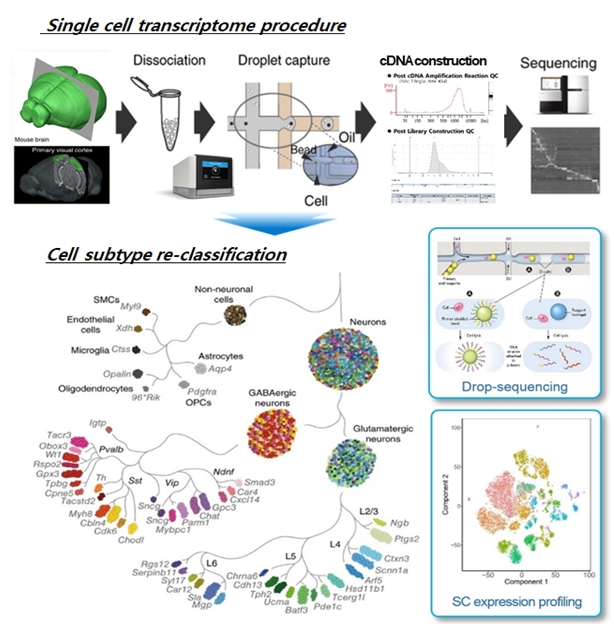
[그림 1] 단일세포수준의 전사체 프로파일링을 통한 뉴런 아형의 재분류 (4)
후성유전학적 기전(Epigenetic mechanism)은 DNA의 염기서열이 변하지 않는 상태에서 유전자 발현이 조절되는 것으로, 히스톤 단백질의 변형, chromatin의 리모델링, 전사인자(tranion factor)의 결합 및 DNA 메틸화(methylation) 등의 현상을 포함한다 (3). 이러한 기전을 통해, 세포들은 내적/외적 요소를 고유의 유전정보에 반영하여 고유한 유전자의 발현에 변화를 준다.
단일뉴런 수준에서 후성유전체를 분석하는 것이 중요한 이유는, 우리의 뇌가 외부환경으로부터의 다량의 정보를 처리하여 반응하는 가장 큰 가소성을 보이는 생물학적 기관이라는 사실과 더불어, 단일세포의 후성유전체를 확인함으로써만이 답할 수 있는 질문이 존재하기 때문이다. 예를 들어, 개별 뉴런마다 연결 특성이나 활성패턴이 따르기 때문에 야기되는 전사체상 차이는 후성유전학적 기전을 통해서 야기된다. 또한, 세포의 기능이나 발달단계에서 변화 및 전이를 보일 때 후성유전학적 기전을 통해서 그 유전자의 발현 즉, 전사가 조절된다 (5).
하지만, 뉴런의 전사체, 후성유전체 분석만으로는 단백질로 번역된 후 일어나는 post-translational modifications (예를 들면, phosphorylatio)나 세포간 또는 세포 안에서의 신호전달 등에 관련된 현상의 특성 및 기능을 예상할 수는 없다. 이러한 단일뉴런의 단백체 분석은 극미량의 단백질을 이용해야 할 뿐만 아니라 단백질은 DAN/RNA처럼 증폭할 수 없기에 어려움이 있어 왔다.
근래에는 mass cytometry의 최신 버전이라고 볼수 있는 matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry 등의 기법을 통해, 단일세포 수준에서 high throughput 스타일의 단백체 분석이 실현 가능해졌다 (6,7). 최근의 단일세포 단백체 분석기법은 3차원 이미징기법과 결합하여 하나의 세포에서의 단백질량, 단백질의 위치, 그리고 근접 세포들의 분자 상태에 관한 정보까지 제공할 수 있으며(그림 2), 이를 통해 단일세포 단백체 분석은 세포간 소통에 관한 연구 뿐만 아니라, 세포의 발달과 기능에 관한 미소환경에 대한 정보 역시 제공할 것으로 기대된다 (8).
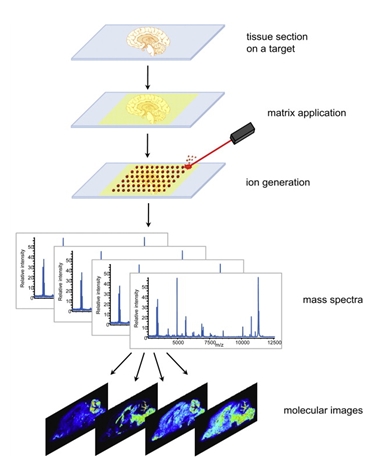
[그림 2] 단일 뉴런 수준에서의 단백체 분석 개량고 (6)
(2) 단일뉴런 표지 및 활성 측정기술
뉴런 안팎의 자극에 의해 활성화되는 뉴런들의 개별 생체신호를 측정하고 표지하는 방법 또한 최근 발전을 거듭하고 있다.
① 단일뉴런의 활성 측정기술 :

[그림 3] in vivo multiunit recording system
자유행동을 하는 동물의 뇌에서 단일뉴런의 활성화된 뇌생체신호는 in vivo recording 방법을 사용하여 측정할 수 있다. 근래에 들어서는 뇌의 생체신호를 다채널로 측정하는 probe로 개량하여 몇몇 개의 뉴런의 활성을 동시에 측정하고 그 측정치를 분석하여 그 위치까지 알아낼 수 있다(그림 3). 하지만 이러한 방법은 세포유형에 따른 활성측정에 한계가 있다. 측정 후 활성 패턴을 면밀하게 분석함으로써 그 활성세포가 어떠한 유형인지 어느 정도 구별이 가능하지만, 관심 뉴런을 표지하고 그 세포만의 활성을 측정해 낼 수는 없다.
역동적으로 변화하는 뇌의 생체신호는 뇌 이미징 기술을 통해서도 측정할 수 있다. 특수한 파장과 빈도수의 광원(예, femtosecond laser)과 생체내 칼슘을 인지하였을때 형광을 띄게 개발된 칼슘 센서(예, GCaMP6)를 이용하는 다광자 현미경은 살아 있는 동물의 뇌 표면으로부터 대략 1mm 깊이에 있는 미세한 개별 뉴런 및 시냅스까지 고해상도(200~400nm)로 관찰할 수 있게 한다.

[그림 4] 가상현실 속에서 인지실험 수행중인 생쥐 뇌에서 다광자 현미경을 통한 칼슘 이미징과 개별 뉴런의 활성 측정
최근에는 바이러스와 유전자 변형 생쥐를 이용한 세포유형 특이적 칼슘이미징 기법을 이용하여, 살아 있는 동물의 뇌 안에서 일어나는 여러 종류의 뉴런들의 구조적·기능적 변화를 개별 뉴런별로 짧게는 밀리초(millisecond) 단위로 관찰할 수 있다. 현재는 가상현실 실험기법과 결합하여 특정 인지 행동을 수행하면서도 다광자 현미경을 통해 각 개별 뉴런의 활성을 측정 분석할 수 있다 (그림 4) (9,10)
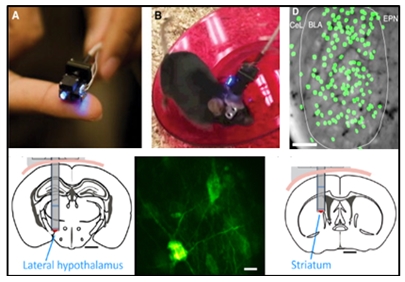
[그림 5] 자유행동 중인 생쥐의 뇌에서 Endomicros를 이용한 뇌심부 생체신호 측정
하지만 다광자 현미경을 이용한 칼슘 이미징 기법은 투과에 한계가 있기 때문에 1mm 정도 깊이의 대뇌피질에서만 관찰 및 실험이 가능하다. 이러한 한계를 극복하기 위하여 최근 이용되는 실험기법이 endomicros이다. 현미경의 렌즈에 해당하는 부분을 얇고 길게 만들어 머리 위 해당부분에 이식한다 (그림 5). 이를 통해 자유행동을 하는 동물의 hypothalamus나 striatum 등 뇌심부에 존재하는 뉴런의 활성을 측정할 수 있으며 (11), 상용화된 제품들도 이용되고 있다.
② 활성화된 단일뉴런 표지 측정기술 :
활성화된 개별 뉴런을 표지하기 위해 직접 probe 등을 타겟 뇌영역에 위치시킬 필요없이, 활성을 반영하는 분자 자체를 표지하는 방법이 존재한다. 지난 10년 동안 외부 자극에 의해 활성되는 뇌의 영역들을 동정하는 데에 Arc나 c-Fos등 immediate early gene이 많이 응용되어 왔다. 근래에는 뉴런의 활성의존적인 전사에 관여하는 이들 유전자의 promoter나 enhancer의 특징을 이용하여 활성 뉴런의 live 이미징까지 가능해졌다 (12).
예를 들어, 2007년 미국 Scripps 연구소의 Mayford 교수 연구실은 c-fos promoter에 의해 유도 발현되는 tetracycline transactivator (tTA)을 이용하여 “TetTag”기법을 만들어냈다(Reijmers et al., 2007). 이 기법은 c-fos promoter의 downstream에 tTA를 발현하는 유전자변형생쥐라인과 tTA에 의해 활성화되는 tetracycline responsive element (TRE)의 downstream에 지시자인 LacZ 을 발현하는 유전자변형생쥐라인을 교배하여 가능하다.

[그림 6] c-fos/tTA 시스템을 이용한 단일뉴런 수준에서의 engram 표지 및 광활성화 방법 (13)
이러한 이중 유전자조작 실험기법은 doxycycline을 처리하지 않는 동안만 활성되는 신경세포들의 특정 마커들이 1-2주 동안 발현하게 함으로써 여러 분야에 이용할 수 있을 길을 열었다. 가령, 이 방법을 통해 연구자들은 공포기억을 훈련하는 동안에만 활성화되는 해마(hippocampus)의 특정 군집을 구별해내고 이들 뉴런을 공포 기억 재생시 광유전학적 방법을 사용하여 다시 활성화시킴으로 engram (기억소, 혹은 기억흔)의 존재를 확인하였다 (13,14) (그림 6).
[그림[7] 특정조건에서(예, 질환, 환경요인, 특정행동) 활성화된 개별 뉴런들의 전사체 프로파일링 개념도
세포활성 의존적인 promoter와 시간-제한적인 약물을 활용하여 특정 행동조건하에서만 활성화되는 뉴런을 표지하는 이 기술은, 특정 행동을 하거나 특정 조건에 처한 생쥐의 뇌에서 활성화된 뉴런을 분리해내어 활성의존적인 오믹스 변화를 단일세포 수준에서 분석한다거나 (그림 7), 그 단일뉴런의 활성을 측정 및 조절하는 기법으로 발전할 것으로 기대되고 있다 (12).
(3) 단일뉴런 신경연결망 분석기술
뇌영역간의 연결성을 보기위해 trans-synaptic retrograde tracing 기법이 그간 큰 성공을 거둬왔다. 뇌 영역간 연결성을 기반으로 한 전사체 프로파일링 또한 성공적으로 이뤄지고 있다 (15,16). 하지만 이러한 방법으로는 특정 뇌영역에서의 ‘개별 뉴런’이 어떤 다른 뇌영역으로부터 input을 받는지, 그리고 어떤 유형의 뉴런들과 연결되어 있는지 알 수 없다.
한편, 뇌영역간 전사체 프로파일링을 통한 분자 정보만을 가지고도 뇌영역간 분자 기능의 연결성을 가늠할 수 있는 분석기법이 존재한다. 예를 들어, Weighted Gene Co-expression Network Analysis(WGCNA) 기법은 유사 기능을 가진 유전자들은 유사한 발현 패턴을 보인다는 전제를 근거하여 여러 관심 뇌영역의 유전체 데이터를 분석하여, 뇌영역간 유전자들의 상호작용을 도출할 수 있는 것이다.
이 분석기법은 전뇌 수준에서 단일세포 수준까지 모두 적용이 가능하며 (17,18), 특정행동이나 질환에서 실제 기능을 하고 있는 회로 활성데이터를 결합 분석함으로써, 다수준 (전뇌-단일세포)에서 기능성 회로망에 기반한 유전자 발현 네트워크 규명까지 가능하게 한다. 특히, 단일세포 수준에서의 공발현 분석법은 매우 높은 수준의 정확도를 요구하기 때문에, 전자현미경과 공초점 현미경 등의 회로망 tracing기법(19)이나, 개별 뉴런의 공간적 배치를 알 수 있는 CLARITY(뇌투명화 기법, 20)등과의 결합 활용이 기대되기도 한다 (그림 8).
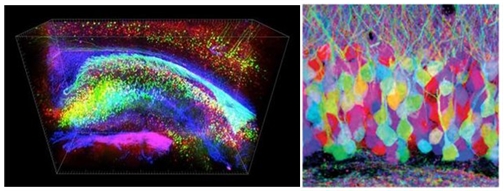
[그림 8] CLARITY(왼쪽)와 Brainbow 기술(오른쪽)
(4) 단일뉴런 정보 통합 네트워크 분석기술
단일 뉴런 수준에서, 앞서 설명했던 오믹스 기술, 뉴런의 활성 측정기술, 그리고 CLARITY-tracing 기술을 통해 얻는 데이터량은 상당할 것으로 누구든 예상할 수 있다. 이러한 데이터를 전통적 통계 방법으로 분석하기는 불가능할 것이다, 따라서 회로를 이루고 있는 단일뉴런들의 활성, 오믹스를 통합 분석하기 위해서는 그 분석 알고리즘을 개발하는 것 자체가 큰 시도가 된다(21). 또한, 데이터분석을 표준화하는 데에도 역시 적지 않은 어려움이 따른다. 극미량의 DNA, RNA의 증폭하는 과정으로 인해 발생되는 노이즈가 존재하며, 세포핵과 세포막을 분리한 후 단일세포 수준에서 단백체를 분석하는 과정에서는 특정 막단백질의 공간정보가 왜곡될 확률이 높다.
무엇보다도, 단일세포 수준의 오믹스 데이터를 분석하는 데에 이용하는 기존의 알고리즘을 차용하기 때문에 여러 가지 오차가 발생한다. 네크워크를 이루고 있는 단일뉴런들의 오믹스를 분석하기 위해서는 이러한 특징을 반영한 다양한 생물정보학적 시각과 노력이 요구된다 (22).
2. 국내외 동향
단일세포 오믹스 프로파일링 기술은 날로 발전하여 세계 여러 그룹에서 뇌 일부 부위를 구성하는 전체 뉴런의 유전자 발현을 단일세포 수준에서 분석하고 있다. 이와는 병렬적으로, 형질전환 생쥐 기술을 활용하여 각각의 뉴런들이 서로 다른 형광을 발현하게 하여 구분 가능하게 하고 그 연결성을 분석할 수 있는 Brainbow technique 역시 진화를 거듭하고 있다 (23). 하지만 개별 뉴런의 형광 발현이 무작위적이며 발현 형광의 수가 한정적이라는 단점을 (그림 8) 해결하기 위해 여러 방법이 개발되고 있다. 그중에서도, 뇌의 각 개별 뉴런을 Sindbis virus를 사용하여 고유의 ‘mRNA 바코드’로 전염시켜 단일세포 수준의 시쿼싱이 가능케 하는 MAPseq (Multiplexed Analysis of Projections by Sequencing) 기법이 소개되었다 (24). 조합에 따라 1018개 정도 종류의 바코드가 가능한 이 기법을 통해 가능한 색깔의 수만큼만 가능했던 Brainbow 기법 기반 뉴런 연결망 규명의 단점을 극복할 수 있다. 이 기법 역시 전뇌 수준으로 회로망을 분석하기에는 시간이 오래 걸린다는 단점이 존재하나, 기능성 오믹스 분석과 결합할 수 있다는 큰 장점이 있다.
...................(계속)
* 로그인 하셔야 자세한 정보를 모두 보실 수 있습니다.
-
이전글
- 인공효소 체인
-
다음글
- 합성배아 (Synthetic embryo)
지식
동향


 단일 뉴런 분석기술.pdf
단일 뉴런 분석기술.pdf
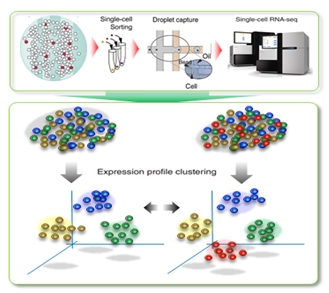
 MAPseq의 개요도
MAPseq의 개요도